锌螯合肽的两端排序法定量构效关系
黄晶晶1,余 敏2,马 敏3,鄢 嫣1,张福生1,殷俊峰1,谢宁宁1,*
(1.安徽省农业科学院农产品加工研究所,安徽 合肥 230031;2.安徽农业大学茶与食品科技学院,安徽 合肥 230036;3.安徽农业大学理学院,安徽 合肥 230036)
摘 要:为了研究锌螯合肽结构与生物活性之间的关系,构建定量构效关系(quantitative structure-activity relationship,QSAR)模型。采取两端排序法将56 条不同长度的合成肽规格化,采用18 种氨基酸描述符,利用偏最小二乘法进行分析。发现5 种氨基酸描述符对应的QSAR模型的相关系数达到建模要求,分别为描述符FASGAI、Z、HESH、C和ST,其中描述符FASGAI最优(R2=0.827 3、Q2=0.602 2、估计均方根误差=0.168 6、Q2ext=0.717 2、预测方根误差=0.255 8)。对描述符FASGAI所构建的模型进一步分析发现,多肽序列中的氨基酸位置对多肽锌螯合活性的影响力依次为C3>N3>C1>N1>N2>C2,同时,多肽各位置上氨基酸残基的立体属性会影响其螯合活性。该模型的成功建立为锌螯合肽定量构效关系的研究提供了探索性思路。
关键词:菜籽源;锌螯合肽;定量构效关系
锌是人体必需的微量元素之一。它是机体内近2 000 种转录因子的必需组成部分,与超过300 种代谢酶的活性有关,影响着机体的免疫系统和氧化还原平衡,可以作为慢性血管疾病、癌症、神经退行性病变、免疫紊乱和衰老等病理状态下的抗氧化因子和抗炎症因子[1]。肽金属螯合物作为一种微量元素补充剂,可以通过肽的吸收途径转运进入血液循环系统,吸收效率高、无竞争性,成为当前研究热点之一。金属螯合肽(metal chelating peptide,MCP)通常能够螯合Fe2+[2]、Fe3+[3]、Cu2+[4]、Zn2+[5]和Ca2+[6]等,但现有研究鲜见提及其结构信息与螯合活性之间的关系。Torres-Fuentes等[7]认为MCP和Cu2+的螯合能力与His含量有相关性(R2=0.68);de la Hoz等[8]发现His、Lys和Arg与酵母肽的Fe3+离子的螯合能力有关。目前只初步证明一些特征氨基酸在MCP结构信息中的作用,同时,有关锌螯合肽结构对螯合活性的影响研究仍显不足。
基于计算机辅助计算的定量构效关系(quantitative structure-activity relationship,QSAR)是将化合物的结构信息、理化参数与生物活性进行分析计算,建立合理的数学模型,已经被成功运用到苦味肽、抗菌肽、抗氧化肽[9]和降压肽[10]等多种食源性活性肽中,为锌螯合肽研究提供了切实可行的方法。QSAR基本思路包括:选择、设计、合成一系列多肽类似物,并测定活性;对多肽类似物序列进行定量表征;统计方法进行建模;分析模型并预测优异肽序列。近年来,QSAR的发展主要是对结构表征中氨基酸描述符和数学建模中统计方法的研究[11]。氨基酸描述符分为2D描述符和3D描述符,2D描述符主要表征电荷性、疏水性、立体性和氢键属性等参数。包括了TOF[12]、Z-scale[13]、VHSE[14]、DPPS[15]、T-scale[16]、HESH[17]、VMEE[18]、VSTV[19]、SZOTT[20]、FASGAI[21]、ST-scale[22]、VSTA[19]、SVG[23]、VSW[23]。Sneath等[24]首先进行了肽的QSAR研究,使用了20 种编码氨基酸的物化属性作为描述符,通过定量序效模型(quantitative sequence-activity model,QSAM),分析了脑下垂体-抗利尿激素类似物。3D描述符主要表征侧链表面积、侧链电荷指数、拓扑参数和几何指数等参数,包括ISA-ECI[25]、MS-WHIM[26]。建模的分析方法有多元线性回归、偏最小二乘法(partial least squares method,PLS)、支持向量机、主成分分析、逐逐步线性回归、人工神经网络和遗传算法-PLS等[27]。
本研究前期采用多种商业蛋白酶水解菜籽蛋白,测得水解物的Zn2+螯合活性为33.35%~86.93%。以菜籽蛋白来源的活性肽ASH为基础,利用多种金属螯合活性较强的氨基酸(H、C、V、M、Y、D、Q、E、W)与其他氨基酸(A、G、I、L、F、S、T)进行C端、N端替换,设计合成一系列多肽,形成待试数据库,采用18 种氨基酸描述符,利用PLS统计方法进行QSAR建模,揭示高螯合活性肽的结构特征。利用两端排序(two-terminal position numbering,TTPN)法将不同长度的多肽进行规格化,最大程度保留其结构信息,使所有的样品都能进行表征建模。最后,对菜籽源的虚拟水解肽进行活性预测,筛选出具有优异螯合活性的小肽。
1 材料与方法
1.1 材料与试剂
系列合成肽 百意欣生物公司;锌试剂、锌标准溶液、无水乙醇(分析纯) 国药集团化学试剂有限公司;谷胱甘肽 上海源叶生物科技有限公司。
1.2 仪器与设备
JA1103N型电子天平 上海民桥精密科学仪器有限公司;ZD-85气浴恒温振荡器 江苏金坛市精达仪器制造有限公司;HR801酶标分析仪 深圳市华科瑞科技有限公司。
1.3 方法
1.3.1 锌螯合肽数据库的建立
测定人工合成的51 条三肽、5 条四肽(序列基于前期研究所获的菜籽源锌螯合活性肽ASH,ASH来源于菜籽蛋白的12S球蛋白序列中的f204~206碎片)的锌螯合率[28],形成待试数据库。所有的数据按照2∶1随机分配成校正集、预测集。
锌螯合活性参照汪婵等[28]的方法测定,其活性是相对于阳性对照的相对值。
1.3.2 氨基酸描述符数据库的建立
查阅收集到的18 种氨基酸描述符分为两类,见表1。
表1 氨基酸描述符
Table1 Amino acid descriptors
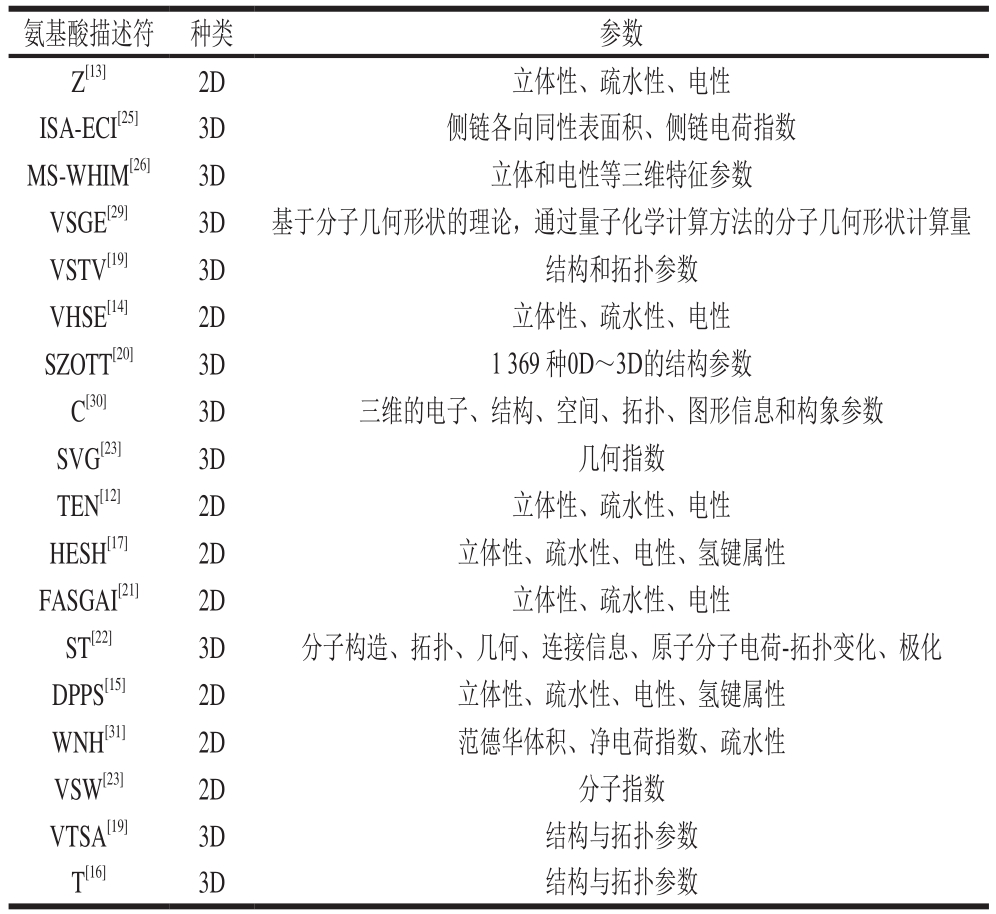
氨基酸描述符 种类 参数Z[13]2D 立体性、疏水性、电性ISA-ECI[25]3D 侧链各向同性表面积、侧链电荷指数MS-WHIM[26]3D 立体和电性等三维特征参数VSGE[29]3D 基于分子几何形状的理论,通过量子化学计算方法的分子几何形状计算量VSTV[19]3D 结构和拓扑参数VHSE[14]2D 立体性、疏水性、电性SZOTT[20]3D 1 369 种0D~3D的结构参数C[30]3D 三维的电子、结构、空间、拓扑、图形信息和构象参数SVG[23]3D 几何指数TEN[12]2D 立体性、疏水性、电性HESH[17]2D 立体性、疏水性、电性、氢键属性FASGAI[21]2D 立体性、疏水性、电性ST[22]3D 分子构造、拓扑、几何、连接信息、原子分子电荷-拓扑变化、极化DPPS[15]2D 立体性、疏水性、电性、氢键属性WNH[31]2D 范德华体积、净电荷指数、疏水性VSW[23]2D 分子指数VTSA[19]3D 结构与拓扑参数T[16]3D 结构与拓扑参数
1.3.3 TTPN法
由于多肽数据库中包含了三肽和四肽,而QSAR建模需要多肽的长度相同,因此利用TTPN法将不同长度的多肽进行规格化,最大程度地保留其中的结构信息,使所有的样本都能进行表征建模。
TTPN法的原理是找到最短序列的肽,并以此为标准,从两端将多肽各复制此长度的序列组成新的多肽。比如最短的为三肽,从两端复制长度为3的序列,组合最终得到六肽,这样最大限度的保留了两端的氨基酸信息。TTPN法原理如下式所示。
式中:y表示活性;i为N或C端的氨基酸位置;j为氨基酸描述符变量的个数;Ni,j和Ci,j分别表示N端和C端第i个氨基酸第j个变量。
1.3.4 肽结构特征变量矩阵的建立
氨基酸描述符是将氨基酸结构信息经过统计分析,压缩变量维数,提取为几个主成分。多肽结构表征就是将多肽序列结构用氨基酸描述符表示出一组特征参数,建立肽的结构特征变量矩阵。对于三肽序列而言,变量总数为氨基酸描述符变量数目的3 倍。例如描述符Z含有3 个变量,则表征三肽的变量总数为3×3=9。将锌螯合肽数据库中的每条肽序列从N端向C端表征,形成结构特征变量矩阵X,同时活性数据形成矩阵Y。
1.3.5 PLS建模
采用PLS建模,模型的拟合能力通过累积决定系数R2和估计均方根误差(root mean square error of estimation,RMSEE)评价。R2越高、RMSEE越低,拟合能力越好。同时,采用留一法对模型数据进行内部验证,评价其预测能力,最后计算交互验证决定系数Q2。一个具有较高拟合能力和预测能力的模型应该具备的条件是:R2>0.6,Q2>0.5,对模型外部预测能力的评价是通过Qext2即外部验证系数以及预测方根误差(root mean square of prediction,RMSEP)。分析平台为Matlab 7.6.0,所有的变量经过中心化处理因而具有均等的权重,样本数据库自动随机分配为校正集和预测集。对于校正集,使用交互验证作为内部验证,主成分的个数是基于预测残差平方和得出的。应用Hotelling’s T2和残差分析两种方式考察异常点。
2 结果与分析
2.1 锌螯合肽数据库的建立
锌螯合肽数据库见表2。在QSAR建模过程中,所有的数据按照2∶1随机分配成校正集、预测集,因此校正集数量为37,预测集数量为19。校正集用于建立QSAR模型,同时进行内部验证,而预测集用于外部验证。
表2 锌螯合肽数据库
Table2 Database of zinc-chelating peptides
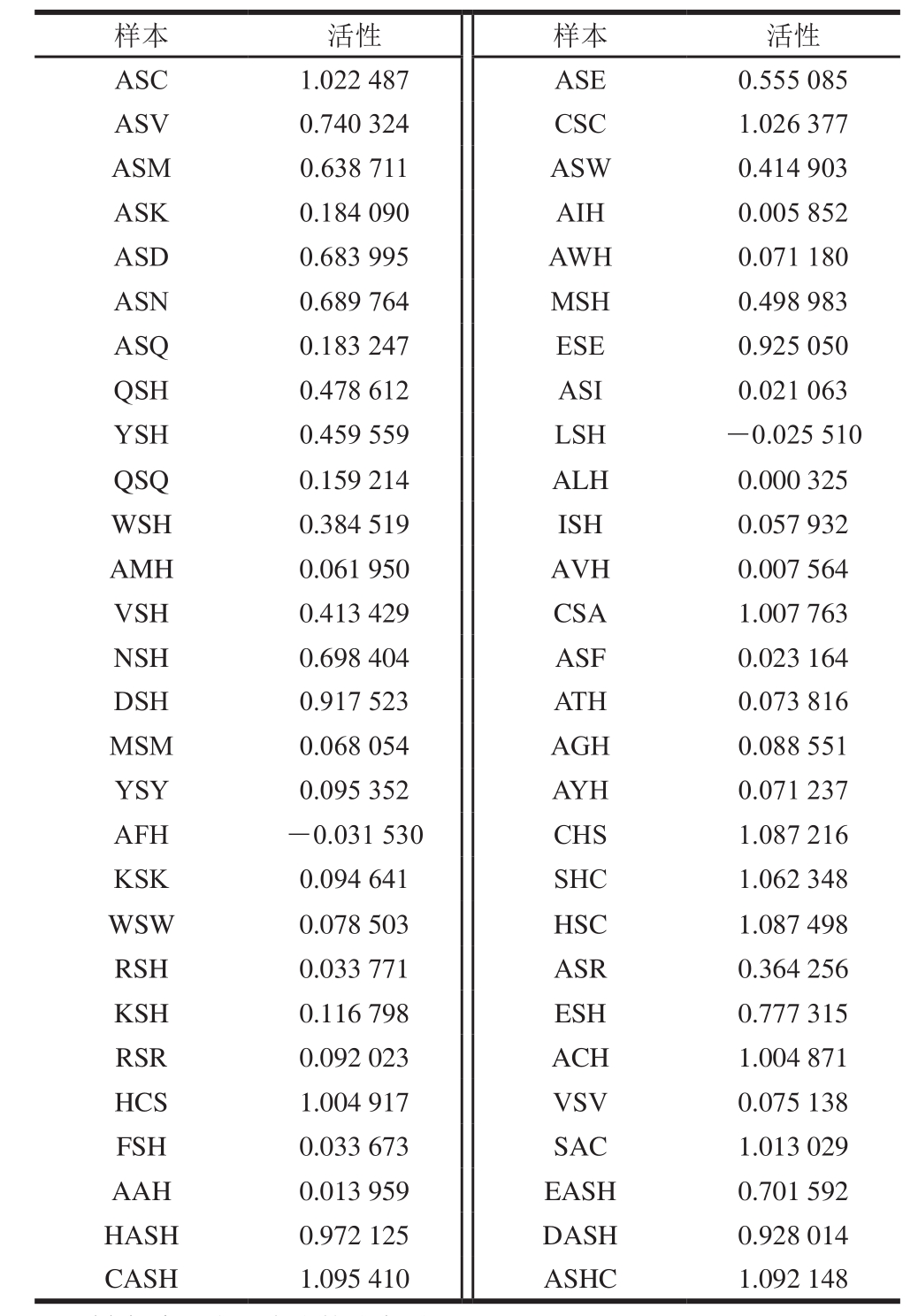
注:样本.来源人工合成的肽序列。
样本 活性 样本 活性ASC 1.022 487 ASE 0.555 085 ASV 0.740 324 CSC 1.026 377 ASM 0.638 711 ASW 0.414 903 ASK 0.184 090 AIH 0.005 852 ASD 0.683 995 AWH 0.071 180 ASN 0.689 764 MSH 0.498 983 ASQ 0.183 247 ESE 0.925 050 QSH 0.478 612 ASI 0.021 063 YSH 0.459 559 LSH -0.025 510 QSQ 0.159 214 ALH 0.000 325 WSH 0.384 519 ISH 0.057 932 AMH 0.061 950 AVH 0.007 564 VSH 0.413 429 CSA 1.007 763 NSH 0.698 404 ASF 0.023 164 DSH 0.917 523 ATH 0.073 816 MSM 0.068 054 AGH 0.088 551 YSY 0.095 352 AYH 0.071 237 AFH -0.031 530 CHS 1.087 216 KSK 0.094 641 SHC 1.062 348 WSW 0.078 503 HSC 1.087 498 RSH 0.033 771 ASR 0.364 256 KSH 0.116 798 ESH 0.777 315 RSR 0.092 023 ACH 1.004 871 HCS 1.004 917 VSV 0.075 138 FSH 0.033 673 SAC 1.013 029 AAH 0.013 959 EASH 0.701 592 HASH 0.972 125 DASH 0.928 014 CASH 1.095 410 ASHC 1.092 148
2.2 锌螯合肽氨基酸描述符以及QSAR模型的选择
选择合适的氨基酸描述符来描述不同种类、长度的多肽是建模过程中关键的一步,匹配的描述符能够最大程度地保留序列中的有用信息。另外,采用PLS建模能够有效解决变量之间的共线问题,即使样本数量小于变量数量也可以分析。研究首先通过TTPN法将不同长度序列的多肽统一长度,经过18 种氨基酸描述符表征量化,并用PLS建模,再通过Hotelling’s T2和残差分析去除异常点后,最终的模型结果如表3所示。
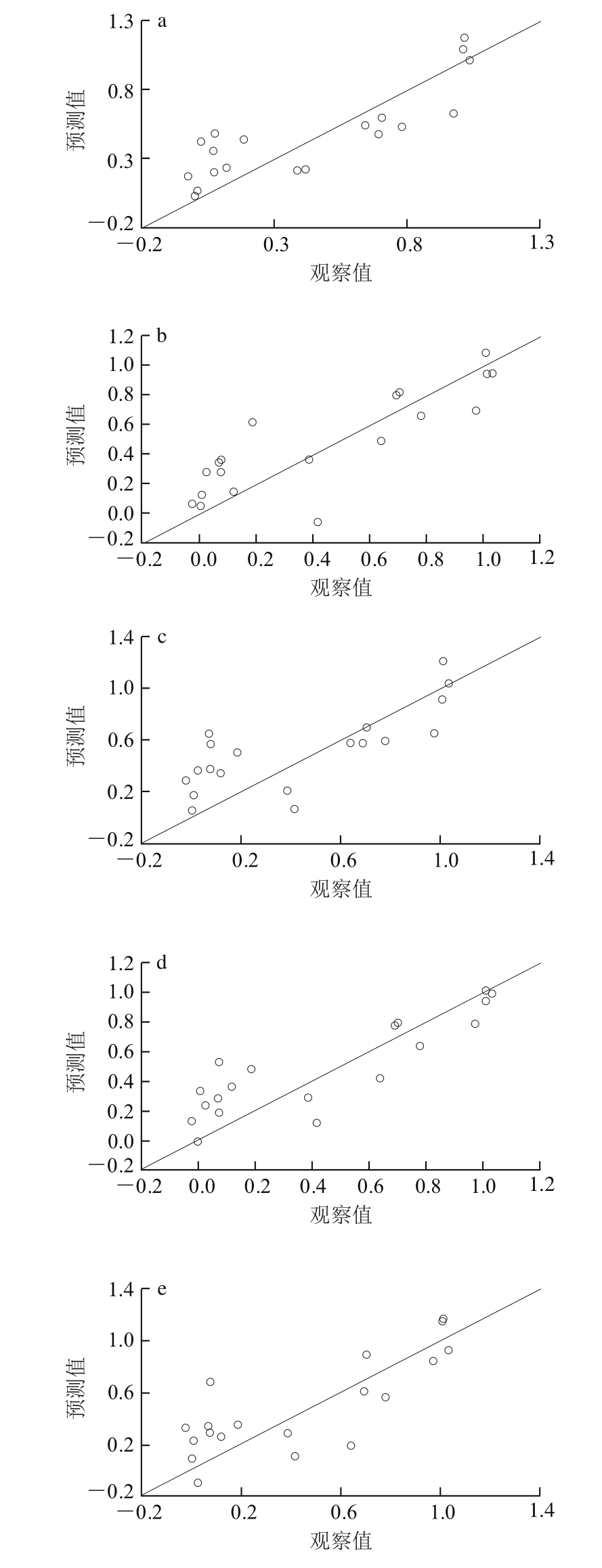
对于一个具有良好拟合能力和预测能力的QSAR模型,相应的标准为:R2>0.6、Q2>0.5。从表3中可以看出18 种氨基酸描述符模型的相关系数,其中描述符Z、C、HESH、FASGAI、ST的模型的相关系数较好(R2为0.684 9~0.827 3;Q2为0.503 9~0.622 3),达到了R2>0.6、Q2>0.5的建模要求。
表3 使用18 种氨基酸描述符的QSAR模型
Table3 QSAR models based on eighteen amino acid descriptors

注:M.主成分数;R2.校正集的累积决定系数,表示校正集中活性的计算值拟合观察值的回归系数;Q2.校正集交互验证的决定系数,表示QSAR模型预测能力和稳定性。
氨基酸描述符 M R2Q2RMSEE Q2extRMSEP 1 0.733 4 0.622 3 0.209 4 0.688 1 0.249 2 ISA-ECI 1 0.384 5 0.112 9 0.318 2 0.416 4 0.382 0 MS-WHIM 1 0.464 7 0.181 5 0.296 7 0.393 6 0.366 9 VSGE 2 0.760 4 0.318 0 0.198 5 0.557 9 0.334 9 VSTV 1 0.597 1 0.309 0 0.259 1 0.516 5 0.337 1 VHSE 1 0.668 9 0.489 7 0.233 4 0.403 2 0.289 7 SZOTT 1 0.706 3 0.458 5 0.219 8 0.473 1 0.298 4 C 1 0.684 9 0.541 9 0.227 6 0.705 1 0.274 5 SVG 1 0.475 4-0.533 2 0.283 5-0.013 0 0.484 6 TEN 1 0.827 2 0.413 0 0.168 6 0.564 9 0.310 7 HESH 1 0.698 7 0.503 9 0.222 6 0.507 1 0.285 7 FASGAI 1 0.827 3 0.602 2 0.168 6 0.717 2 0.255 8 ST 1 0.763 6 0.505 0 0.197 2 0.580 8 0.285 3 DPPS 1 0.658 8 0.480 3 0.236 8 0.532 5 0.292 4 WNH 1 0.633 3 0.441 9 0.245 6 0.595 5 0.303 0 VSW 1 0.714 2 0.461 9 0.216 8 0.446 4 0.297 5 VTSA 1 0.621 9 0.392 4 0.249 4 0.409 9 0.316 1 T 1 0.525 2 0.327 0 0.276 5 0.392 1 0.332 7 Z
同时,使用2D描述符普遍比使用3D描述符具有更好的相关系数和验证结果。在2D描述符的模型中,Z(R2=0.733 4,Q2=0.622 3)、FASGAI(R2=0.827 3,Q2=0.602 2)描述符模型最优,TEN描述符模型虽然具有较高的校正集累积决定系数(R2=0.827 2),但是交互验证系数(Q2=0.413 0)不足以验证模型具有良好预测能力。描述符Z、FASGAI、VHSE、TEN的X矩阵中包括了立体性、疏水性、电性3 个属性,而HESH、DPPS的X矩阵在这3 个变量的基础上增加了氢键属性,其QSAR模型的相关参数有所降低。虽然Z和FASGAI在X矩阵中的属性相同,但它们是从物理化学属性的不同范围中提取,因此具有不同的变量数目(Z:3;FASGAI:6)。另外,从相关系数上比较,描述符FASGAI的模型的拟合能力更好。但是,在3D描述符的模型中,只有C、ST符合建模要求,虽然C描述符模型的R2较小,拟合能力低于ST,但是模型的内部和外部预测能力都高于ST。
Z、C、HESH、FASGAI、ST这5 种描述符模型校正集的观察值与计算值的相关关系结果如图1所示,其中,图1d是使用FASGAI氨基酸描述符的QSAR模型。虽然有系统误差存在,但是对于37 个样本量的校正集,模型拟合较好,大部分落在对角线附近。
2.3 QSAR模型对锌螯合多肽能力的预测
预测集不参与QSAR模型的建立,仅用于模型的外部验证,检测模型的实际预测能力。表3显示出使用FASGAI描述符的决定系数Q2ext为0.717 2、RMSEP为0.255 8,优异于描述符Z、C、HESH、ST等相关系数,而其余的13 种描述符,其Q2ext均小于0.5,表明对应的QSAR模型不具备良好的预测能力。
图2 形象表明了不同描述符模型的预测能力,显示了预测值与观察值之间的关系。其中,图2d是使用FASGAI氨基酸描述符的QSAR模型的外部验证效果。对于19 个样本量的预测集,模型拟合较好,大部分落在对角线附近。基于以上的结果,使用FASGAI氨基酸描述符的QSAR模型最为优良,因而FASGAI氨基酸描述符是最适合用于描述锌螯合肽的结构特征。
2.4 氨基酸描述符的属性与活性之间的关系
在QSAR模型中,每个X变量对活性的贡献大小可以通过测定变量投影重要性指标(variable importance in project,VIP)值和对应的回归系数来判定[32]。
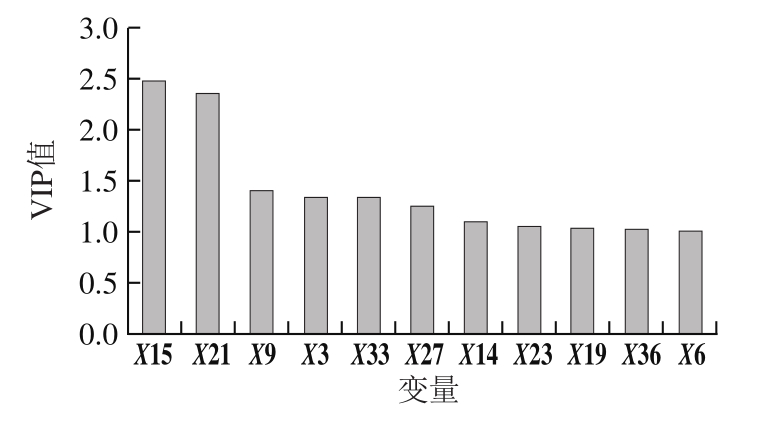
描述符FASGAI有6 个变量,因此通过TTPN处理后得到的六肽总共有36 个变量(6×6=36),表示为X1~X36。图3表明使用描述符FASGAI模型各变量的VIP值,可见变量X15、X21、X9、X3、X33、X27、X14、X23、X19、X36、X6对变量Y有较大的贡献(VIP值>1)。上述11 个VIP值大于1的变量也可表示为N3,3(FASGAI描述符中N端第3个位置上第3个变量)、C3,3(FASFAI描述符中C端第3个位置第3个变量)、N2,3、N1,3、C1,3、C2,3、N3,2、C3,5、C3,1、C1,1、N1,6,因为每一个变量都表示了序列中氨基酸残基的位置以及对应的理化属性。
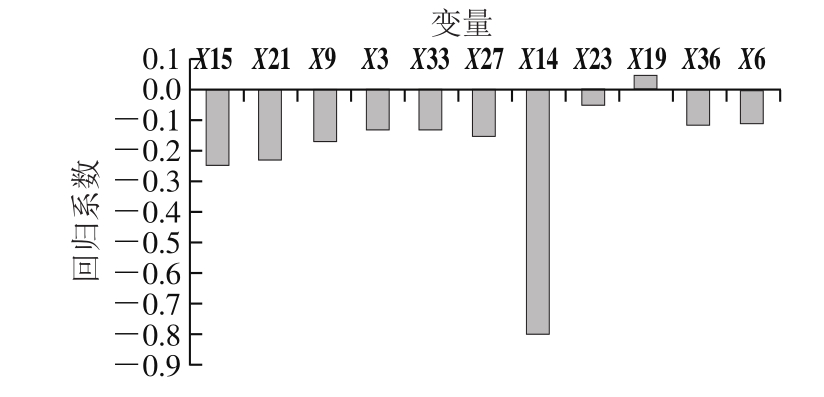
图4 是上述11 个变量的回归系数图,表明变量矩阵X与变量矩阵Y之间的正负相关性:除了X19(C3,1)与锌螯合肽活性呈正相关性,其余X15(N3,3)、X21(C3,3)、X9(N2,3)、X3(N1,3)、X33(C1,3)、X27(C2,3)、X14(N3,2)、X23(C3,5)、X36(C1,1)、X6(N1,6)都与锌螯合肽活性呈负相关性。
氨基酸描述符FASGAI包括3 个物化属性:疏水性、立体性、电性,立体性又具体分为α转角、立体属性、综合特性、局部弹性4 个属性,表4展示了上述11 个变量在序列中的位置以及对应的物化属性。
表4 数据库经过PLS回归分析后的VIP值、回归系数以及位置信息
Table4 Important properties of the peptides after PLS regression analysis

变量 位置 属性 VIP值 回归系数X15 N3 立体属性 2.464 8 -0.245 9 X21 C3 立体属性 2.339 9 -0.228 8 X9 N2 立体属性 1.399 4 -0.168 7 X3 N1 立体属性 1.335 7 -0.131 2 X33 C1 立体属性 1.334 3 -0.129 7 X27 C2 立体属性 1.247 4 -0.151 3 X14 N3 α转角 1.093 8 -0.800 0 X23 C3 局部弹性 1.056 4 -0.049 1 X19 C3 疏水性 1.042 7 0.049 7 X36 C1 疏水性 1.025 4 -0.113 5 X6 N1 电性 1.007 1 -0.108 8
表5 基于VIP值总和确定肽序列中位置及属性重要性
Table5 Importance of structural properties and positions based on calculated VIP values

注:—.不含该属性。
位置 疏水性 α转角 立体属性 综合特性 局部弹性 电子属性 总和C31.042 7—2.339 9—1.056 4—4.439 0 N3—1.093 82.464 8— — —3.558 6 C11.025 4—1.334 3— — —2.359 7 N1— —1.335 7— —1.007 12.342 8 N2— —1.399 4— — —1.399 4 C2— —1.334 3— — —1.334 3
为了更清楚地研究序列中氨基酸残基的位置对锌螯合肽活性的影响,将序列中每个位置上不同属性的VIP值求和(表5)。C3的VIP值最高,表明了序列中C3的位置对活性的影响最大,其中包括了变量X21(C3,3)、X19(C3,1)、X23(C3,5),属性贡献大小依次是立体属性>局部弹性>疏水性,再根据表4中对应的回归系数,说明在C3位置上,低空间属性[X21(C3,3)]、局部弹性[X23(C3,5)]和高疏水性[X19(C3,1)]的氨基酸残基可能提高锌螯合肽活性。N3的VIP值排在C3之后,其中有变量X15(N3,3),X14(N3,2),属性贡献大小依次是空间属性>α转角,并且在N3位置上,低空间属性[X15(N3,3)]、低α转角的氨基酸残基可能提高锌螯合肽活性。VIP值排在第三位的是C1,包括了变量X33(C3,1)、X36(C1,1),属性贡献大小为空间属性>疏水性,表明C1位置上,低空间属性、低疏水性的氨基酸残基可能有利于锌螯合肽活性。第四位是N1,变量包括X3(N1,3)、X6(N1,6),贡献大小为空间属性>电性,在N1位置上,低空间属性、低电性的氨基酸残基可能利于对锌螯合肽活性。VIP值排在最后的分别是N2、C2,只有一个属性空间属性贡献较大,说明在N2、C2位置上低空间属性的氨基酸残基对活性的预测有一定的作用。综上可见,C3、N3在多肽序列中是主要影响锌螯合活性的位置,其中较低立体属性利于锌螯合肽的活性。氨基酸微量元素鳌合物是以二价阳离子与给电子体的氨基酸成配位键,同时又与给电子体的羰基中的氧构成离子键形成五元环或六元环,而较低的立体属性可能能够减少螯合的空间位阻。从直观比对数据库中56 条多肽的活性来看,含有丝氨酸和半胱氨酸的多肽活性较高,丝氨酸、半胱氨酸的R基分别是—CH2—OH、—CH2—SH,具有很低的空间属性、空间位阻。
3 结 论
本研究采用18 种氨基酸描述符,利用PLS统计方法,对56 条合成肽进行统计分析。发现FASGAI、Z、HESH、C和ST这5 种氨基酸描述符对应的QSAR模型相关系数达到建模要求,其中描述符FASGAI相关系数最优,R2=0.827 3、Q2=0.602 2、RMSEE=0.168 6、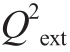 =0.717 2、RMSEP=0.255 8。随后,采用TTPN法对描述符FASGAI所构建的模型进一步分析发现,在菜籽源肽多肽序列中,氨基酸位置对多肽锌螯合活性的影响力为C3>N3>C1>N1>N2>C2。结果初步表明了在菜籽源的锌螯合肽序列中C3、N3、C1这3 个位置的重要性,而且各位置氨基酸立体属性较低的多肽拥有较高的锌螯合活性,这与直观比对数据库中56 条多肽活性的结论相似。
=0.717 2、RMSEP=0.255 8。随后,采用TTPN法对描述符FASGAI所构建的模型进一步分析发现,在菜籽源肽多肽序列中,氨基酸位置对多肽锌螯合活性的影响力为C3>N3>C1>N1>N2>C2。结果初步表明了在菜籽源的锌螯合肽序列中C3、N3、C1这3 个位置的重要性,而且各位置氨基酸立体属性较低的多肽拥有较高的锌螯合活性,这与直观比对数据库中56 条多肽活性的结论相似。
参考文献:
[1] PRASAD A S. Discovery of human zinc def i ciency: 50 years later[J].Journal of Trace Elements in Medicine and Biology, 2012, 26(2/3): 66-69. DOI:10.1016/j.jtemb.2012.04.004.
[2] DENG S G, HUO J C, XIE C. Preparation by enzymolysis and bioactivity of iron complex of fi sh protein hydrolysate (Fe-FPH) from low value fish[J]. Chinese Journal of Oceanology and Limnology,2008, 26(3): 300-306. DOI:10.1007/s00343-008-0300-4.
[3] LÜ Y, LIU Q, BAO X, et al. Identif i cation and characteristics of ironchelating peptides from soybean protein hydrolysates using IMACFe3+[J]. Journal of Agricultural and Food Chemistry, 2009, 57(11):4593-4597. DOI:10.1021/jf9000204.
[4] CARRASCO-CASTILLA J, HERNÁNDEZ-ÁLVAREZ A J,JIMÉNEZ-MARTÍNEZ C, et al. Antioxidant and metal chelating activities of peptide fractions from phaseolin and bean protein hydrolysates[J]. Food Chemistry, 2012, 135(3): 1789-1795.DOI:10.1016/j.foodchem.2012.06.016.
[5] WANG C, LI B, AO J. Separation and identif i cation of zinc-chelating peptides from sesame protein hydrolysate using IMAC-Zn2+and LC-MS/MS[J]. Food Chemistry, 2012, 134 (2): 1231-1238.DOI:10.1016/j.foodchem.2012.02.204.
[6] LV Y, BAO X, LIU H, et al. Purification and characterization of caclium-binding soybean protein hydrolysates by Ca2+/Fe3+immobilized metal affinity chromatography (IMAC)[J].Food Chemistry, 2013, 141(3): 1645-1650. DOI:10.1016/j.foodchem.2013.04.113.
[7] TORRES-FUENTES C, ALAIZ M, VIOQUE J. Aff i nity purif i cation and characterisation of chelating peptides from chickpea protein hydrolysates[J]. Food Chemistry, 2011, 129(2): 485-490. DOI:10.1016/j.foodchem.2011.04.103.
[8] DE LA HOZ L, PONEZI A N, MILANI R F, et al. Iron-binding properties of sugar cane yeast peptides[J]. Food Chemistry, 2014, 142:166-169. DOI:10.1016/j.foodchem.2013.06.133.
[9] LI Y W, LI B. Characterization of structure-antioxidant activity relationship of peptides in free radical systems using QSAR models: key sequence positions and their amino acid properties[J].Journal of Theoretical Biology, 2013, 318: 29-43. DOI:10.1016/j.jtbi.2012.10.029.
[10] JING P, QIAN B, HE Y, et al. Screening milk-derived antihypertensive peptides using quantitative structure activity relationship (QSAR)modeling and in vitro/in vivo studies on their bioactivity[J].International Dairy Journal, 2014, 35(1): 95-101. DOI:10.1016/j.idairyj.2013.10.009.
[11] 卢永忠, 康道乐. 抗菌肽的定量构效关系研究进展[J]. 食品科学,2015, 36(15): 299-304. DOI:10.7506/spkx1002-6630-201515055.
[12] KIDERA A, KONISHI Y, OKA M, et al. Statistical analysis of the physical properties of the 20 naturally occurring amino acids[J].Journal of Protein Chemistry, 1985, 4(1): 23-55. DOI:10.1007/BF01025492.
[13] HELLBERG S, SJOESTROEM M, SKAGERBERG B, et al. Peptide quantitative structure-activity relationships, a multivariate approach[J].Journal of Medicinal Chemistry, 1987, 30: 1126-1135. DOI:10.1021/jm00390a003.
[14] MEI H, LIAO Z H, ZHOU Y, et al. A new set of amino acid descriptors and its application in peptide QSARs[J]. Biopolymers,2005, 80: 775-786. DOI:10.1002/bip.20296.
[15] TIAN F, YANG L, LÜ F, et al. In silico quantitative prediction of peptides binding affinity to human MHC molecule: an intuitive quantitative structure-activity relationship approach[J]. Amino Acids,2009, 36: 535-554. DOI:10.1007/s00726-008-0116-8.
[16] TIAN F F, ZHOU P, LI Z L. T-scale as a novel vector of topological descriptors for amino acids and its application in QSARs of peptides[J]. Journal of Molecular Structure, 2007, 830: 106-115.DOI:10.1016/j.molstruc.2006.07.004.
[17] SHU M, MEI H, YANG S, et al. Structural parameter characterization and bioactivity simulation based on peptide sequence[J]. Qsar &Combinatorial Science, 2009, 28(2): 27-35. DOI:10.1002/qsar.200710169.
[18] LI S Z, FU B. On structural parameterization and molecular modeling of peptide analogues by molecular electronegativity edge vector (VMEE): estimation and prediction for biological activity of dipeptides[J]. Chinese Journal of Chemistry, 2001, 48(1): 937-944.DOI:10.1002/jccs.200100137.
[19] MEI H, ZHOU Y, SUN L L. A new descriptor of amino acids and its application in peptide QSAR[J]. Acta Physico-chimica Sinica, 2004,20(2): 821-825. DOI:10.3866/PKU.WHXB20040808.
[20] 梁桂兆, 周鹏, 周原, 等. 一组新氨基酸描述子用于肽定量构效关系研究[J]. 化学学报, 2006(5): 393-396.
[21] LIANG G, LI Z. Factor analysis scale of generalized amino acid information as the source of a new set of descriptors for elucidating the structure and activity relationships of cationic antimicrobial peptides[J]. QSAR & Combinatorial Science, 2007, 26(3): 754-763.DOI:10.1002/qsar.200630145.
[22] YANG L, SHU M, MA K, et al. ST-scale as a novel amino acid descriptor and its application in QSAM of peptides and analogues[J].Amino Acids, 2010, 38(2): 805-816. DOI:10.1007/s00726-009-0287-y.
[23] TONG J, LIU S, ZHOU P, et al. A novel descriptor of amino acids and its application in peptide QSAR[J]. Journal of Theoretical Biology,2008, 253(3): 90-97. DOI:10.1016/j.jtbi.2008.02.030.
[24] SNEATH P H. Relations between chemical structure and biological activity in peptides[J]. Journal of Theoretical Biology, 1966, 12(2):157-195. DOI:10.1016/0022-5193(66)90112-3.
[25] COLLANTES E R, DUNN W J. Amino acid side chain descriptors for quantitative structure-activity relationship studies of peptide analogs[J]. Journal of Medicinal Chemistry, 1995, 38: 2705-2713.
[26] BRAVI G, GANCIA E, MASCAGNI P, et al. MS-WHIM, new 3D theoretical descriptors derived from molecular surface proper-ties:a comparative 3D QSAR study in a series of steroids[J]. Journal of Computer-Aided Molecular Design, 1997, 11: 79-92. DOI:10.1023/A:1008079512289.
[27] 苏满秀, 王立峰, 代志军, 等. 多肽一级结构表征与抗菌肽QSAM建模[J]. 高等学校化学学报, 2012, 33(11): 2526-2531.
[28] 汪婵, 陈敏, 李博. 芝麻蛋白制备金属螯合肽的酶解工艺研究[J].食品科技, 2011, 36(9): 184-189.
[29] TONG J B, ZHANG S W. A new 3D-descriptor of amino acid and its application in quantitative structure activity relationship of peptide drugs[J]. Acta Physico-chimica Sinica, 2007, 23(1): 37-43.DOI:10.3866/PKU.WHXB20070108.
[30] 丁俊杰, 丁晓琴, 赵立峰, 等. 新型三维氨基酸结构描述符的研究及其在多肽QSAR中的应用[J]. 药学学报, 2005, 40(4): 340-346.
[31] LIN Z, LONG H, BO Z, et al. New descriptors of amino acids and their application to peptide QSAR study[J]. Peptides, 2008, 29(10):1798-1805. DOI:10.1016/j.peptides.2008.06.004.
[32] 陈季旺, 刘珊珊, 蔡广霞, 等. 食物源血管紧张素转化酶抑制三肽的定量构效关系[J]. 食品科学, 2013, 34(9): 19-23. DOI:10.7506/spkx1002-6630-201309005.
Quantitative Structure-Activity Relationship of Zinc-Chelating Peptides by Two-Terminal Position Numbering
HUANG Jingjing1, YU Min2, MA Min3, YAN Yan1, ZHANG Fusheng1, YIN Junfeng1, XIE Ningning1,*
(1. Institute of Agro-products Processing, Anhui Academy of Agricultural Sciences, Hefei 230031, China;2. School of Tea and Food Science, Anhui Agricultural University, Hefei 230036, China;3. School of Science, Anhui Agricultural University, Hefei 230036, China)
Abstract:In order to explore the relationship between the structure and bioactivity of zinc-chelating peptides, quantitative structure-activity relationship (QSAR) models were established. A new method called two-terminal position numbering was proposed to describe the structures of 56 synthetic zinc-chelating peptides with different lengths. Then, these peptides were statistically analyzed using 18 amino acid descriptors and partial least squares regression. Results showed that the correlation coeff i cients of the QSAR models based on 5 amino acids descriptors FASGAI, Z, HESH, C and ST met the requirements.FASGAI was found to be the best among these descriptors (R2= 0.827 3, Q2= 0.602 2, root mean square error of estimation = 0.168 6, Q2ext= 0.717 2, and root mean square error of prediction = 0.255 8). Further analysis of the FASGAI-based model revealed that the influence of amino acid positions in the peptides on their zinc-chelating activity was C3 > N3 > C1 > N1 > N2 > C2. Meanwhile, the bulky properties of amino acid residues influenced the zinc-chelating activity of the peptides. These models have the potential to provide new ideas to explore the quantitative structure-activity relationships of zinc-chelating peptides.
Keywords:rapeseed source; zinc chelating peptide; quantitative structure-activity relationship
HUANG Jingjing, YU Min, MA Min, et al. Quantitative structure-activity relationship of zinc-chelating peptides by twoterminal position numbering[J]. Food Science, 2018, 39(21): 11-17. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-201821002. http://www.spkx.net.cn
黄晶晶, 余敏, 马敏, 等. 锌螯合肽的两端排序法定量构效关系[J]. 食品科学, 2018, 39(21): 11-17. DOI:10.7506/spkx1002-6630-201821002. http://www.spkx.net.cn
引文格式:
文章编号:1002-6630(2018)21-0011-07
文献标志码:A
中图分类号:TS229
DOI:10.7506/spkx1002-6630-201821002
*通信作者简介:谢宁宁(1984—),男,副研究员,博士,研究方向为蛋白资源利用。E-mail:ningxie512@163.com
第一作者简介:黄晶晶(1988—),女,助理研究员,硕士,研究方向为功能食品。E-mail:jingjingcau@163.com
基金项目:国家自然科学基金青年科学基金项目(31401620);安徽省自然科学基金项目(1508085QC54);安徽省农业科学院学科建设项目(16A1234)
收稿日期:2017-06-23



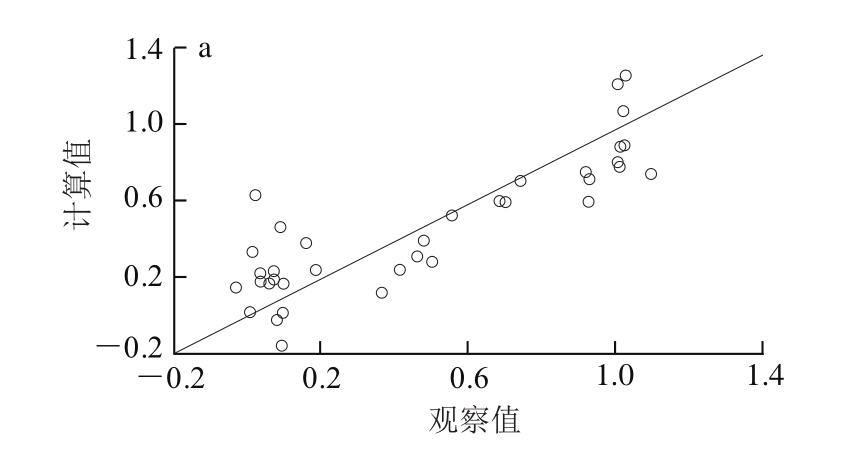



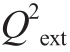 =0.717 2、RMSEP=0.255 8。随后,采用TTPN法对描述符FASGAI所构建的模型进一步分析发现,在菜籽源肽多肽序列中,氨基酸位置对多肽锌螯合活性的影响力为C3>N3>C1>N1>N2>C2。结果初步表明了在菜籽源的锌螯合肽序列中C3、N3、C1这3 个位置的重要性,而且各位置氨基酸立体属性较低的多肽拥有较高的锌螯合活性,这与直观比对数据库中56 条多肽活性的结论相似。
=0.717 2、RMSEP=0.255 8。随后,采用TTPN法对描述符FASGAI所构建的模型进一步分析发现,在菜籽源肽多肽序列中,氨基酸位置对多肽锌螯合活性的影响力为C3>N3>C1>N1>N2>C2。结果初步表明了在菜籽源的锌螯合肽序列中C3、N3、C1这3 个位置的重要性,而且各位置氨基酸立体属性较低的多肽拥有较高的锌螯合活性,这与直观比对数据库中56 条多肽活性的结论相似。