表1 虾酱的物理化学及微生物指标
Table 1 Physicochemical and microbiological parameters of shrimp sauce
戴玲瑛1,齐曼婷2,王立梅3,齐 斌3,*
(1.江南大学食品学院,江苏 无锡 214122;2.谱尼测试集团江苏有限公司,江苏 苏州 215000;
3.常熟理工学院生物与食品工程学院,苏州市食品生物技术重点实验室,发酵工程技术研究中心,江苏 常熟 215500)
摘 要:为更全面地探究虾酱中微生物的多样性,运用高通量测序技术,对虾酱中细菌16S rDNA V3-V4区测序,从而对虾酱中的细菌群落组成和多样性进行分析。研究共获得61 107 条序列,4 358 个操作分类单元,多样性分析表明虾酱中具有高度的微生物群落多样性。虾酱微生物群落组成分析表明虾酱中变形菌门占92.02%,γ-变形菌纲占90.58%,弧菌目占85.91%,弧菌科占85.91%,发光杆菌属占71.94%。研究结果加深了对虾酱中微生物群落组成和多样性的认识,为保证虾酱的质量与安全,提高虾酱的品质,优化虾酱的生产工艺提供理论依据。
关键词:虾酱;微生物多样性;高通量测序技术
虾酱又名虾膏,全国科学技术名词审定委员会将其定义为“毛虾等小型虾类经腌制、捣碎、发酵制成的糊状食品”[1],是我国及东南亚地区的常用的调味料之一[2]。虾酱主要包括对虾酱、白虾酱和麻线酱。捕捞后的麻线虾去杂、加盐,自然发酵1 个月后即可制成麻虾酱。虾酱中含有丰富的蛋白质、钙、类胡萝片素和几丁质[3-4],具有抗氧化活性[5]、降胆固醇、降血压及增强机体免疫力等生物功能活性[6],在功能性食品领域中具有广阔的应用空间。目前为止,对于虾酱的微生物群落组成及结构的研究极少,因此,开展虾酱中微生物群落多样性的研究具有重要意义。
高通量测序技术可对数百万个DNA分子同时测序,对物种的转录组和基因组进行细致而全面的分析[7]。由于其具有高读长、高精度、高通量、无偏性等优势[8],现已被广泛用于对许多发酵食品的微生物群体结构分析,如汾酒、奶酪、开菲尔谷粒等[9-12]。自然发酵的海产品通常会有来自原料以及与海产品的生长环境相关的微生物[13],而细菌是海产品发酵的主要微生物,所以对海产品的细菌微生物多样性进行研究很有必要。目前,海洋多样性的研究是众多学者研究的热点[14],针对于虾的微生物多样性研究也大多集中在虾本身以及虾的养殖环境[15-17],但对于用虾作为原料制成的虾酱的研究少之又少。因此,利用高通量测序技术对虾酱中微生物的多样性及组成进行研究十分重要,可更好地保证生产出安全健康的虾酱产品。
本研究的目的是利用高通量测序方法对虾酱中间产物的细菌群落组成和结构进行分析,更全面地呈现出虾酱中微生物的多样性,剖析各类微生物的结构比例,明确优势菌群,了解优势菌群在虾酱中可能产生的影响,以此更好地保证虾酱的安全与卫生,提高虾酱品质,完善虾酱的生产过程。
虾酱(未经过灭菌处理)由连云港市海娃食品有限公司提供。
E.Z.N.ATMMag-Bind Soil DNA Kit 美国OMEGA公司;Qubit 2.0 DNA检测试剂盒 美国Life公司;Taq DNA Polymerase 美国Thermo公司;Agencourt AMPure XP 美国Beckman公司。
台式离心机 美国Thermo Fisher公司;旋涡混合器海门市其林贝尔仪器制造有限公司;混匀型干式恒温器深圳拓能达科技有限公司;电泳仪、电泳槽 北京六一仪器厂;凝胶成像系统 美国UVP公司;Qubit®2.0荧光计美国Invitrogen公司;聚合酶链式反应(polymerase chain reaction,PCR)仪 美国Bio-Rad公司;移液器 德国Eppendorf公司。
1.3.1 理化指标分析
水分的测定:按照GB 5009.3—2016《食品中水分的测定》,采用直接干燥法;灰分的测定:按照GB 5009.4—2016《食品中灰分的测定》,采用加助灰化剂法;蛋白质的测定:按照GB 5009.5—2016《食品中蛋白质的测定》,采用凯氏定氮法;脂肪的测定:按照GB 5009.6—2016《食品中脂肪的测定》,采用酸水解法;亚硝酸盐的测定:按照GB 5009.33—2016《食品中亚硝酸盐与硝酸盐的测定》,采用分光光度法;挥发性盐基氮的测定:按照GB 5009.228—2016《食品中挥发性盐基氮的测定》,采用半微量定氮法;氨基酸态氮的测定:按照GB 5009.235—2016《食品中氨基酸态氮的测定》,采用酸度计法;食盐的测定:按照GB/T 5009.39—2003《酱油卫生标准的分析方法》,采用直接滴定法;矿物元素的测定:虾酱经微波消解后[18]进行等离子体原子发射光谱仪检测。
1.3.2 微生物指标分析
菌落总数:按照GB 4789.2—2016《食品微生物学检验 菌落总数测定》进行测定;大肠菌群:按照GB 4789.3—2016《食品微生物学检验 大肠菌群计数》进行测定;沙门氏菌:按照GB 4789.4—2016《食品微生物学检验 沙门氏菌检验》进行测定;志贺氏菌:按照GB 4789.5—2016《食品微生物学检验 志贺氏菌检验》进行测定;金黄色葡萄球菌:按照GB 4789.10—2016《食品微生物学检验 金黄色葡萄球菌检验》进行测定;单核细胞增生李斯特氏菌:按照GB 4789.30—2016《食品微生物学检验 单核细胞增生李斯特氏菌检验》进行测定;致病性弧菌:按照SN/T 2564—2010《水产品中致病性弧菌检测》进行测定。
1.3.3 微生物多样性分析
1.3.3.1 DNA提取和PCR扩增
采用E.Z.N.ATMMag-Bind Soil DNA Kit的试剂盒对虾酱样品的细菌基因组DNA进行提取,并用琼脂糖凝胶检测DNA的完整性。利用Qubit 2.0 DNA检测试剂盒对基因组DNA精确定量以确保分离得到足够量的高质量基因组DNA。
将提取到的基因组D N A对细菌1 6 S r D N A的V3-V4区域进行两轮P C R扩增。第1轮扩增以3 4 1 F(C C T A C G G G N G G C W G C A G)和8 0 5 R(GACTACHVGGGTATCTAATCC)为引物。94 ℃预变性3 min,首先以94 ℃变性30 s,45 ℃退火20 s,65 ℃延伸30 s为5个循环;接着进行20个循环的94 ℃变性20 s,55 ℃退火20 s,72 ℃延伸30 s;最后72 ℃延伸5 min。第2轮扩增引入Illumina桥式PCR兼容引物,95 ℃预变性3 min,94 ℃变性20 s,55 ℃退火20 s,72 ℃延伸30 s,5个循环,最后72 ℃延伸5 min。反应结束后配制1%琼脂糖凝胶电泳检测PCR产物。
1.3.3.2 测序数据的处理优化
为得到准确有效的数据,运用Qiime(version 1.8.0)软件对序列进行过滤,标准为:去除3’端的引物接头;去除引物错配比率大于0.1的序列;去除样本中序列尾部质量值小于20的碱基;去除含N的序列;去除长度小于200 bp的序列;去除嵌合体序列,运用Mothur(version 1.30.1)软件中Uchime的方法去除嵌合体序列,保证信息分析质量,得到优质序列。
1.3.3.3 多样性分析
利用Usearch(version 5.2.236)软件对97%的相似水平下的操作分类单元(operational taxonomic units,OTU)进行聚类分析,选择丰度最高的序列作为OTU代表性序列。
利用Mothur(version 1.30.1)软件,可计算出群落分布丰度指数Chao指数和ACE指数以及群落分布多样性指数Shannon指数、Simpson指数和Coverage指数。
1.3.3.4 群落结构分析
利用BLAST将OTU序列与对应数据库进行比对,筛选出OTU序列最佳比对结果,并对比对结果进行过滤,去除相似度小于10%且Coverage小于10%的序列,数据库为:http://ncbi.nlm.nih.gov/。根据分类学分析结果,可得到样品在不同分类水平上(门、纲、目、科、属)的分类学比对情况,主要包括:样品中含有何种微生物;样品中微生物的相对丰度。
表1 虾酱的物理化学及微生物指标
Table 1 Physicochemical and microbiological parameters of shrimp sauce

由表1可知,虾酱中含有7.924%的蛋白质和12.775 mg/L的Ca元素,说明本实验原料中含有丰富的人体所必须的蛋白质和无机盐等营养成分。另外,虾壳中的不溶性钙在经过发酵过程中所产生的乳酸和醋酸等有机酸作用后可形成大量的可溶性钙更有利于人体的吸收[19]。虾酱中的挥发性盐基氮和氨基酸态氮分别为4.233 mg/100 g和0.361%,符合国家卫生安全标准。虾酱发酵过程中,硝酸盐在微生物和酶的作用下产生亚硝酸盐,亚硝酸盐是一种致癌性物质[20],本实验中虾酱的亚硝酸盐含量为0.530 mg/kg,远低于国际酱腌菜卫生安全标准(20 mg/kg)。挥发性盐基氮反映了水产品原料的新鲜度[21],本实验中虾酱的挥发性盐基氮含量较低,表明食品新鲜度很高,可以安全食用。张德友[22]采用低盐技术发酵生产的虾酱,食盐质量分数为15%~18%,而本实验中虾酱的食盐质量分数为7.254%,更符合发酵食品的低盐标准,可满足消费者对低盐健康食品的需求。本实验的虾酱中未检出有害的重金属Pb、Cd、As以及致病菌,经过灭菌而制成的成品虾酱中的菌落总数小于10 CFU/g。因此,本实验所用虾酱的理化指标和微生物指标均符合食用安全标准。
对基因组DNA进行PCR扩增(图1),扩增后得到清晰明显的条带,DNA浓度达到扩增的要求,可用于后续的研究分析,委托生工生物工程(上海)有限公司进行高通量测序。
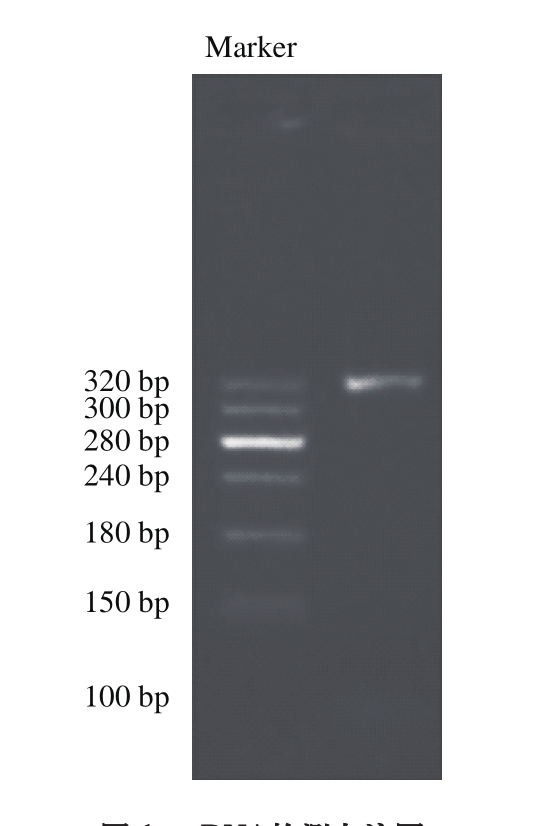
图1 DNA检测电泳图
Fig. 1 Electrophoresis of PCR amplified genomic DNA from bacteria in shrimp sauce
通过细菌的16S rDNA基因V3-V4测序,共获得61 255 个序列,平均长度479.19 bp。质控之后,共有61 107 个序列,平均长度427.20 bp。所有序列在97%的相似水平下进行OTU统计分析,共获得4 358 个OTU。
表2 虾酱样品的序列丰度和微生物多样性
Table 2 Sequence abundance and microbial diversity in shrimp sauce

虾酱样品的ACE和Chao1指数可以反映出群落分布丰度,由表2可以看出,虾酱样品中的微生物群落的种群多样性和总体丰度较高。Shannon和Simpson以及Coverage指数可以反映出群落分布多样性,其中Shannon指数越大,表明微生物群落多样性越高,而Simpson指数越大,表明微生物群落多样性越低,Coverage指数则反映了样品的覆盖率[23]。由表2可以看出,虾酱中微生物群落多样性处于较高的水平,覆盖率可达94%,说明样品覆盖率高,样品中序列没有被测出的概率较低,测序结果具有真实性。
本实验中共鉴定出虾酱的1 4 个细菌门,分别为变形菌门(Proteobacteria)、梭杆菌门(Fusobacteria)、厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)、放线菌门(Actinobacteria)、酸杆菌门(Acidobacteria)、浮霉菌门(Planctomycetes)、疣微菌门(V e r r u c o m i c r o b i a)、芽单胞菌门(Gemmatimonadetes)、绿弯菌门(Chloroflexi)、螺旋菌门(Spirochaetes)、Candidatus Saccharibacteria、Candidate division WPS-1、未分类菌群(unclassified)。对虾酱主要组成的14 个门进行门水平菌群比例分布绘图,结果如图2所示。由图2、3可知,虾酱中变形菌门为优势的菌群门,达到92.02%,而在变形菌门中γ-变形菌纲比例最高,是主要的菌群纲。梭杆菌门和厚壁菌门比例较低,仅为3.97%和3.38%。变形菌门是革兰氏阴性菌的主要组成部分,在人体[24]、虾类[25]、蟹类[26-27]中广泛存在。Jack等[28]研究了普利茅斯沿海地区的海水,发现变形菌门是优势菌群门。对养殖池塘中的水体研究也发现变形菌门为优势类群[29-31]。本研究所用的麻虾来源于黄海,其中主要的细菌类群是变形菌门[32],因此以变形菌门为虾酱中占主导作用的菌群可能与其原材料来源相关。变形菌门是细菌中最大的一门,包括了很多的病原菌,如大肠杆菌、沙门氏菌、幽门螺杆菌等[33],而变形菌带菌率的高低主要和食品的新鲜、运输和贮存的卫生条件密切相关。因此,变形菌对虾酱的品质影响极大,需要在麻虾酱的发酵生产和储存过程中严格控制变形菌的生长,实现生产标准化和规范化。
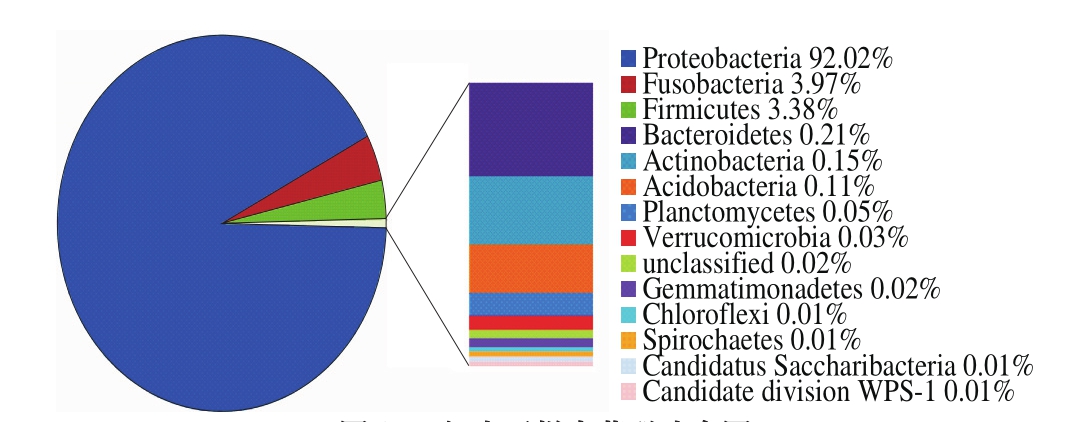
图2 门水平样本菌群分布图
Fig. 2 Distribution frequency at phylum level of microbial communities from shrimp sauce
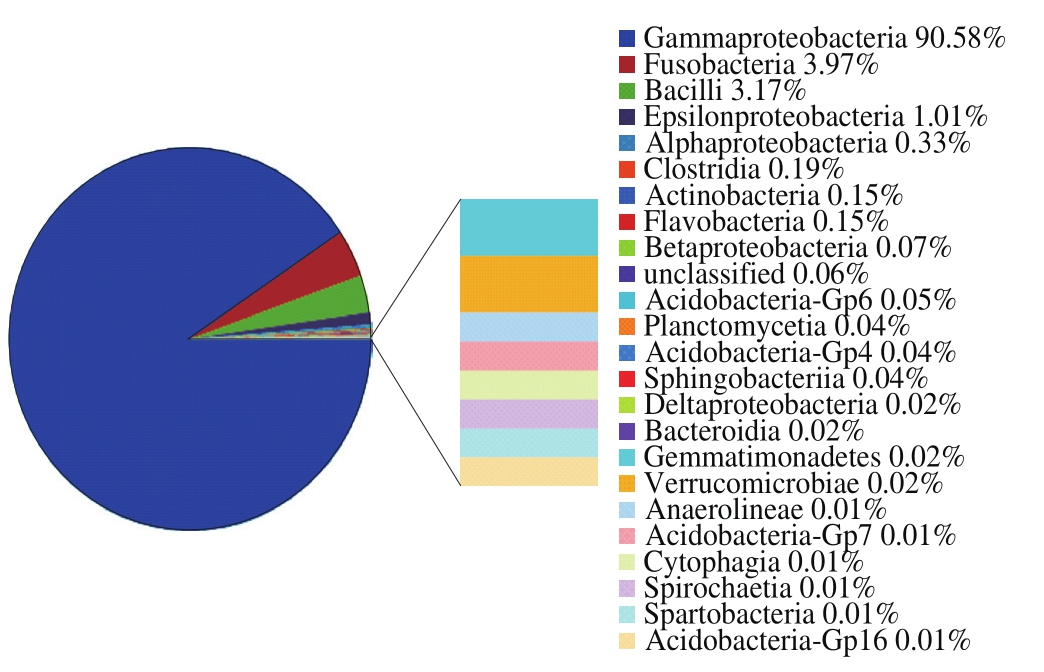
图3 纲水平样本菌群分布图
Fig. 3 Distribution frequency at class level of microbial communities from shrimp sauce
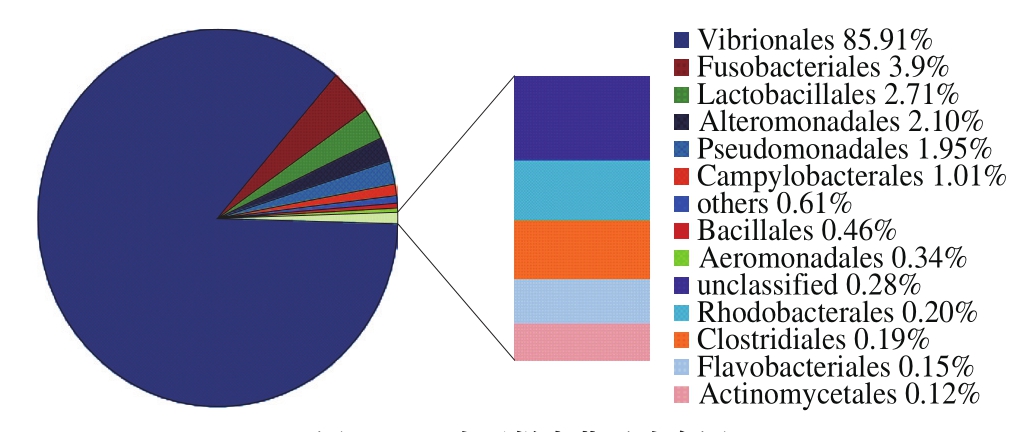
图4 目水平样本菌群分布图
Fig. 4 Distribution frequency at order level of microbial communities from shrimp sauce
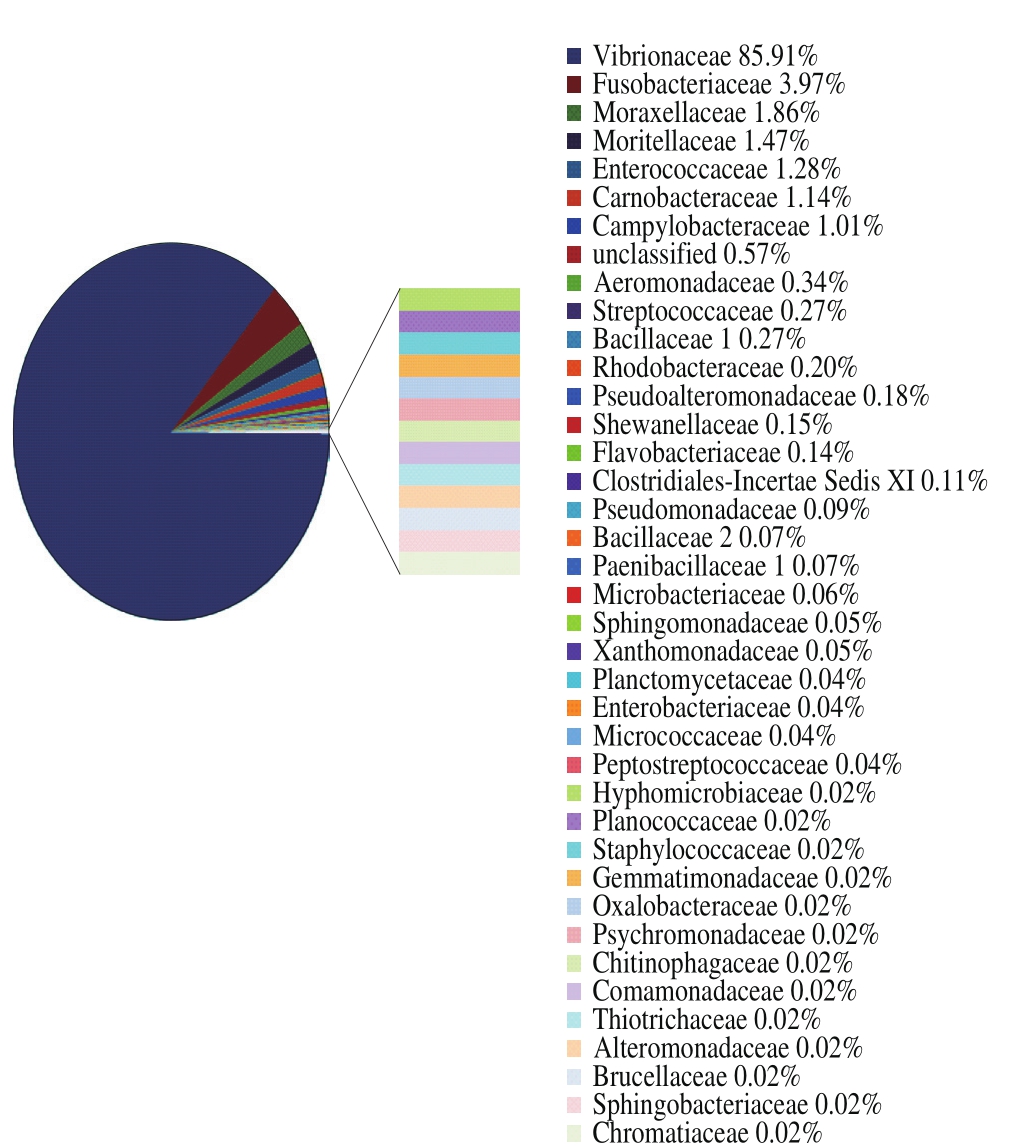
图5 科水平样本菌群分布图
Fig. 5 Distribution frequency at family level of microbial communities from shrimp sauce
如图4所示,本研究中共得到3 4 种细菌目,主要有弧菌目(V i b r i o n a l e s)、梭杆菌目(Fusobacteriales)、乳杆菌目(Lactobacillales)、交替单胞菌目(Alteromonadales)、假单胞菌目(Pseudomonadales)、弯曲菌目(Campylobacterales)和一些其他的菌群目。其中弧菌目比例达到85.91%,而梭杆菌目和乳杆菌目分别为3.97%和2.71%。如图5所示,在科水平上,弧菌目和梭杆菌目中分别为弧菌科和梭杆菌科,而乳杆菌目中有1.28%的肠菌科、1.14%的肉杆菌科以及0.27%的链球菌科。因此,在目和科的水平上,弧菌目和弧菌科是最占优势的菌群。弧菌通常是海产品引起食物中毒的主要微生物源[34],因此虾酱的发酵过程需严格控制发酵条件,避免弧菌的滋生。本实验中未检测出弧菌,说明虾酱具有极高的安全性。
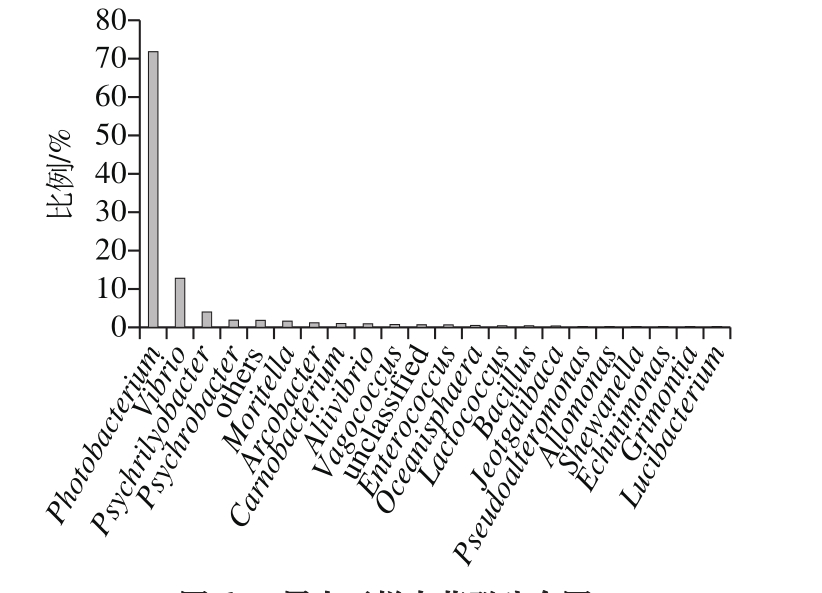
图6 属水平样本菌群分布图
Fig. 6 Distribution frequency at genus level of microbial communities from shrimp sauce
由图6可知,本研究共获得20 个主要的细菌属,分别是发光杆菌属(Photobacterium)、弧菌属(Vibrio)、冷泥杆属(Psychrilyobacter)、嗜冷菌属(P s y c h r o b a c t e r)、摩替亚氏菌属(Moritella)、弓形杆菌属(Arcobacter)、肉食杆菌属(Carnobacterium)、漫游球菌属(Vagococcus)、肠球菌属(Enterococcus)、乳球菌属(Lactococcus)、芽孢杆菌属(Bacillus)、假交替单胞菌属(Pseudoalteromonas)、异单胞菌属(Allomonas)、希瓦氏菌属(Shewanella)、射光杆菌属(Lucibacterium)、Aliivibrio、Oceanisphaera、Jeotgalibaca、Echinimonas、Grimontia。其中发光杆菌属是优势菌属,所占比例达到71.94%;其次是弧菌属,比例为12.54%;而冷泥杆属、嗜冷菌属、摩替亚氏菌属和弓形杆菌属比例较低,仅有3.93%、1.82%、1.47%和1.01%。Wanilada等[35]研究不同生长阶段下黑虎虾肠道细菌多样性,发现在黑虎虾幼虫期的后期发光杆菌属是主要的细胞菌属,而整个幼虫期是以弧菌属为主,发光杆菌属和弧菌属在虾肠道中普遍存在[36-37]。对凡纳滨对虾肠道和三疣梭子蟹肠道进行多样性分析后也可得出相同的结论[38-39]。另外,发光杆菌属能产生抗菌物质,具有一定的抑菌活性,因此可以在一定程度上产生特异生物活性代谢物,抑制虾酱中病原菌的生长[40]。但也有研究发现,发光杆菌属是引起食品腐败的主要菌属[41],且发光杆菌可在高盐的环境中产生生物胺,食品中生物胺含量达到1 000 mg/kg时便会对人体健康造成极大的危害[42]。高温对降低生物胺含量具有显著效果,因此在虾酱罐装前,可通过适当高温煮制,降低虾酱中生物胺含量[43]。
本研究利用高通量测序技术分析自然发酵产品虾酱中的细菌群落结构及多样性,研究共获得61 107 条序列,4 358 个OTU。对虾酱进行微生物多样性分析后共得到26 个门、41 个纲、59 个目、124 个科、315 个属。虾酱中的优势菌群分别是变形菌门、γ-变形菌纲、弧菌目、弧菌科、发光杆菌属和弧菌属,就门水平来说,变形菌门是最占优势的菌群门,占到92.02%,在虾中广泛存在,且与虾的养殖环境密切相关。通过探讨虾酱中的细菌群落组成,可以把握虾酱中优势菌群的分布情况,了解到优势菌群会对虾酱的质量与安全以及品质产生一定的影响。其中变形菌门、弧菌和发光杆菌在某种程度上都对虾酱具有潜在的危害性,因此为控制这些安全隐患,可以从麻线虾的处理、虾酱的发酵环境、罐装温度等生产工艺条件进行改进,将生产标准化、规范化以降低风险。这对于保证虾酱的质量与安全,提高虾酱的品质和营养价值具有重要意义。
参考文献:
[1] 康宪, 戴萍, 林捷, 等. 传统虾酱生产技术的研究进展[J]. 中国酿造,2013, 32(9): 21-23.
[2] DUAN S, HU X, LI M, et al. Composition and metabolic activities of the bacterial community in shrimp sauce at the fl avor-forming stage of fermentation as revealed by metatranscriptome and 16S rRNA gene sequencings[J]. Journal of Agricultural and Food Chemistry, 2016,64(12): 2591-2603. DOI:10.1021/acs.jafc.5b05826.
[3] MAO X, ZHANG J, KAN F, et al. Antioxidant production and chitin recovery from shrimp head fermentation with Streptococcus thermophilus[J]. Food Science and Biotechnology, 2013, 22(4): 1023-1032. DOI:10.1007/s10068-013-0179-5.
[4] MAO X, LIU P, HE S, et al. Antioxidant properties of bio-active substances from shrimp head fermented by Bacillus licheniformis OPL-007[J]. Applied Biochemistry and Biotechnology, 2013, 171(5):1240-1252. DOI:10.1007/s12010-013-0217-z.
[5] PONGSETKUL J, BENJAKUL S, SAMPAVAPOL P, et al. Chemical compositions, sensory and antioxidative properties of salted shrimp paste (Ka-pi) in Thailand[J]. International Food Research Journal,2015, 22(4): 1454-1465.
[6] HALDER S K, ADAK A, MAITY C, et al. Exploitation of fermented shrimp-shells hydrolysate as functional food: assessment of antioxidant, hypocholesterolemic and prebiotic activities[J]. Indian Journal of Experimental Biology, 2013, 51(12): 924-934.
[7] SOON W W, HARIHARAN M, SNYDER M P. High-throughput sequencing for biology and medicine[J]. Molecular Systems Biology,2013, 9(1): 640-648. DOI:10.1038/msb.2012.61.
[8] DI BELLA J M, BAO Y, GLOOR G B, et al. High throughput sequencing methods and analysis for microbiome research[J]. Journal of Microbiological Methods, 2013, 95(3): 401-414.
[9] ERCOLINI D. High-throughput sequencing and metagenomics:moving forward in the culture-independent analysis of food microbial ecology[J]. Applied and Environmental Microbiology, 2013, 79(10):3148-3155. DOI:10.1128/AEM.00256-13.
[10] LI X R, MA E B, YAN L Z, et al. Bacterial and fungal diversity in the starter production process of Fen liquor, a traditional Chinese liquor[J].Journal of Microbiology, 2013, 51(4): 430-438. DOI:10.1007/s12275-013-2640-9.
[11] WOLFE B E, BUTTON J E, SANTARELLI M, et al. Cheese rind communities provide tractable systems for in situ and in vitro studies of microbial diversity[J]. Cell, 2014, 158(2): 422-433. DOI:10.1016/j.cell.2014.05.041.
[12] GAO J, GU F, HE J, et al. Metagenome analysis of bacterial diversity in Tibetan kefir grains[J]. European Food Research and Technology,2013, 236(3): 549-556. DOI:10.1007/s00217-013-1912-2.
[13] JUNG J Y, LEE S H, LEE H J, et al. Microbial succession and metabolite changes during fermentation of saeu-jeot: traditional Korean salted seafood[J]. Food Microbiology, 2013, 34(2): 360-368.
[14] VICK-MAJORS T J, ACHBERGER A, SANTIBÁNEZ P, et al.Biogeochemistry and microbial diversity in the marine cavity beneath the McMurdo Ice Shelf, Antarctica[J]. Limnology and Oceanography,2016, 61(2): 572-586. DOI:10.1002/lno.10234.
[15] REZAEE S, FARAHMAND H, NEMATOLLAHI M A. Genetic diversity status of Pacific white shrimp (Litopenaeus vannamei) using SSR markers in Iran[J]. Aquaculture International, 2016, 24(2): 479-489. DOI:10.1007/s10499-015-9939-y.
[16] VASEEHARAN B, RAJAKAMARAN P, JAYASEELAN D, et al.Molecular markers and their application in genetic diversity of penaeid shrimp[J]. Aquaculture International, 2013, 21(2): 219-241.DOI:10.1007/s10499-012-9582-9.
[17] RAJAKUMARAN P, VASEEHARAN B. Survey on Penaeidae shrimp diversity and exploitation in south east coast of India[J]. Fisheries and Aquaculture Journal, 2015. DOI:10.4172/ 2150-3508.1000103.
[18] JARAPALA S R, KANDLAKUNTA B, THINGNGANING L.Evaluation of trace metal content by ICP-MS using closed vessel microwave digestion in fresh water fi sh[J]. Journal of Environmental and Public Health, 2014, 2014(2): 201506. DOI:10.1155/2014/201506.
[19] 宋翔, 王锡昌, 刘源. 南极磷虾干虾头与虾壳的化学成分分析[J].食品工业科技, 2013, 34(18): 347-335. DOI:10.13386/j.issn1002-0306.2013.18.095.
[20] JUNG W Y, LEE H J, JEON C O. Halomonas garicola sp. nov.,isolated from saeu-jeot, a Korean salted and fermented shrimp sauce[J]. International Journal of Systematic and Evolutionary Microbiology, 2016, 66(2): 731-737. DOI:10.1099/ijsem.0.000784.
[21] JINADASA B. Determination of quality of marine fishes based on total volatile base nitrogen test (TVB-N)[J]. Nature and Science, 2014,5(12): 106-111.
[22] 张德友. 低盐无腥虾酱: 02146366.2[P/OL]. 2003-5-7[2017-5-20].http://dbpub.cnki.net/grid2008/dbpub/detail.aspx?dbcode=SCPD&dbn ame=SCPD0003&filename=CN1415235.
[23] 张敏, 张艳, 黄丽丽, 等. 基于16S rDNA高通量测序方法比较新疆西北部地区乳品中微生物的多样性[J]. 食品科学, 2017, 38(20): 27-33.DOI:10.7506/spkx1002-6630-201720005.
[24] QIN N, YANG F, LI A, et al. Alterations of the human gut microbiome in liver cirrhosis[J]. Nature, 2014, 513: 59-78. DOI:10.1038/nature13568.
[25] XIONG J, WANG K, WU J, et al. Changes in intestinal bacterial communities are closely associated with shrimp disease severity[J].Applied Microbiology and Biotechnology, 2015, 99(16): 6911-6919.DOI:10.1007/s00253-015-6632-z.
[26] LI S, SUN L, WU H, et al. The intestinal microbial diversity in mud crab (Scylla paramamosain) as determined by PCR-DGGE and clone library analysis[J]. Journal of Applied Microbiology, 2012, 113(6):1341-1351. DOI:10.1111/jam.12008.
[27] ZHANG M, SUN Y, CHEN L, et al. Symbiotic bacteria in gills and guts of Chinese mitten crab (Eriocheir sinensis) differ from the free-living bacteria in water[J]. PLoS ONE, 2016, 11(1):e0148135-e0148147. DOI:10.1371/journal.pone.0148135.
[28] GILBERT J A, STEELE J A, CAPORASO J G, et al. Defining seasonal marine microbial community dynamics[J]. The ISME Journal,2012, 6(2): 298-308.
[29] 李晓. 团头鲂池塘养殖系统菌群结构及组成多样性研究[D]. 南京:南京农业大学, 2014: 17-18.
[30] 王春忠, 林国荣, 严涛, 等. 长毛对虾海水养殖环境以及虾肠道微生物群落结构研究[J]. 水产学报, 2014, 38(5): 706-712. DOI:10.3724/SP.J.1231.2014.49053.
[31] 窦妍, 丁君, 王轶南, 等. 黄、渤海春季刺参肠道及养殖池塘细菌菌群的多样性[J]. 大连海洋大学学报, 2014, 29(6): 572-576.DOI:10.3969/J.ISSN.2095-1388.2014.06.006.
[32] 徐君怡, 靳艳, 虞星炬, 等. 黄海繁茂膜海绵中微生物多样性的研究[J]. 微生物学报, 2004, 44(5): 576-579. DOI:10.13343/j.cnki.wsxb.2004.05.004.
[33] KILINÇ B, BESLER A. Seafood toxins and poisonings[J]. Turkish Journal of Aquatic Sciences, 2015, 30(1): 35-52. DOI:10.18864/iujfas.60084.
[34] 韩小龙, 张海燕, 曹明秀, 等. 我国海产品中副溶血性弧菌的污染现状与控制策略分析[J]. 食品与发酵工业, 2015, 41(7): 263-267.DOI:10.13995/j.cnki.11-1802/ts.201507048.
[35] RUNGRASSAMEE W, KLANCHUI A, CHAIYAPECHARA S, et al.Bacterial population in intestines of the black tiger shrimp (Penaeus monodon) under different growth stages[J]. PLoS ONE, 2013, 8(4):e60802-e60813. DOI:10.1371/journal.pone.0060802.
[36] RUNGRASSAMEE W, KLANCHUI A, MAIBUNKAEW S, et al.Characterization of intestinal bacteria in wild and domesticated adult black tiger shrimp (Penaeus monodon)[J]. PLoS ONE, 2014, 9(3):e91853-e91864. DOI:10.1371/journal.pone.0091853.
[37] CHAIYAPECHARA S, RUNGRASSAMEE W, SURIYACHAY I,et al. Bacterial community associated with the intestinal tract of P. monodon in commercial farms[J]. Microbial Ecology, 2012, 63(4):938-953. DOI:10.1007/s00248-011-9936-2.
[38] 温崇庆, 何瑶瑶, 薛明, 等. 高通量测序分析提取引起的对虾肠道菌群结构偏差[J]. 微生物学报, 2016, 56(1): 130-142. DOI:10.13343/j.cnki.wsxb.20150164.
[39] 曾廷兰, 叶央芳, 母昌考, 等. 三疣梭子蟹肠道菌群结构及代谢表型分析[J]. 分析化学, 2016, 44(12): 1867-1873. DOI:10.11895/j.issn.0253-3820.160432.
[40] 肖尧, 杨秀芬, 杨怀文. 致病杆菌和发光杆菌抗菌代谢产物研究进展[J]. 中国生物防治学报, 2011, 27(4): 553-558. DOI:10.16409/j.cnki.2095-039x.2011.04.022.
[41] CHAILLOU S, CHAULOT-TALMON A, CAEKEBEKE H, et al.Origin and ecological selection of core and food-specific bacterial communities associated with meat and seafood spoilage[J]. The ISME Journal, 2015, 9(5): 1105-1118.
[42] 王光强, 俞剑燊, 胡健, 等. 食品中生物胺的研究进展[J]. 食品科学,2016, 37(1): 269-278. DOI:10.7506/spkx1002-6630-201601046.
[43] 李志军, 吴永宁, 薛长湖. 生物胺与食品安全[J]. 食品与发酵工业,2004, 30(10): 84-91. DOI:10.13995/j.cnki.11-1802/ts.2004.10.019.
Bacterial Community Structure and Diversity in Unsterilized Shrimp Sauce
DAI Lingying1, QI Manting2, WANG Limei3, QI Bin3,*
(1. School of Food Science and Technology, Jiangnan University, Wuxi 214122, China;2. Pony Testing International Group, Suzhou 215000, China;3. Key Laboratory of Food and Biotechnology of Suzhou, Fermentation Engineering Technology Research Center,College of Biological and Food Engineering, Changshu Institute of Technology, Changshu 215500, China)
Abstract:This study aimed to fully understand the microbial diversity in shrimp sauce. The V3-V4 region of the bacterial 16S rDNA gene from shrimp sauce was sequenced by high-throughput sequencing, and the composition and diversity of the bacterial communities were analyzed. A total of 61 107 effective sequences and 4 358 operational taxonomic units (OTUs)were obtained. The result of diversity analysis showed that the semi-finished product had a high microbial community diversity. The analysis of bacterial composition indicated that Proteobacteria, Gammaproteobacteria, Vibrionales,Vibrionaceae and Photobacterium accounted for 92.02%, 90.58%, 85.91%, 85.91% and 71.94%, respectively. This research gives a better understanding of the microbial communities in shrimp sauce, which can provide a theoretical basis for ensuring the quality and safety, improving the quality and optimizing the production of shrimp sauce.
Keywords:shrimp sauce; microbial diversity; high-throughput sequencing
DAI Lingying, QI Manting, WANG Limei, et al. Bacterial community structure and diversity in unsterilized shrimp sauce[J]. Food Science,2018, 39(22): 173-178. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-201822027. http://www.spkx.net.cn
引文格式:戴玲瑛, 齐曼婷, 王立梅, 等. 虾酱中间产物细菌群落结构及多样性[J]. 食品科学, 2018, 39(22): 173-178. DOI:10.7506/spkx1002-6630-201822027. http://www.spkx.net.cn
文章编号:1002-6630(2018)22-0173-06
文献标志码:A
中图分类号:Q935
DOI:10.7506/spkx1002-6630-201822027
*通信作者简介:齐斌(1965—),男,教授,博士,研究方向为食品生物技术。E-mail:qibin65@126.com
第一作者简介:戴玲瑛(1994—),女,硕士研究生,研究方向为食品生物技术。E-mail:dailingying666@126.com
基金项目:江苏省科技厅重点研发计划项目(BE2017316;BE2017326)
收稿日期:2017-08-31