单增李斯特菌(Listeria monocytogenes)是一种重要的食源性致病菌,广泛存在于各种食品基质之中,尤其在低温下具很强的生长能力[1],可引起人畜共患的疾病,致死率高达20%~70%[2]。副溶血性弧菌(Vibrio parahaemolyticus)是水产品中常见的病原菌,在夏季其污染率高达90%[3-4],误食被该菌感染的水产品易导致败血症和肠胃炎等疾病的发生[5]。研究表明,这两种食源性致病菌常共存于同一食品体系中[6],加工、贮运和销售过程中的不规范操作都能增加其交叉感染的概率[7-8],从而导致极大的食品安全隐患。预测微生物模型是一种重要的工具,可用于描述特定状况下微生物的生长、死亡及存活情况,广泛应用于食源性致病菌患病风险的评估及控制。传统的预测微生物学模型基于微生物培养的方法,具有灵敏度低、耗时长、操作繁琐等缺陷,而且往往只局限于单一微生物存在的生长失活情况,明显不符合实际情况。随着科学技术的发展,研究人员将先进的实时荧光定量聚合酶链式反应(quantitative real-time polymerase chain reaction,qPCR)、变性梯度凝胶电泳(denaturing gradient gel electrophoresis,DGGE)、DNA探针法、宏基因组学测序技术等分子生物学技术应用于预测微生物学研究[9],构建了新型的“分子预测模型”,该模型具有极高的特异性,可同时描述多株致病菌并存状态下的生长趋势,有效地弥补了传统预测微生物模型的不足。
现阶段,关于单增李斯特菌和副溶血性弧菌的传统预测模型的研究已相对比较成熟[10-12],也有部分学者构建了单增李斯特菌和副溶血性弧菌各自的分子预测模型[5,13-16]。但是,关于单增李斯特菌与副溶血性弧菌混合共存于同一环境下的研究还鲜有报道。因此,本研究运用qPCR技术,模拟超市、冷链的温度环境(4 ℃和10 ℃),监测即食虾中单增生李斯特菌与副溶血性弧菌共存时菌量的动态变化,旨在描述低温环境下单增生李斯特菌与副溶血性弧菌在即食虾中的命运变化,建立混合致病菌共存的分子预测模型,为有效控制这两种食源性致病菌提供可靠的技术支持。
南美白对虾购自上海浦东新区古棕路农工商超市,于-80 ℃保存;qPCR引物由上海生工生物工程有限公司合成(单增李斯特菌:hlyA-F:5’-ACTTCGGCGCAATCAGTGA-3’,hlyA-R:5’-TTGCA ACTGCTCTTTAGTAACAGCTT-3’[17];副溶血性弧菌:tlh-F:5’-ACTCAACACAAGAAGAGATCGACAA-3’,tlh-R:5’-GATGAGCGGTTGATGTCCAA-3’)[18]。
硫代硫酸盐柠檬酸盐胆盐蔗糖琼脂(thiosulfate citrate bile salts sucrose agar,TCBS)培养基、PALCAM琼脂、胰蛋白胨大豆肉汤(tryptic soy broth,TSB)培养基 北京陆桥技术有限责任公司;2×SYBR Green Master Mix 瑞士罗氏公司;细菌基因组DNA提取试剂盒 天根生化科技有限公司。
离心机 德国Eppendorf公司;BagMixer 400VW型拍打式均质器 法国Interscience公司;高精度恒温培养箱 日本Sanyan公司;7500fast qPCR仪 美国ABI公司;多功能酶标仪美国伯腾仪器有限公司。
1.3.1 菌种活化
取-80 ℃保存的单增李斯特菌ATCC19115与副溶血性弧菌ATCC33847菌种,分别划线接种于PALCAM琼脂培养基和TCBS培养基中,37 ℃过夜培养24 h,选取特异性的单菌落,分别接种于9 mL TSB和9 mL TSB培养基(含质量分数3% NaCl溶液)中,于37 ℃、200 r/min摇床培养16 h后获得成熟的细菌培养液。
1.3.2 虾样品准备及DNA提取
南美白对虾样品于4 ℃下解冻,通过加热含质量分数2.5% NaCl的去离子水(100 ℃、20 min)进行背景微生物去除,置于生物安全柜中冷却至室温。取单增李斯特菌与副溶血性弧菌培养液于离心管中,3 000×g、25 ℃离心10 min,去除上清液,在磷酸盐缓冲液中进行重悬,得到最终菌量为108CFU/mL的菌悬液。将菌液以质量比1∶1进行混合,接种于南美白对虾之中,使得每只虾样品中致病菌的数量为105~106CFU/g。人工接种后的样品置于4 ℃及10 ℃恒温培养箱中培养。定时从培养箱中随机取出样品,于90 mL生理盐水中使用均质机拍打2 min,吸取2 mL均质液,200×g、4 ℃离心1 min,取上清液1 mL于1.5 mL离心管中,12 000 r/min、4 ℃离心2 min,弃去上清液,制得菌体,按照试剂盒说明书提取DNA,于-80 ℃保存备用。
1.3.3 qPCR测定
qPCR体系为20 µL:2×SYBR Green Master Mix 10 μL,引物F 1.5 µL(10 µmol/µL),引物R 1.5 µL(10 µmol/µL),DNA模板2 µL及ddH2O 5 µL。程序为:50 ℃、2 min;95 ℃、10 min;95 ℃、20 s,55 ℃、20 s,72 ℃、30 s,40 个循环。
1.3.4 标准曲线建立
取1.3.2节最终菌量为108CFU/mL的单增李斯特菌与副溶血性弧菌菌悬液进行梯度稀释,获得最终菌量为101~108CFU/mL的稀释液,按照1.3.2节和1.3.3节方法分别进行DNA抽提和qPCR,输出相应的循环阈值(Ct值)作为标准曲线Y轴。将不同菌量的稀释液涂布于PALCAM琼脂及TCBS培养基上,计数结果作为X轴,构建该反应的标准曲线,并计算曲线的斜率(K),利用公式(1)计算反应的扩增效率E。
1.3.5 模型的建立
运用Origin 8.0软件对实验结果进行处理,选用生长模型(修正Gompertz、Logistic、Baranyi)和失活模型(Log-linear、Weibull)分别拟合两株菌的生长或失活趋势[19-22]。
1.3.5.1 修正Gompertz模型
修正Gompertz模型的数学表达式如式(2)、(3)所示。
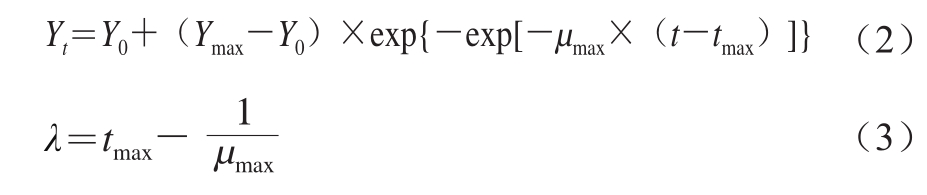
式中:Yt为t时的细胞数(lg(CFU/g));Y0与Ymax为拟合的初始数量和最大数量(lg(CFU/g));tmax为达到相对最大生长速率的时间/h;μmax为tmax时的最大比生长速率/((lg(CFU/g))/d);λ为延滞期/d。
1.3.5.2 Logistic模型
Logistic模型的数学表达式如式(4)所示。
1.3.5.3 Baranyi模型
Baranyi模型的数学表达式如式(5)~(7)所示。
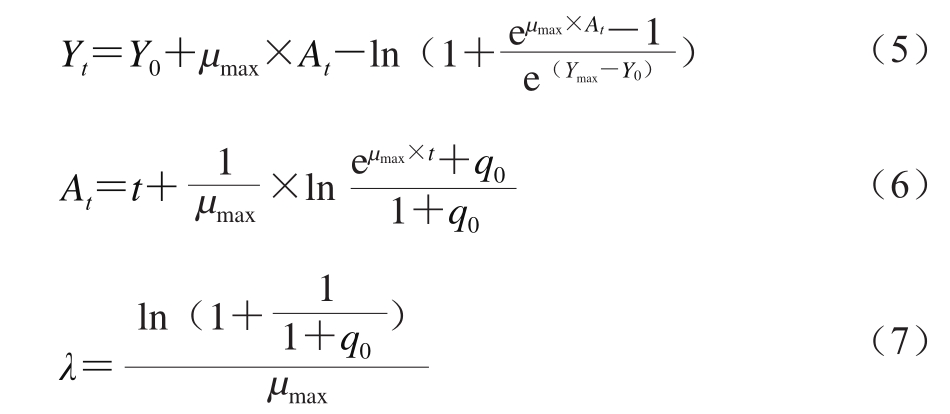
1.3.5.4 Log-linear模型
Log-linear模型的数学表达式如式(8)所示。
式中:K是最大比失活率/((lg(CFU/g))/d)。
1.3.5.5 Weibull模型
Weibull模型的数学表达式如式(9)所示。
式中:b是速率参数;n是拟合参数。
1.3.6 模型的评价
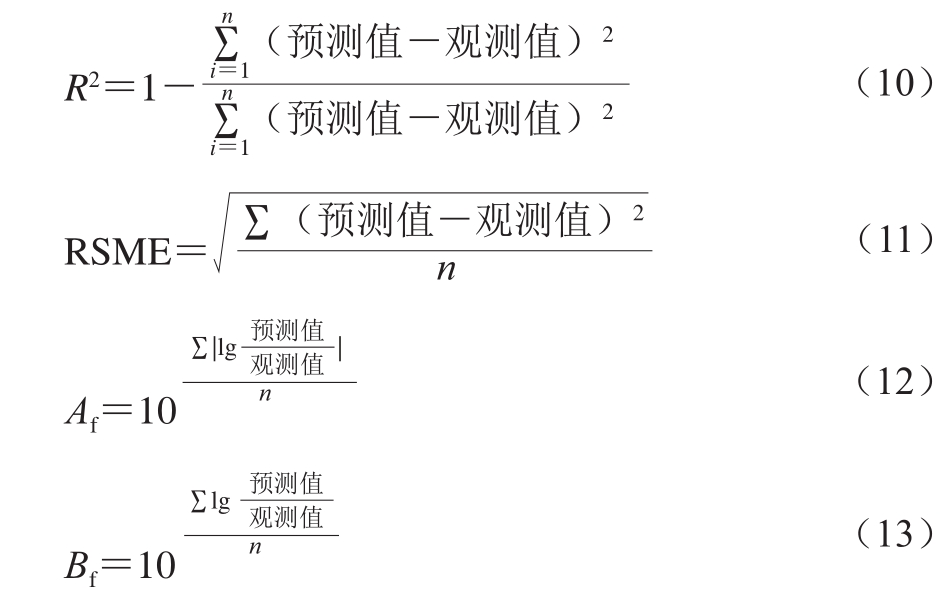
运用决定系数(R2)、均方根误差(the root mean square error,RMSE)、准确因子(accuracy factors,Af)和偏差因子(bias factors,Bf)评价模型的拟合效果,公式分别如式(10)~(13)所示[11]。
运用Origin 2017软件对两组数据进行拟合,建立标准曲线,采用生长模型(修正Gompertz、Logistic、Baranyi)和失活模型(Log-linear、Weibull)分别拟合两株菌的生长或失活趋势。
单增李斯特菌的标准曲线为:y=-3.27x+46.66(R2=0.974,E=102.2%)(图1A),副溶血性弧菌的标准曲线为:y=-3.49x+44.61(R2=0.980,E=93.4%)(图1B),扩增效率均在可接受的范围内(80%~120%)[23],所建立的标准曲线可适用于后续监测即食虾中单增生李斯特菌与副溶血性弧菌共存时的菌量变化。
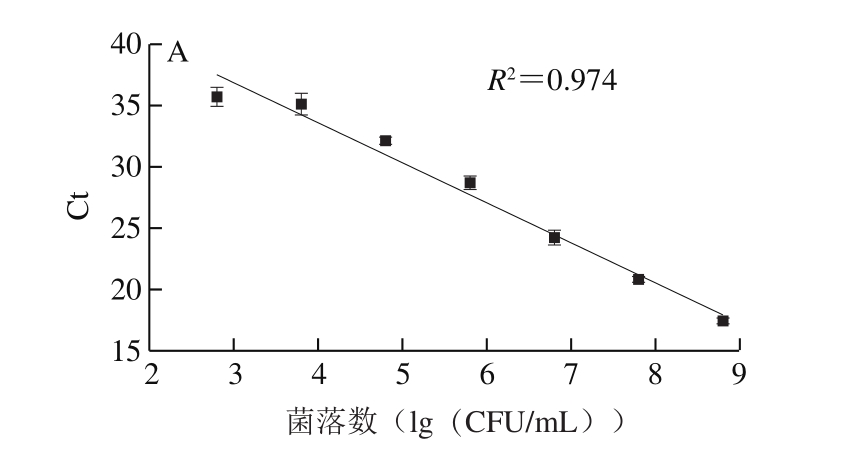
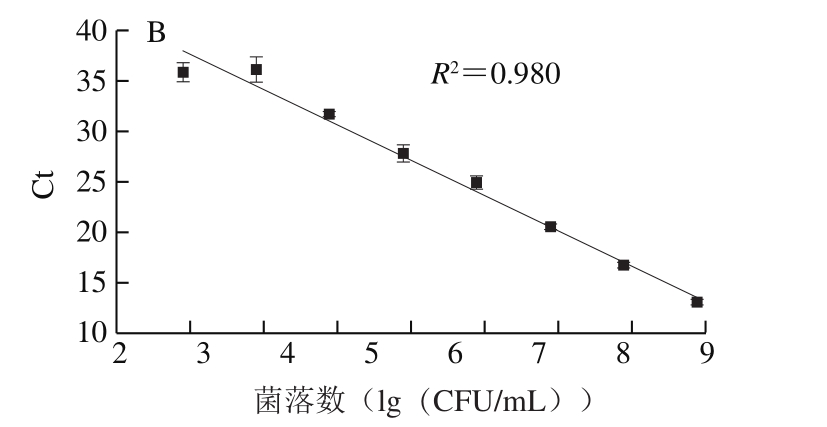
图1 单增李斯特菌(A)与副溶血性弧菌(B)标准曲线的构建
Fig.1 Standard curves for quantification of L. monocytogenes (A)and V. parahaemolyticus (B)
通过Origin 8.0软件对qPCR数据进行模型拟合,获得4 ℃和10 ℃条件下即食虾中单增李斯特菌的生长曲线(图2)和生长动力学参数(表1)。在4 ℃和10 ℃条件下,单增李斯特菌均呈S型生长趋势,对比于4 ℃条件,10 ℃时单增李斯特菌的延滞期更短,最大比生长速率更大。
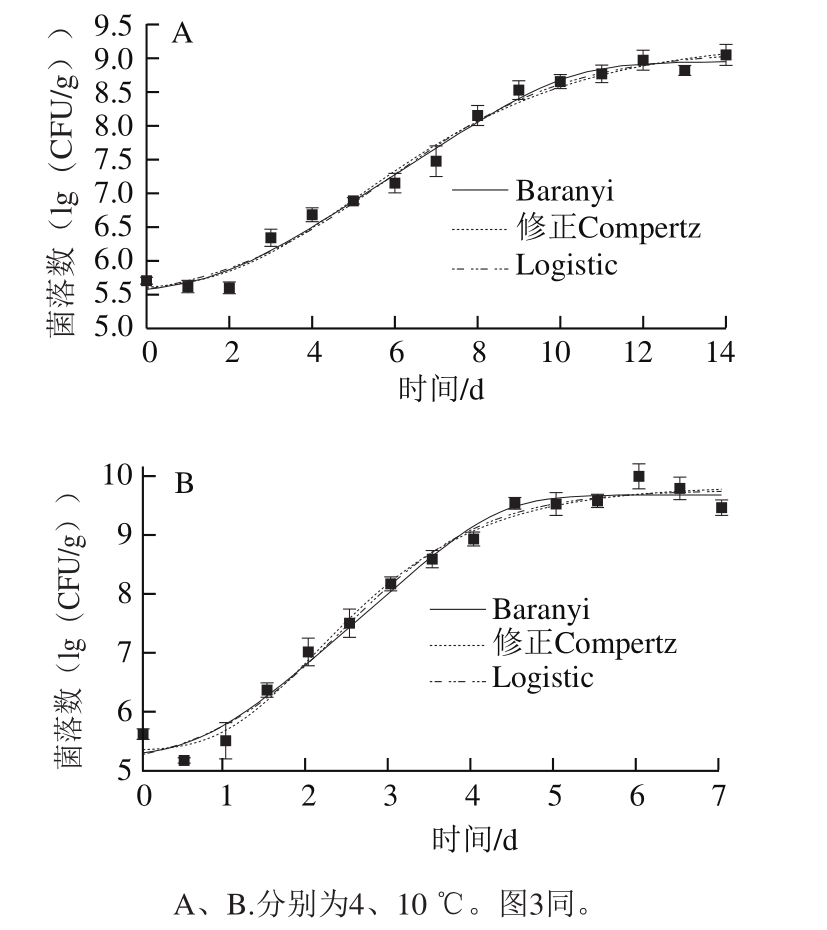
图2 即食虾中单增李斯特菌生长曲线拟合结果
Fig.2 Growth curves of L. monocytogenes on cooked shrimps
表1 不同生长动力学模型下即食虾中单增李斯特菌的生长参数
Table1 Growth kinetic parameters of L. monocytogenes on cooked shrimps

?
本研究运用R2、Af和Bf评价模型的拟合效果。R2用于描述预测值和观测值的相关程度[24],R2越趋近于1,表明预测模型参考价值越高;R2越趋近于0,则表明其参考价值越低[25]。Af能反映观测值与预测值间的接近程度:若Af=1,则表明所有观测值与预测值均相等,可视为完美模型[26];Af越大,则其平均精确度越低;Af一般用来验证预测模型的准确度,衡量模型预测的平均精确[27]。Bf一般作为判断预测模型偏差度的参数,表明了所建预测模型的结构性偏差,用于评价预测值偏离等值线的程度,且可判断预测值在等值线的上方还是下方。对于微生物代时(generation time,GT),如果Bf<1,说明该模型是失效保护模型,即GT平均预测值比观测值要小,这样预测模型可以给出一个安全的预测界限;Bf若在0.90~1.05之间,则说明拟合效果较好,使用该模型具有较小的偏差[28];Bf在0.70~0.90或1.06~1.15的范围内是可接受的;Bf<0.70或Bf>1.15则为不可接受,说明模型偏差较大、不可信[27]。
表2 修正Gompertz、Logistic、Baranyi、Log-linear和Weibull模型的R2、RMSE、Af和Bf
Table2 R2, RMSE, Afand Bfvalues of modified Gompertz, Logistic,Baranyi, Log-linear and Weibull models

?
由表2可知,Baranyi、修正Gompertz和Logistic模型R2均在0.98以上,各自的Af和Bf都非常接近1,表明这3 种模型都能较好地描述不同低温下即食虾中单增李斯特菌的生长状况,qPCR技术能够应用于混菌条件下单增李斯特菌生长趋势的描述。
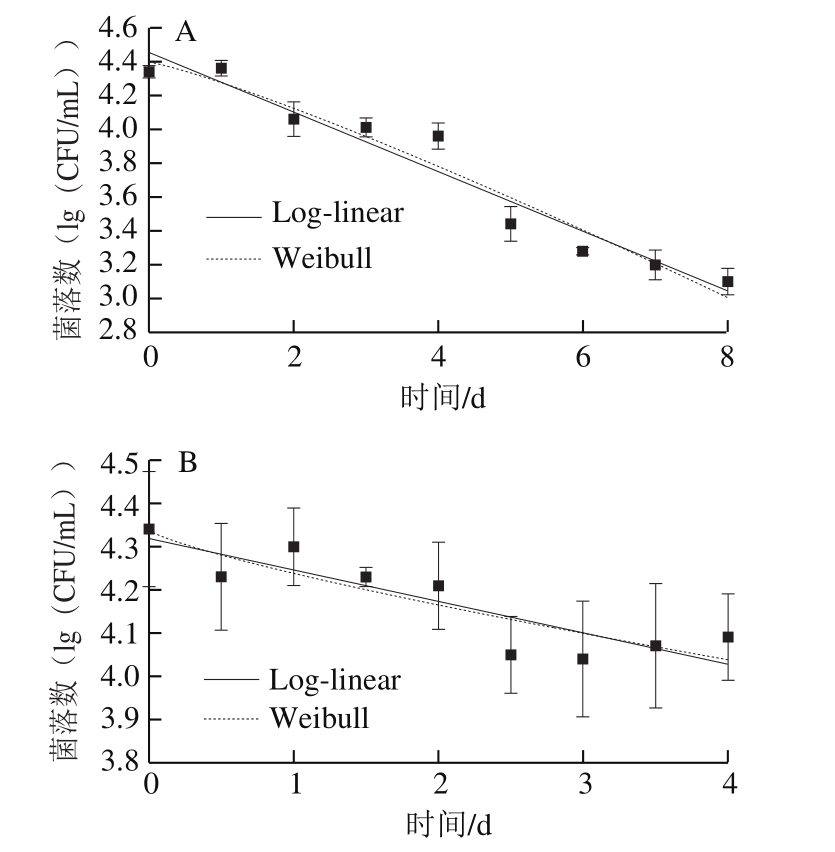
图3 即食虾中副溶血性弧菌失活曲线拟合结果
Fig.3 Inactivation curves of V. parahaemolyticus on cooked shrimps
表3 不同失活模型下即食虾中副溶血性弧菌失活参数
Table3 Inactivation kinetic parameters of V. parahaemolyticus on cooked shrimps

注:b(t)和n(t)是与温度相关的系数。
?
如图3、表3所示,4 ℃条件下Weibull模型(Yt=4.397-0.121×t1.176)与Log-linear模型(Yt=4.455-0.176×t)的R2分别是0.950和0.945(表2),各自的Af都接近1,Bf大于1且在可接受范围内(1.06~1.15),表明拟合效果较好。然而在10 ℃条件下,副溶血性弧菌的失活模型R2仅为0.784和0.775,这表明在10 ℃条件下,Weibull与Log-linear模型对副溶血性弧菌失拟,qPCR技术难以用于描述混菌条件下副溶血性弧菌的失活趋势。
本研究运用qPCR技术建立了低温下(4 ℃和10 ℃)单增李斯特菌与副溶血性弧菌共存于即食虾中的混合生长模型,对于单增李斯特菌,3 种模型的R2均在0.98以上,表明3 种模型均可以较好地描绘即食虾中单增李斯特菌的生长情况。qPCR具有特异性强、快速、高效等优势,且能够提供传统培养方法无法量化的信息,虽然不能完全替代传统方法,但弥补了传统预测微生物模型的不足[9,29-30]。
在利用qPCR所建立的副溶血性弧菌在4 ℃和10 ℃的失活模型中,Log-linear模型和Weibull模型的R2在4 ℃时分别为0.950和0.945,所得副溶血性弧菌的生长曲线呈急剧下降趋势,比单株副溶血性弧菌在4 ℃生长条件下失活更严重[5]。表明混菌状态下副溶血性弧菌的失活不仅与环境因子(温度)有关,还可能受到单增李斯特菌影响。而在10 ℃条件下,Log-linear模型和Weibull模型的R2分别为0.775和0.784,所得副溶血性弧菌生长曲线呈下降趋势,这与孙文烁[5]与黄和[31]等的报道稍有不同。在10 ℃条件下,孙文烁等的研究表明由平板涂布所得到的副溶血性弧菌的生长曲线呈现先下降后上升的趋势,且生长曲线不符合任何一级生长模型[5]。而黄和等也利用qPCR方法,得到的生长曲线则呈现缓慢上升的趋势,说明在10 ℃条件下qPCR很可能更适合揭示微生物的生长情况[5,31]。而本研究中模型失拟,且其生长呈失活趋势。混菌状态下建立的微生物预测模型更贴近真实情况,能更准确地反映微生物在真实环境中生长的实际规律。
本研究建立了基于混菌环境下的分子预测模型,描述低温下共存于即食虾中的副溶血性弧菌和单增李斯特菌的生长失活情况。为利用分子预测模型作为工具研究混菌环境下共存微生物的生长失活提供了依据,为微生物风险评估研究的逐步深入及完善提供理论技术支持,可以进一步揭示实际样品中微生物的生长状况,为微生物风险评估过程中可能忽视的潜在风险提供更加全面的信息。
[1] HANNING I B, JOHNSON M G, RICKE S C. Precut prepackaged lettuce: a risk for Listeriosis?[J]. Foodborne Pathogens and Disease,2008, 5(6): 731-746. DOI:10.1089/fpd.2008.0142.
[2] 董庆利, 郑丽敏, 党维鑫, 等. 即食食品中单增李斯特菌的半定量风险评估[J]. 食品工业科技, 2012, 33(11): 321-323; 366. DOI:10.13386/j.issn1002-0306.2012.11.087.
[3] 刘弘, 罗宝章, 秦璐昕, 等. 生食三文鱼片副溶血性弧菌污染的定量风险评估研究[J]. 中国食品卫生杂志, 2012, 24(1): 18-22.DOI:10.13590/j.cjfh.2012.01.017.
[4] PU Y H, SUN L J, WANG Y L, et al. Modeling inhibitory activity of a novel antimicrobial peptide AMPNT-6 from Bacillus subtilis against Vibrio parahaemolyticus in shrimp under various environmental conditions[J]. Food Control, 2013, 33(1): 249-253. DOI:10.1016/j.foodcont.2013.03.010.
[5] 孙文烁, 靳梦曈, 王敬敬, 等. 运用Real-time PCR建立即食虾中副溶血性弧菌分子预测模型[J]. 现代食品科技, 2014, 30(7): 142-148.DOI:10.13982/j.mfst.1673-9078.2014.07.033.
[6] ZAREI M, MAKTABI S, GHORBANPOUR M. Prevalence of Listeria monocytogenes, Vibrio parahaemolyticus, Staphylococcus aureus, and Salmonella spp. in seafood products using multiplex polymerase chain reaction[J]. Foodborne Pathogens and Disease, 2012, 9(2): 108-112.DOI:10.1089/fpd.2011.0989.
[7] FALLAH A A, SAEI-DEHKORDI S S, MAHZOUNIEH M.Occurrence and antibiotic resistance profiles of Listeria monocytogenes isolated from seafood products and market and processing environments in Iran[J]. Food Control, 2013, 34(2): 630-636. DOI:10.1016/j.foodcont.2013.06.015.
[8] WU S, WU Q P, ZHANG J M, et al. Listeria monocytogenes prevalence and characteristics in retail raw foods in China[J]. PLoS ONE, 2015, 10(8): e0136682. DOI:10.1371/journal.pone.0136682.
[9] 张昭寰, 娄阳, 杜苏萍, 等. 分子生物学技术在预测微生物学中的应用与展望[J]. 食品科学, 2017, 38(9): 248-257. DOI:10.7506/spkx1002-6630-201709040.
[10] YOON K S, MIN K J, JUNG Y J, et al. A model of the effect of temperature on the growth of pathogenic and nonpathogenic Vibrio parahaemolyticus isolated from oysters in Korea[J]. Food Microbiology, 2008, 25(5): 635-641. DOI:10.1016/j.fm.2008.04.007.
[11] YANG Z Q, JIAO X A, LI P, et al. Predictive model of Vibrio parahaemolyticus growth and survival on salmon meat as a function of temperature[J]. Food Microbiology, 2009, 26(6): 606-614.DOI:10.1016/j.fm.2009.04.004.
[12] PARVEEN S, DASILVA L, DEPAOLA A, et al. Development and validation of a predictive model for the growth of Vibrio parahaemolyticus in post-harvest shellstock oysters[J]. International Journal of Food Microbiology, 2013, 161(1): 1-6. DOI:10.1016/j.ijfoodmicro.2012.11.010.
[13] 彭织云, 王敬敬, 唐晓阳, 等. 运用Real-time quantification PCR方法建立副溶血性弧菌在即食虾中的生长预测模型[J]. 食品工业科技,2013, 34(8): 108-110. DOI:10.13386/j.issn1002-0306.2013.08.042.
[14] 赵新, 王永, 刘娜, 等. 金针菇中单增李斯特菌生长预测模型的建立[J]. 生物加工过程, 2016, 14(6): 12-17. DOI:10.3969/j.issn.1672-3678.2016.06.003.
[15] METSELAAR K I, ABEE T, ZWIETERING M H, et al. Modeling and validation of the ecological behavior of wild-type Listeria monocytogenes and stress-resistant variants[J]. Applied and Environmental Microbiology, 2016, 82(17): 5389-5401. DOI:10.1128/AEM.00442-16.
[16] SZCZAWIŃSKI J, SZCZAWIŃSKA M E, ŁOBACZ A, et al.Modeling the effect of temperature on survival rate of Listeria monocytogenes in yogurt[J]. Polish Journal of Veterinary Sciences,2016, 19(2): 317-324. DOI:10.1515/pjvs-2016-0039.
[17] OMICCIOLI E, AMAGLIANI G, BRANDI G, et al. A new platform for real-time PCR detection of Salmonella spp., Listeria monocytogenes and Escherichia coli O157 in milk[J]. Food Microbiology, 2009, 26(6): 615-622. DOI:10.1016/j.fm.2009.04.008.
[18] NORDSTROM J L, VICKERY M C L, BLACKSTONE G M, et al.Development of a multiplex real-time PCR assay with an internal amplification control for the detection of total and pathogenic Vibrio parahaemolyticus bacteria in oysters[J]. Applied and Environmental Microbiology, 2007, 73(18): 5840-5847. DOI:10.1128/AEM.00460-07.
[19] ZWIETERING M H, JONGENBURGER I, ROMBOUTS F M, et al.Modeling of the bacterial growth curve[J]. Applied and Environmental Microbiology, 1990, 56(6): 1875-1881.
[20] BARANYI J, ROBERTS T A. A dynamic approach to predicting bacterial growth in food[J]. International Journal of Food Microbiology, 1994, 23(3/4): 277-294. DOI:10.1016/0168-1605(94)90157-0.
[21] XIONG R, XIE G, EDMONDSON A E, et al. A mathematical model for bacterial inactivation[J]. International Journal of Food Microbiology, 1999, 46(1): 45-55. DOI:10.1016/S0168-1605(98)00172-X.
[22] PELEG M, COLE M B. Reinterpretation of microbial survival curves[J]. Critical Reviews in Food Science and Nutrition, 1998,38(5): 353-380. DOI:10.1080/10408699891274246.
[23] WANG J J, SUN W S, JIN M T, et al. Fate of Vibrio parahaemolyticus on shrimp after acidic electrolyzed water treatment[J]. International Journal of Food Microbiology, 2014, 179: 50-56. DOI:10.1016/j.ijfoodmicro.2014.03.016.
[24] DING T, RAHMAN S M E, PUREV U, et al. Modelling of Escherichia coli O157:H7 growth at various storage temperatures on beef treated with electrolyzed oxidizing water[J]. Journal of Food Engineering,2010, 97(4): 497-503. DOI:10.1016/j.jfoodeng.2009.11.007
[25] STEELE R. Understanding and measuring the shelf-life of food[M].Cambridge: Woodhead Publishing, 2004: 189-206.
[26] LEBERT I, ROBLES-OLVERA V, LEBERT A. Application of polynomial models to predict growth of mixed cultures of Pseudomonas spp. and Listeria in meat[J]. International Journal of Food Microbiology,2000, 61(1): 27-39. DOI:10.1016/S0168-1605(00)00359-7.
[27] ROSS T. Indices for performance evaluation of predictive models in food microbiology[J]. Journal of Applied Microbiology, 1996, 81(5):501-508. DOI:10.1111/j.1365-2672.1996.tb03539.x.
[28] ROSS T, DALGAARD P, TIENUNGOON S. Predictive modelling of the growth and survival of Listeria in fishery products[J]. International Journal of Food Microbiology, 2000, 62(3): 231-245. DOI:10.1016/S0168-1605(00)00340-8.
[29] ZHANG Z H, LIU H Q, LOU Y, et al. Quantifying viable Vibrio parahaemolyticus and Listeria monocytogenes simultaneously in raw shrimp[J]. Applied Microbiology & Biotechnology, 2015, 99(15):6451-6462. DOI:10.1007/s00253-015-6715-x.
[30] ZHANG Z H, XIAO L L, LOU Y, et al. Development of a multiplex real-time PCR method for simultaneous detection of Vibrio parahaemolyticus, Listeria monocytogenes, and Salmonella spp.in raw shrimp[J]. Food Control, 2015, 51: 31-36. DOI:10.1016/j.foodcont.2014.11.007.
[31] 黄和, 蒋志红, 雷晓凌, 等. 冻熟虾中副溶血性弧菌生长模型[J]. 广东海洋大学学报, 2009, 29(1): 94-98. DOI:10.3969/j.issn.1673-9159.2009.01.020.
Development of Predictive Models Based on qPCR for the Growth ofListeria monocytogenesand Vibrio parahaemolyticus Coexisting on Cooked Shrimps Stored at Low Temperatures