表1 空肠弯曲菌分离菌株信息
Table 1 Information about C. jejuni strains used in this study

菌株编号 食品来源 种类 采样地点Cj 1~19 养殖场 鸭源 江苏南京Cj 20~65 养殖场 鸡源 江苏南京Cj 66~80 活禽市场 鸡源 江苏苏州Cj 81~86 零售 鸡源 江苏苏州
自然界中存在着大量的可移动遗传元件,如转座子、质粒等,这些外源核酸片段在原核生物中的水平转移推动了原核生物进化的多样性,同时也促使细菌、古菌等进化出一套抵御基因水平转移的机制[1]。规律成簇间隔短回文重复序列(clustered regularly interspaced short palindromic repeats,CRISPR)及其相关蛋白(Cas蛋白等)就是在细菌和古菌中新发现的针对噬菌体、质粒等外源核酸片段入侵的原核生物免疫系统。目前研究表明,在已测序的基因组中,大约有40%的细菌和90%的古菌基因组中至少存在1个CRISPR座位(locus)[2]。完整的CRISPR-cas系统通常包括一个前导序列(leader)、重复间隔序列以及CRISPR相关蛋白编码基因(CRISPR associated genes,cas)。重复间隔序列是CRISPR结构最明显的特征,由一段不连续的同向DNA重复序列(通常为24~47 bp)以及插入其中的间隔序列(通常为21~72 bp)组成。重复序列具有高度的保守性,多具有回文结构,体现细菌在进化中的保守性;间隔序列大多来源于外源可移动遗传元件,如噬菌体、质粒等,高度可变反映出多样性[3]。有研究发现,重复间隔序列、前导序列和cas基因之间存在共进化,构成一套保守而完整的系统,新间隔序列的插入往往伴随着原有间隔序列的删除,同时,CRISPR的进化与其所处的环境也有关[4]。
空肠弯曲菌(Campylobacter jejuni)是一种重要的食源性人畜共患病病原菌。空肠弯曲菌感染通常导致空肠弯曲菌肠炎,主要症状为细菌性腹泻。目前在很多国家,它是引起人细菌性腹泻的最重要原因之一。欧洲食品安全局报告指出,在欧盟该菌已连续多年成为导致食源性疾病病例最多的细菌[5]。根据美国疾控中心2016年度对美国食源性致病微生物引起发病人数的统计资料,空肠弯曲菌导致的病例数排名第一[6]。我国由于缺乏较为完整和系统的空肠弯曲菌流行病学数据,目前尚无法评估其确切的危害性。空肠弯曲菌广泛存在于家禽、家畜以及鸟类的肠道内,在很多动物中属于正常携带细菌,能够通过食物链传递给人类,引起人类发病。世界卫生组织已经将空肠弯曲菌列为最常见的食源性病原菌之一。空肠弯曲菌污染的肉、奶、蛋以及水源等是目前已报道的空肠弯曲菌感染的主要来源。采用有效的分子分型方法对空肠弯曲菌进行分型,对于掌握空肠弯曲菌的污染分布规律,建立完善的溯源图谱系统非常重要。本研究通过对在禽源中分离的86 株空肠弯曲菌进行CRISPR结构分析,统计重复序列和间隔序列数量信息,分析间隔序列与外源基因的同源性,以期能更好地理解空肠弯曲菌CRISPR结构特征与分布情况,为今后食源性空肠弯曲菌的溯源和控制提供帮助。
空肠弯曲菌标准菌株(NCTC11168和ATCC49943)由江苏出入境检验检疫局馈赠。实验用86株空肠弯曲菌菌株从江苏地区禽肉中分离,详细信息见表1。分离及鉴定方法参考GB 4789.9—2014《食品微生物学检验 空肠弯曲菌检验》[7]。鉴定过的菌株使用Wang’s保存液[8]保存于-70 ℃低温冰箱中。
表1 空肠弯曲菌分离菌株信息
Table 1 Information about C. jejuni strains used in this study

菌株编号 食品来源 种类 采样地点Cj 1~19 养殖场 鸭源 江苏南京Cj 20~65 养殖场 鸡源 江苏南京Cj 66~80 活禽市场 鸡源 江苏苏州Cj 81~86 零售 鸡源 江苏苏州
哥伦比亚血平板 英国Oxoid公司;绵羊全血 北京陆桥技术股份有限公司;微需氧产气袋 法国梅里埃公司;细菌基因组提取试剂盒 天根生化科技有限公司;DNA Marker DL-2000、DreamTaq Green聚合酶链式反应(polymerase chain reaction,PCR)Master Mix 美国Thermo公司。
SD115恒温培养箱 德国Binder公司;2X4ATT梯度PCR仪 德国Biometra公司;Gel Doc XR+凝胶成像系统 美国伯乐公司。
1.3.1 提取细菌基因组DNA
配制哥伦比亚血平板,灭菌冷却至50 ℃后添加5%的绵羊血混匀倒平板。将冷冻保藏的菌株接种到哥伦比亚血平板上,42 ℃微需氧环境下(5% O2,10% CO2,85% N2)进行培养,48 h后挑取单菌落划线在哥伦比亚血平板上并培养。将培养好的细菌菌落用无菌生理盐水洗出收集,使用试剂盒方法提取基因组DNA。提取到的DNA样品分装后,-20 ℃冰箱保存待用。
1.3.2 CRISPR引物设计
根据Price等[9]对210 株澳大利亚空肠弯曲菌和结肠弯曲菌CRISPR结构的研究报道,同时比对数据库CRISPR database(http://crispr.i2bc.paris-saclay.fr/)公布的空肠弯曲菌CRISPR位点信息[10],确定空肠弯曲菌CRISPR位点引物为:正向引物CRISPR-For(5’-GCAACCTCCTT TTAGTGGAGTAATTAG-3’),反向引物CRISPR-Rev(5’-AAGCGGTTTTAGGGGATTGTAAC-3’)。引物由上海生工生物工程技术公司有限公司合成。
1.3.3 CRISPR序列PCR扩增和测序
PCR条件:95 ℃预变性2 min,94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸2 min,进行30 个循环,72 ℃延伸5 min。反应结束后,取3 μL产物进行1.0%琼脂糖凝胶电泳鉴定。所得阳性结果送至上海生工生物工程股份有限公司进行双向测序。
1.3.4 CRISPR序列分析及同源性比对1.3.4.1 CRISPR序列识别
测序结果采用SeqMan软件进行拼接,拼接后的序列使用CRISPRs finder(http://crispr.i2bc.paris-saclay.fr/Server/)进行分析[11],结果可以得到确认的和存疑的两种CRISPR结构,对于存疑的CRISPR结构,需进行人工比对。
1.3.4.2 重复序列二级结构预测
对完整CRISPR结构(至少包括2 个重复序列和1个间隔序列)的所有重复序列分别进行多序列比对,利用Weblogo(3.0版本,http://weblogo.berkeley.edu/logo.cgi)进行序列保守性分析[12]。依据碱基频率,使用RNAfold web server(http://rna.tbi.univie.ac.at/cgi-bin/RNAWebSuite/RNAfold.cgi)分别预测其RNA二级结构。
1.3.4.3 间隔序列的同源性分析
通过CRISPR finder网站、CRISPR recognition tool(CRT)软件和Mega 5.0多序列比对获得每个CRISPR结构重复序列和间隔序列的信息[11,13]。将序列差异低于10%的两个间隔序列,认为是相同序列[14]。对间隔序列进行多序列比对,去除重复的间隔序列。使用CRISPR Target(http://bioanalysis.otago.ac.nz/CRISPRTarget/crispr_analysis.html)对间隔序列的同源性进行分析,选择的数据库包含GenBank-Phage、RefSeq-Plasmid、RefSeq-Microbial和RefSeq-viral等,比对相关参数选择默认值[15],获得间隔序列与外源基因的同源性信息。
以88 株空肠弯曲菌基因组DNA为模板,PCR扩增CRISPR片段,测序结果输入CRISPRs finder分析查找CRISPR结构。结果显示,经过PCR扩增共有51 株细菌的基因组DNA扩增出片段产物。通过分析发现在51 个PCR产物中,有13 株对应的菌株未检出任何确认的CRISPR结构,有32 个有确定的CRISPR结构,6 个有疑似的CRISPR结构,CRISPR检出率为36%。
截止到2017年9月,CRISPRs database数据库(http://crispr.i2bc.paris-saclay.fr/crispr/)共收录111 株空肠弯曲菌全基因组序列,经数据库分析其中有50 株菌株含有确定的CRISPR结构,占总菌株数的45%[10]。其中2 株菌(C. jejuni OD267 GCF_001587015)被确定有2 个CRISPR结构(1个位于质粒上),菌株(C. jejuni subsp. jejuni M129 GCF_001865595)的CRISPR结构只在质粒上有1 个,其余菌株全基因组均只有1 个CRISPR结构且均位于染色体DNA上。在发现的CRISPR结构中单菌株CRISPR的长度为101~959 bp,最小间隔序列数量为1,最大间隔序列为14(表2)。
表2 空肠弯曲菌CRISPR结构统计信息
Table 2 Information about CRISPRs of C. jejuni obtained in this study and from the database
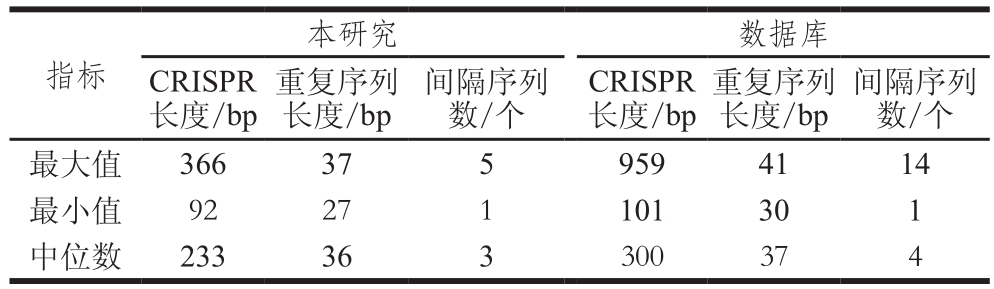
间隔序列数/个最大值 366 37 5 959 41 14最小值 92 27 1 101 30 1中位数 233 36 3 300 37 4指标本研究 数据库CRISPR长度/bp重复序列长度/bp间隔序列数/个CRISPR长度/bp重复序列长度/bp
本研究检测空肠弯曲菌的CRISPR结构比率为36%,较数据库略低。32 株菌株中每个菌株均只有1个CRISPR结构,其中CRISPR序列长度范围在92~366 bp,重复序列长度范围为27~37 bp,与数据库数据接近(30~41 bp)。间隔序列数量为1~5 个,与参考数据库相比波动小很多(表2)。
在所发现的32 个CRISPR结构中共有4 种序列有差异的重复序列,但这4 种序列之间的差异仅仅是部分碱基突变导致的个别碱基差异,且差异都小于10%,因此将其视为相同序列。识别到的重复序列为5’-GTTTTAGTCC CTTTTTAAATTTCTTTATGGTAAAAT-3’。这条序列与空肠弯曲菌CRISPR数据库中出现频率最高的重复序列一致,因此本研究并未发现新的重复序列。经weblogo在线分析所有重复序列(共145 条)的保守型(图1)发现,在重复序列的起始端位点存在较多的变异,该重复序列在空肠弯曲菌的CRISPR结构中普遍存在,进一步验证了重复序列具有高度的保守性[16]。有研究认为,重复序列可以转录且形成稳定的RNA二级结构,这个现象在古菌中已被验证[17-18]。使用RNAfold web server对本研究发现的重复序列进行二级结构预测,结果发现,该重复序列并不能形成较为稳定的茎环结构。通常认为,大部分细菌的CRISPR结构的重复序列能形成较为稳定的二级茎环结构,如嗜热链球菌、志贺菌、嗜水气单胞菌等[19-21],但是,在近几年的很多研究中也发现并不是所有的重复序列都能够形成稳定的二级结构[17,22-23],有研究[17,24]对195 个微生物基因组中的CRISPR重复序列进行结构分析,根据重复序列能否形成稳定的二级结构可以将CRISPR分为12 个大类,在细菌和古菌基因组中都存在不能形成稳定二级结构的类群,他们发挥作用可能是依赖其序列的特异性和特异性蛋白相互作用来实现的,而该特性已在古菌Sulfolobus的CRISPR结构中得到验证[23]。
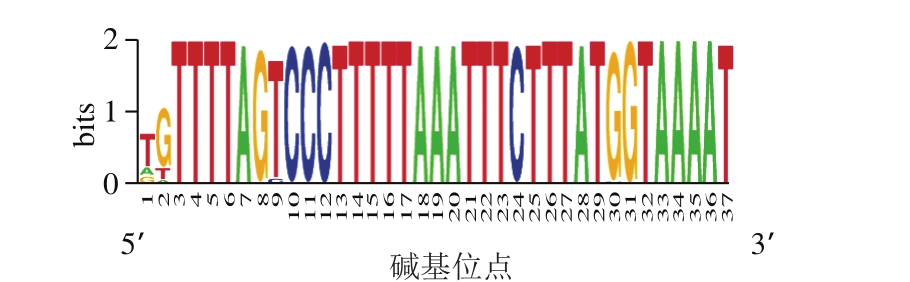
图1 重复序列的碱基频率分析图
Fig. 1 Base frequency of the direct repeats
CRISPR Finder识别到的间隔序列共有111 条,序列相似性分析显示共包括17 种不同类型的间隔序列,有10 种类型的间隔序列与CRISPR数据库中公布的一致;有7 种间隔序列为新发现的间隔序列,共55 条,占总间隔序列的49.5%,目前空肠弯曲菌CRISPR数据库中信息还是比较有限,本研究将为空肠弯曲菌CRISPR结构的进化和功能机制研究提供更丰富的信息。
之前的研究已经揭示了细菌CRISPR结构的间隔序列与噬菌体、质粒等外源基因组,以及同一细菌基因组的其他部分具有高度同源性[25-26],因此本研究将在空肠弯曲菌中发现的111 条间隔序列与GenBank-Phage、RefSeq-Plasmid、RefSeq-Microbial和RefSeq-viral 4 个数据库中的序列进行了BLAST比对分析。表3结果显示,有57 个间隔序列与质粒或噬菌体序列具有同源性片段,其中有9 个间隔序列与弯曲菌的噬菌体具有高度同源性,23 个间隔序列与弯曲菌携带的质粒高度同源。这一现象充分证实了细菌通过CRISPR获得免疫能力的机制:弯曲菌在抵御外源质粒或噬菌体时,将该相应的外源片段整合到自身的CRISPR结构,形成自身免疫机制。另外,经比对发现有24 个间隔序列与食源性致病菌产气荚膜梭菌携带质粒存在同源性,产气荚膜梭菌也是一类较为常见的食源性致病菌[27],且常存在于禽类动物的肠道中,与空肠弯曲菌的生存环境相似[28],这一现象表明,动物肠道环境为致病菌之间的信息交流提供了场所和条件,外源质粒在这一环境中存在基因的水平转移。另外,还发现一个间隔序列比对结果与弧菌噬菌体具有同源性,弧菌科与弯曲菌科亲缘关系较近,可能存在弧菌噬菌体同样能够侵染弯曲菌的现象,这种基因水平转移现象在许多细菌CRISPR结构中都被发现[29-30],这也是CRISPR系统限制细菌间的水平基因转移,帮助细菌防御噬菌体侵染的有利途径。
表3 间隔序列同源比对结果
Table 3 Homologous spacers from plasmids or phages

注:*. CRISPR Target比对结果输出的置信分数,计算法则为间隔序列逐个碱基与库中比对,比对匹配+1,比对不匹配-1,算出最终得分(分数30即代表大于99%同源性)。
间隔序列 5’-3’序列 数目 比对结果 比对分数* 序列编码产物Sp8 AAAGAACTTTCAATAACTGAGGCTTATATG 23 C. jejuni strain OD267 plasmid pCJDM67 30 假定蛋白Sp10 CCTTCAAGTCGTGCAATTTTAATACACTG 7 Campylobacter phage CJIE4-1 30 假定蛋白Sp16 AATACTTTAAGAAGTATTACAAAAAAAGAT 1 Campylobacter phage CJIE4-5 30 假定蛋白Sp3 CTACAAGAATGAGGATGATGATATTTTACA 1 Campylobacter phage PC5 26 假定蛋白Sp9 GATTGGAATTTAGATGAGATTAGAGAAATA 23 Clostridium perfringens plasmid pCP8533etx 20 DNA腺嘌呤甲基化酶Sp7 ATTTGCAAGGTATATAAAAAATTTGATTTT 1 Clostridium baratii strain CDC51267 plasmid pNPD11_1 20 假定蛋白Sp2 TTTCCAAAGTTTCATTAGTTGAATTTAACT 1 Vibrio phage N4 20 RNA聚合酶
已有研究认为,CRISPR结构的差异可作为细菌分型的一项指标[3,31]。88 株空肠弯曲菌中,包含确定的CRISPR结构的菌株32 株,疑似CRISPR结构的菌株6 株。由于疑似CRISPR结构在一定程度上代表了在该CRISPR座位上的结构特征,有可能会为菌株的分型提供更多的信息,因此将疑似CRISPR结构与确定的CRISPR结构合并进行分析。根据CRISPR位点间隔序列的多样性,共组成了7 种不同的CRISPR谱型(图2)。通过CRISPR位点分布图发现,所测菌株大部分分布在C谱型,与标准菌株NCTC11168和ATCC49943相比较有较大差异。NCTC11168分离自人体腹泻样本[32],本次检测到的CRISPR结构与以往报道一致,该菌株含有一个由5 个重复序列和4 个间隔序列构成的CRISPR[33],可能由于其分离环境与本研究菌株不同,菌株获得的外源序列有所不同;标准菌株ATCC49943也检测到由5 个重复序列和4 个间隔序列构成的CRISPR,然而其间隔序列与NCTC11168所含有的间隔序列完全不同,值得指出的是,与本实验室菌株的主要谱型(C谱型)相比,菌株ATCC49943的第1个间隔序列和最后1个间隔序列与C谱型完全相同,而中间第2、3个间隔序列与C谱型不同,这可能暗示着空肠弯曲菌CRISPR结构在不断进化的过程中伴随着间隔序列的删除和插入[16]。同时,本研究发现在所有空肠弯曲菌CRISPR结构中,间隔序列Sp8在B、C、E 3个谱型中都出现,且均在空肠弯曲菌CRISPR结构的末端,表明该间隔序列可能在空肠弯曲菌中存在一定的普遍性和保守性。
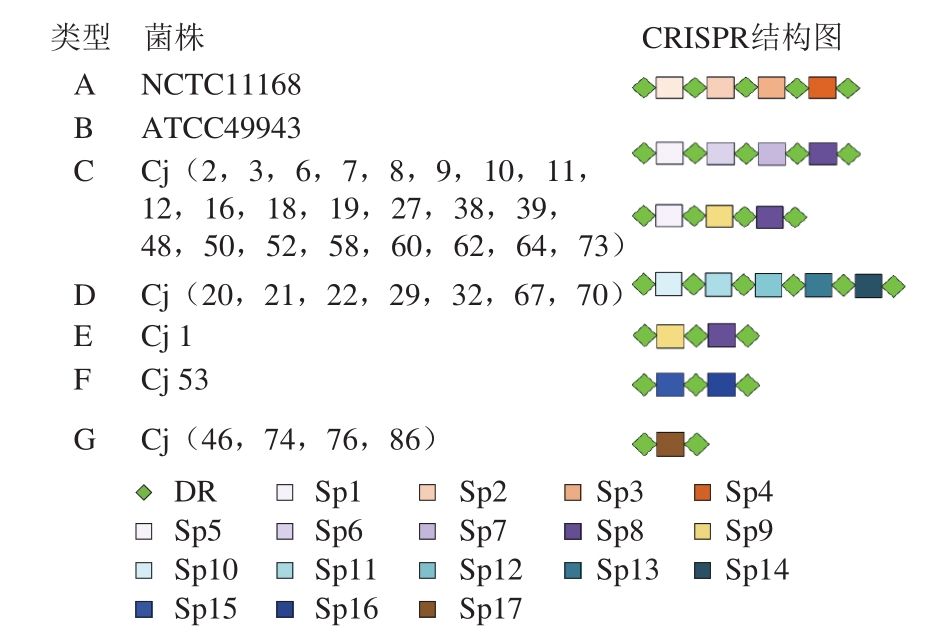
图2 38 株空肠弯曲菌CRISPR结构分布图
Fig. 2 CRISPR structure distribution of 38 strains of C. jejuni
86 株野生分离株中,其中分离自江苏南京养殖场的鸭源空肠弯曲菌(Cj 1~19)CRISPR结构分布比率最高(68.4%),而且主要都在C谱型中,仅Cj 1属于E谱型,且仅含有2个间隔序列(图2)。分离自江苏南京养殖场的鸡源空肠弯曲菌(Cj 20~65)中共检测到17 个CRISPR结构,主要分布在C、D、F和G谱型中,其中以C谱型和D谱型结构较多(图2)。从江苏苏州活禽市场和零售鸡肉中分离到的空肠弯曲菌所检测到的CRISPR结构比率相对较低,从活禽市场样品中检测到的CRISPR结构主要为D谱型和G谱型。而零售鸡肉中分离到的6 株空肠弯曲菌菌株仅1 株检测到CRISPR结构,结构为G谱型(图2)。以上结果表明,不同样品来源分离得到的空肠弯曲菌在CRISPR结构比率和谱型上差距较大,间隔序列排列方式和序列差异性也较为显著,表明CRISPR结构在细菌分型和溯源上的巨大潜力。目前CRISPR已经被成功应用于沙门菌[34]、大肠杆菌[35-36]、布氏乳杆菌[37]等的分型中,但对空肠弯曲菌的CRISPR信息了解甚少[38-39]。本研究为空肠弯曲菌的CRISPR结构提供了序列信息,对其CRISPR谱型的研究为后续分型和溯源研究奠定了数据基础,然而,本研究发现,CRISPR结构在空肠弯曲菌中的存在率较低(36%),根据报道,其在细菌中的存在率为40%左右[2],其较低的存在率极大地限制了CRISPR分型的普适性。与传统的分子分型方法,如:脉冲场凝胶电泳(pulsed field gel electrophoresis,PFGE),多位点序列分型(multilocus sequence typing,MLST)等相比,基于细菌全基因组的PFGE分型方法和基于细菌管家基因序列差异性的MLST分型方法则不存在该限制。但是,CRISPR结构检测、数据处理和分析程序方面相较于PFGE、MLST等方法简化了很多,且由于CRISPR本身的特点,间接序列携带的外源基因片段反映了其遗传进化过程中的轨迹,更能区分同一物种下的细微差异。因此,对含有CRISPR结构的菌株进行进一步分型可以更好地完善和补充现有分型方法的不足。
本研究对江苏地区分离的86株空肠弯曲菌野毒株进行CRISPR结构的检测和生物信息学分析,通过携带CRISPR结构的特征,对空肠弯曲菌进行了CRISPR分型的研究探索。88 株空肠弯曲菌(含标准菌株2 株)中,36%的菌株中含有确定的CRISPR结构(43%含疑似CRISPR结构),且菌株均只含有1个CRISPR结构。检测到的111 条间隔序列根据其序列的差异性可分为17 种类型,其中7 种为首次发现的间隔序列。间隔序列同源性分析发现,菌株中的间隔序列多来源于弯曲菌、产气荚膜梭菌和弧菌携带的质粒,以及噬菌体。根据CRISPR结构差异,本研究将检出CRISPR结构的38 株菌株分为7 个CRISPR谱型,不同样品来源分离得到的空肠弯曲菌在CRISPR结构比率和谱型上差距较大,间隔序列排列方式在不同谱型之间存在一定的保守性,其序列差异性也表明了CRISPR结构在不断进化过程中存在序列的删除和插入。本研究表明CRISPR结构在空肠弯曲菌分型和溯源上的潜力,但并不是所有空肠弯曲菌中都存在CRISPR结构,采用CRISPR结构分析与其他分子分型(如MLST、PFGE等)相结合的方法进行分子分型可能是未来空肠弯曲菌分子分型的一个发展方向。
[1] KOONIN E V, WOLF Y I. Genomics of bacteria and archaea: the emerging dynamic view of the prokaryotic world[J]. Nucleic Acids Research, 2008, 36(21): 6688-6719. DOI:10.1093/nar/gkn668.
[2] MARRAFFINI L A, SONTHEIMER E J. CRISPR interference: RNA-directed adaptive immunity in bacteria and archaea[J]. Nat Rev Genet,2010, 11(3): 181-190. DOI:10.1038/nrg2749.
[3] SOREK R, KUNIN V, HUGENHOLTZ P. CRISPR: a widespread system that provides acquired resistance against phages in bacteria and archaea[J]. Nature Reviews Microbiology, 2008, 6(3): 181-186.DOI:10.1038/nrmicro1793.
[4] HORVATH P, ROMERO D A, COUTE-MONVOISIN A C, et al.Diversity, activity, and evolution of CRISPR loci in Streptococcus thermophilus[J]. Journal of Bacteriology, 2008, 190(4): 1401-1412.DOI:10.1128/JB.01415-07.
[5] EFSA. The european union summary report on trends and sources of zoonoses, zoonotic agents and food-borne outbreaks in 2014[J]. EFSA Journal, 2015, 13(12): 4329.
[6] Centers for Disease Control and Prevention. Campylobacter,Salmonella led bacterial foodborne illnesses in 2016[N]. CDC Newsroom Releases, 2017-04-20.
[7] 国家卫生与计划生育委员会. 食品微生物检验 空肠弯曲菌检验:GB 4789.9—2014[S]. 北京: 中国标准出版社, 2014.
[8] National Advisory Committee on Microbiological Criteria for Foods.Analytical utility of Campylobacter methodologies[J]. Journal of Food Protection, 2007, 70(1): 241-250.
[9] PRICE E P, SMITH H, HUYGENS F, et al. High-resolution DNA melt curve analysis of the clustered, regularly interspaced shortpalindromic-repeat locus of Campylobacter jejuni[J]. Applied and Environmental Microbiology, 2007, 73(10): 3431-3436. DOI:10.1128/AEM.02702-06.
[10] GRISSA I, VERGNAUD G, POURCEL C. The CRISPRdb database and tools to display CRISPRs and to generate dictionaries of spacers and repeats[J]. BMC Bioinformatics, 2007, 8: 172-181.DOI:10.1186/1471-2105-8-172.
[11] GRISSA I, VERGNAUD G, POURCEL C. CRISPR Finder: a web tool to identify clustered regularly interspaced short palindromic repeats[J].Nucleic Acids Research, 2007, 35: W52-W57. DOI:10.1093/nar/gkm360.
[12] CROOKS G E, HON G, CHANDONIA J M, et al. Weblogo: a sequence logo generator[J]. Genome Research, 2004, 14(6): 1188-1190. DOI:10.1101/gr.849004.
[13] BLAND C, RAMSEY T L, SABREE F, et al. CRISPR recognition tool (CRT): a tool for automatic detection of clustered regularly interspaced palindromic repeats[J]. BMC Bioinformatics, 2007, 8: 209.DOI:10.1186/1471-2105-8-209.
[14] TOUCHON M, CHARPENTIER S, CLERMONT O, et al. CRISPR distribution within the Escherichia coli species is not suggestive of immunity-associated diversifying selection[J]. Journal of Bacteriology,2011, 193(10): 2460-2467. DOI:10.1128/JB.01307-10.
[15] BISWAS A, GAGNON J N, BROUNS S J, et al. CRISPRTarget:Bioinformatic prediction and analysis of crrna targets[J]. RNA Biology, 2013, 10(5): 817-827. DOI:10.4161/rna.24046.
[16] MAKAROVA K S, HAFT D H, BARRANGOU R, et al. Evolution and classification of the CRISPR-Cas systems[J]. Nucleic Acids Research, 2011, 9(6): 467-477. DOI:10.1038/nrmicro2577.
[17] KUNIN V, SOREK R, HUGENHOLTZ P. Evolutionary conservation of sequence and secondary structures in CRISPR repeats[J]. Genome Biology, 2007, 8(4): R61. DOI:10.1186/gb-2007-8-4-r61.
[18] TANG T H, BACHELLERIE J P, ROZHDESTVENSKY T, et al.Identification of 86 candidates for small non-messenger RNAs from the archaeon Archaeoglobus fulgidus[J]. Proceedings of the National Academy of Sciences of the United States of America, 2002, 99(11):7536-7541. DOI:10.1073/pnas.112047299.
[19] 邓凯波, 霍贵成. 嗜热链球菌中CRISPR序列的检测与同源性分析[J].食品科学, 2013, 34(3): 153-158.
[20] 王鹏飞, 王颖芳, 段广才, 等. 志贺菌成簇的规律间隔短回文重复序列的检测及同源性分析[J]. 吉林大学学报(医学版), 2015, 41(2):261-269. DOI:10.13481/j.1671-587x.20150211.
[21] 焦雪, 张培军, 冷东泽, 等. 温和气单胞菌CRISPR位点的检测与分析[J]. 中国兽医学报, 2017, 37(5): 839-843. DOI:10.16303/j.cnki.1005-4545.2017.05.14.
[22] 王琰, 喻婵, 王阶平, 等. 蜡状芽孢杆菌群中规律成簇间隔短回文重复序列的生物信息学分析[J]. 中国生物工程杂志, 2011, 31(7): 72-78. DOI:10.13523/j.cb.20110712.
[23] PENG X, BRUGGER K, SHEN B, et al. Genus-specific protein binding to the large clusters of DNA repeats (short regularly spaced repeats) present in Sulfolobus genomes[J]. Journal of Bacteriology,2003, 185(8): 2410-2417. DOI:10.1128/JB.185.8.2410-2417.2003.
[24] KOONIN E V, MAKAROVA K S, ZHANG F. Diversity, classification and evolution of crispr-cas systems[J]. Current Opinion in Microbiology,2017, 37: 67-78. DOI:10.1016/j.mib.2017.05.008.
[25] GODDE J S, BICKERTON A. The repetitive DNA elements called CRISPRs and their associated genes: evidence of horizontal transfer among prokaryotes[J]. Journal of Molecular Evolution, 2006, 62(6): 718-729. DOI:10.1007/s00239-005-0223-z.
[26] MARRAFFINI L A, SONTHEIMER E J. CRISPR interference limits horizontal gene transfer in Staphylococci by targeting DNA[J]. Science,2008, 322: 1843-1845. DOI:10.1126/science.1165771.
[27] 贾珊珊, 李沛军, 陈从贵. 肉类食品中产气荚膜梭菌及其控制研究进展[J]. 肉类研究, 2016, 30(6): 45-51. DOI:10.15922/j.cnki.rlyj.2016.06.010.
[28] YOUNG K T, DAVIS L M, DIRITA V J. Campylobacter jejuni:molecular biology and pathogenesis[J]. Nature Reviews Microbiology,2007, 5(9): 665-679. DOI:10.1038/nrmicro1718.
[29] CHAKRABORTY S, SNIJDERS A P, CHAKRAVORTY R, et al.Comparative network clustering of direct repeats (DRs) and cas genes confirms the possibility of the horizontal transfer of CRISPR locus among bacteria[J]. Molecular Phylogenetics and Evolution, 2010, 56(3):878-887. DOI:10.1016/j.ympev.2010.05.020.
[30] LIU M, SIEZEN R J, NAUTA A. In silico prediction of horizontal gene transfer events in Lactobacillus bulgaricus and Streptococcus thermophilus reveals protocooperation in yogurt manufacturing[J].Applied and Environmental Microbiology, 2009, 75(12): 4120-4129.DOI:10.1128/AEM.02898-08.
[31] 申进玲, 吴瑜凡, 崔思宇, 等. 成簇的规律间隔短回文重复序列在致病菌中分型研究及应用[J]. 生物技术, 2016, 27(2): 186-192.DOI:10.16519/j.cnki.1004-311x.2017.02.0031.
[32] PARKHILL J, WREN B W, MUNGALL K, et al. The genome sequence of the food-borne pathogen Campylobacter jejuni reveals hypervariable sequences[J]. Nature, 2000, 403: 665-668. DOI:10.1038/35001088.
[33] RELMAN D A, FOUTS D E, MONGODIN E F, et al. Major structural differences and novel potential virulence mechanisms from the genomes of multiple Campylobacter species[J]. PLoS Biology, 2005, 3(1): e15.DOI:10.1371/journal.pbio.0030015.
[34] SHARIAT N, DIMARZIO M J, YIN S, et al. The combination of CRISPR-MVLST and PFGE provides increased discriminatory power for differentiating human clinical isolates of Salmonella enterica subsp.enterica serovar enteritidis[J]. Food Microbiology, 2013, 34(1): 164-173.DOI:10.1016/j.fm.2012.11.012.
[35] YIN S, JENSEN M A, BAI J, et al. The evolutionary divergence of Shiga toxin-producing Escherichia coli is reflected in Clustered Regularly Interspaced Short Palindromic Repeat (CRISPR) spacer composition[J].Applied and Environmental Microbiology, 2013, 79(18): 5710-5720.DOI:10.1128/AEM.00950-13.
[36] TORO M, CAO G, JU W, et al. Association of clustered regularly interspaced short palindromic repeat (CRISPR) elements with specific serotypes and virulence potential of Shiga toxin-producing Escherichia coli[J]. Applied and Environmental Microbiology, 2014, 80(4): 1411-1420. DOI:10.1128/AEM.03018-13.
[37] BRINER A E, BARRANGOU R. Lactobacillus buchneri genotyping on the basis of clustered regularly interspaced short palindromic repeat(CRISPR) locus diversity[J]. Applied and Environmental Microbiology,2014, 80(3): 994-1001. DOI:10.1128/AEM.03015-13.
[38] CALLEROS L, BETANCOR L, IRAOLA G, et al. Assessing the intra-species genetic variability in the clonal pathogen campylobacter fetus: CRISPRS are highly polymorphic DNA markers[J]. Journal of Microbiological Methods, 2017, 132: 86-94. DOI:10.1016/j.mimet.2016.11.012.
[39] HOOTON S P, BRATHWAITE K J, CONNERTON I F, The bacteriophage carrier state of campylobacter jejuni features changes in host non-coding rnas and the acquisition of new host-derived crispr spacer sequences[J]. Frontiers in Microbiology, 2016, 7: 355.DOI:10.3389/fmicb.2016.00355.
Detection and Structural Analysis of Clustered Regularly Interspaced Short Palindromic Repeat(CRISPR) Regions in Campylobacter jejuni