基于高通量测序的南美白对虾中微生物群落分析
邓晓影,张 宾*,汤 贺,郝桂娟,章样扬
(浙江海洋大学食品与医药学院,浙江省海产品健康危害因素关键技术研究重点实验室,浙江 舟山 316022)
摘 要:目的:分析不同状态下南美白对虾表面附着的微生物菌群组成情况。方法:以活体对虾、冰鲜对虾、冰鲜虾仁和冷冻虾仁为对象,通过对虾表面附着微生物提取及高通量测序分析,获得不同对虾样品中微生物组成及变化情况。结果:1)各样本有效序列范围为30 591~42 043 条、操作分类单元(operational taxonomic units,OTU)范围为68~168,其中冷冻虾仁OTU数量最低,而活体对虾和冰鲜对虾OTU数量相对较高。2)多样性指数分析发现,所有样品中以冷冻虾仁的ACE、Chao1和Shannon指数值最低、Simpson指数最高,表明其菌群丰度及多样性较低。3)在科水平上,相比于活体对虾,冰鲜对虾中莫拉氏菌科(Moraxellaceae)相对丰度显著增加,其可能与虾体死亡、微生物污染繁殖有关;冰鲜虾仁弧菌科(Vibrionaceae)相对丰度显著增加且含量最高,主要是虾仁加工制备环境及加工器具中微生物接触污染所致;而冷冻虾仁中主要以莫拉氏菌科(Moraxellaceae)、假单胞菌科(Pseudomonadaceae)、李斯特菌科(Listeriaceae)为腐败优势菌。4)热图分析发现,活体对虾和冰鲜对虾菌群结构整体相似度较高,二者与冰鲜虾仁、冷冻虾仁的相似度较低,表明虾仁制备过程、低温冻藏对于虾仁中菌群结构影响较大。5)主成分分析表明,冷冻虾仁与其他组别样本均相距较远,说明冷冻虾仁中微生物种类及组成与其他样品存在着较大差异。结论:南美白对虾样品处于不同状态时,其附着优势微生物、菌群结构、丰度及相互比例等存在显著差异,需针对于不同类型虾类制品,采取不同的控制工艺以保障虾类制品的质量与安全。
关键词:南美白对虾;表面微生物;高通量测序;菌群分析
南美白对虾(Litopenaeus vannamei)学名凡纳滨对虾,是世界对虾养殖中产量较高的三大优良虾种之一,同时也是目前养殖对虾中单产量最高的虾种[1]。南美白对虾作为一种高度易腐水产品,虾体死亡后,污染微生物及其代谢物和体内内源酶均可加速虾体品质劣变及腐败进程,因此南美白对虾捕后极不耐贮藏、商品货架期相对较短。南美白对虾受生长环境、加工工艺及贮藏过程的影响,虾体表面附着的微生物通常数量较多,且种类组成也十分复杂[2-4]。这些微生物不仅影响着南美白对虾及其制品的食用安全,而且对南美白对虾贮藏期间的品质保障起到主要作用[5]。
近年来发展起来的宏基因组学技术避开了传统微生物的分离培养方法,而直接从测试样品中提取总DNA进行分析,能更完整地反映样品中微生物的群落特征。目前,高通量测序技术已成为现代生命科学研究的常用技术,其实现了同时对几百万DNA分子的测序,达到了鉴定微生物单一基因或全基因组的目的。高通量测序主要有454焦磷酸测序平台(Roche公司)、HiSeq和MiSeq测序平台(Illumina公司)以及Solid测序平台(ABI公司)。近年来,随着现代测序技术快速发展,研究者已将高通量测序技术成功应用于多个学科领域,如土壤多样化,海洋和肠道环境、发酵食品、人类健康与疾病等方面[6-7]。目前,关于虾类中微生物菌群研究,绝大部分集中于养殖水体微生物多样性、虾肠道微生物菌群结构及冷冻虾类产品中细菌菌群结构等,而关于对虾在不同鲜度及加工过程中的菌群变化情况还鲜见报道[8]。本实验以不同状态下的南美白对虾为研究对象,采用Illumina MiSeq高通量测序技术分析对虾样品中的菌群组成情况,为南美白对虾制品的品质保障与质量安全提供基础信息。
1 材料与方法
1.1 材料、试剂与仪器
活南美白对虾(Litopenaeus vannamei)(体长14~16 cm),2017年购自浙江舟山市东河水产品市场,将活虾置于呈有养殖水体的暂存箱内,15~20 min内运至实验室进行处理。
TransStart FastPfu DNA Polymerase 美国Qiagen公司;TruSeqTMDNA Sample Prep Kit等测序用试剂 美国Illumina公司;AxyPrepDNA凝胶回收试剂盒 美国Axygen Biosciences公司。
GeneAmp®9700型聚合酶链式反应(polymerize chain reaction,PCR)仪 美国ABI公司;QuantiFluor™-ST蓝色荧光定量系统 美国Promega公司。
1.2 方法
1.2.1 实验分组与预处理
将南美白对虾分为以下处理组:活体对虾(带壳整虾)、冰鲜对虾(4 ℃条件下死亡3~5 h后的鲜虾)、冰鲜虾仁(实验室条件下,将死亡鲜虾去头壳,不去虾肠,获得虾仁)和冷冻虾仁(冰鲜虾仁密封于无菌袋中,-18 ℃冻藏4 周后的虾仁)。
1.2.2 虾体表面微生物提取[9]
将以上各组虾类样品,在无菌三角瓶中加入9 倍体积的无菌生理盐水后,室温条件下在培养摇床中以300 r/min提取60 min,将虾体表面微生物提取到无水生理盐水中。采用纱布过滤、无菌水淋洗后,滤液以12 000 r/min离心15 min,将离心获得菌体沉淀物进行微生物菌落分析。
1.2.3 虾体表面微生物群落分析
首先采用基因组DNA提取试剂盒(按产品说明书进行)对虾体表面微生物进行基因组DNA抽提,进而利用1%琼脂糖凝胶电泳,检测抽提的基因组DNA;按指定测序区域,合成带有条码特异引物,进行PCR扩增。针对16S rRNA基因V3/V4区合成特异引物,利用338F(5’-ACTCCTACGGGAGGCAGCA-3’)和806R(5’-GGACTACHVGGGTWTCTAAT-3’)引物进行PCR扩增。采用AxyPrepDNA凝胶回收试剂盒切胶回收PCR产物,QuantiFluor™-ST蓝色荧光定量系统进行检测定量,而后进行Illumina MiSeq 2×300 bp高通量测序及生物信息学分析。
1.2.4 虾体表面微生物信息分析
对MiSeq测序得到的PE reads,首先依据overlap关系进行拼接,同时对序列质量进行质控和过滤,区分样本后进行操作分类单元(operational taxonomic units,OTU)聚类分析和物种分类学分析。基于OTU聚类分析结果,进行多种多样性指数分析,以及对测序深度进行检测。基于分类学信息,在各个分类水平上进行群落结构统计[10]。在上述分析基础上,最后进行群落结构和系统发育等统计学和可视化分析(具体由微基生物科技(上海)有限公司协助完成)。
2 结果与分析
2.1 测序数据统计
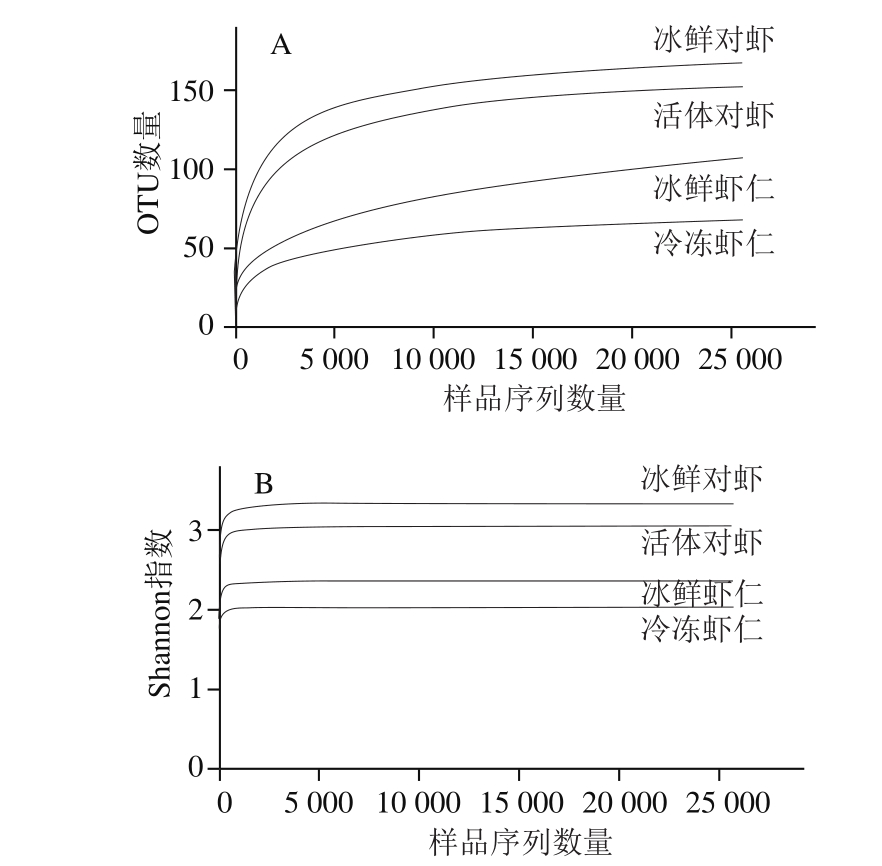
采用Illumina高通量测序获得不同对虾样本原始序列数据后,采用Trimmomatic软件进行序列质控,Flash软件进行序列拼接,再经序列优化及统计后得到各实验组有效序列范围为30 591~42 043条,其平均长度范围为444.73~449.87 bp。其中,有效序列长度范围在441~460 bp之间的序列,占有效序列总数的95.61%。进而对样品序列进行聚类分析,在97%相似水平下的OTU生物信息统计发现,各处理组得到的OTU数量范围为68~168,其中冷冻虾仁OTU数量最低为68,而活体对虾和冰鲜对虾OTU数量较高,分别为153和168(表1)。由以上聚类结果可知,活体对虾和冰鲜对虾中特有微生物相对种类较多,而冷冻后虾仁中微生物种类明显减少,其原因一方面来自于虾壳及头部的去除减少了部分微生物,更重要的是低温冷冻对部分微生物也产生了较强的抑制或杀灭作用[11-13]。
表1 各组样品有效序列与OTU数量统计
Table 1 Numbers of valid sequences and OTUs in different shrimp samples
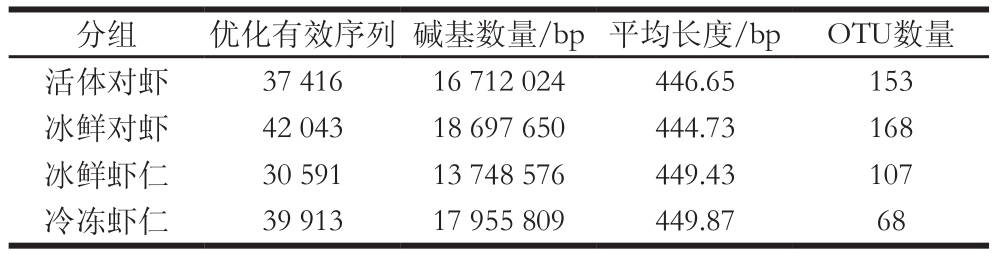
分组 优化有效序列 碱基数量/bp 平均长度/bp OTU数量活体对虾 37 416 16 712 024 446.65 153冰鲜对虾 42 043 18 697 650 444.73 168冰鲜虾仁 30 591 13 748 576 449.43 107冷冻虾仁 39 913 17 955 809 449.87 68
2.2 多样性指数分析
可通过单样本多样性(Alpha多样性)分析反映微生物群落的丰度和多样性。各样本文库覆盖率的数值越高,则样本中序列被测出的概率越高,而没有被测出的概率越低,直接反映测序结果是否代表样本中微生物的真实情况[14-15]。由表2可知,所有样本测序覆盖率均在0.998以上,说明测序对样本中细菌的覆盖率很高,测序深度适合,可满足样品中细菌多样性分析的需要[16]。此外,当样品测序数量超过20 000时,各样品稀释曲线均趋向饱和(图1A),表明样品测序数据量合理,更多数据量只会产生少量新OTU[17]。同时,Shannon-Wiener曲线趋也逐渐向平坦(图1B),说明测序数据量足够大,可反映样本中绝大多数的微生物信息。上述现象表明,随着测序量增加新的菌系不会被发现,微生物多样性也不会发生显著变化,菌群分析结果合理、充分。
样品菌群丰度的表征主要有ACE和Chao1指数,而菌群多样性的表征主要有Shannon和Simpson指数[18]。相比于活体对虾,死亡后冰鲜对虾样品的ACE、Chao1和Shannon指数值升高、Simpson指数降低(表2),表明虾体死亡及冰鲜贮藏过程中微生物污染及繁殖不断加剧,该阶段也是虾体微生物种类及丰度显著增加的阶段。对于冰鲜虾仁样品,其ACE、Chao1和Shannon指数均低于活虾和冰鲜虾样品,即去头、壳过程可一定程度上降低虾仁中微生物的多样性,但需要指出的是由于实际加工过程往往为非无菌操作,也致使虾仁中残留相当数量的微生物甚至有污染致病微生物的可能。相比于其他各组,冷冻虾仁样品的ACE、Chao1和Shannon指数最低,Simpson指数最高,表明其菌群丰度及多样性较低,其原因可能由于低温作用抑制了部分微生物的生长与繁殖,致使虾仁中微生物结构发生了显著性变化。
表2 各组样品的多样性指数分析
Table 2 Bacterial community diversity of different shrimp samples
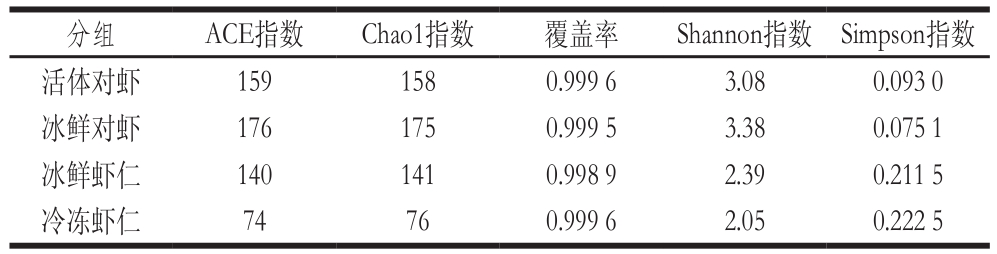
分组 ACE指数 Chao1指数 覆盖率 Shannon指数 Simpson指数活体对虾 159 158 0.999 6 3.08 0.093 0冰鲜对虾 176 175 0.999 5 3.38 0.075 1冰鲜虾仁 140 141 0.998 9 2.39 0.211 5冷冻虾仁 74 76 0.999 6 2.05 0.222 5
2.3 不同分类水平上的物种注释及分析
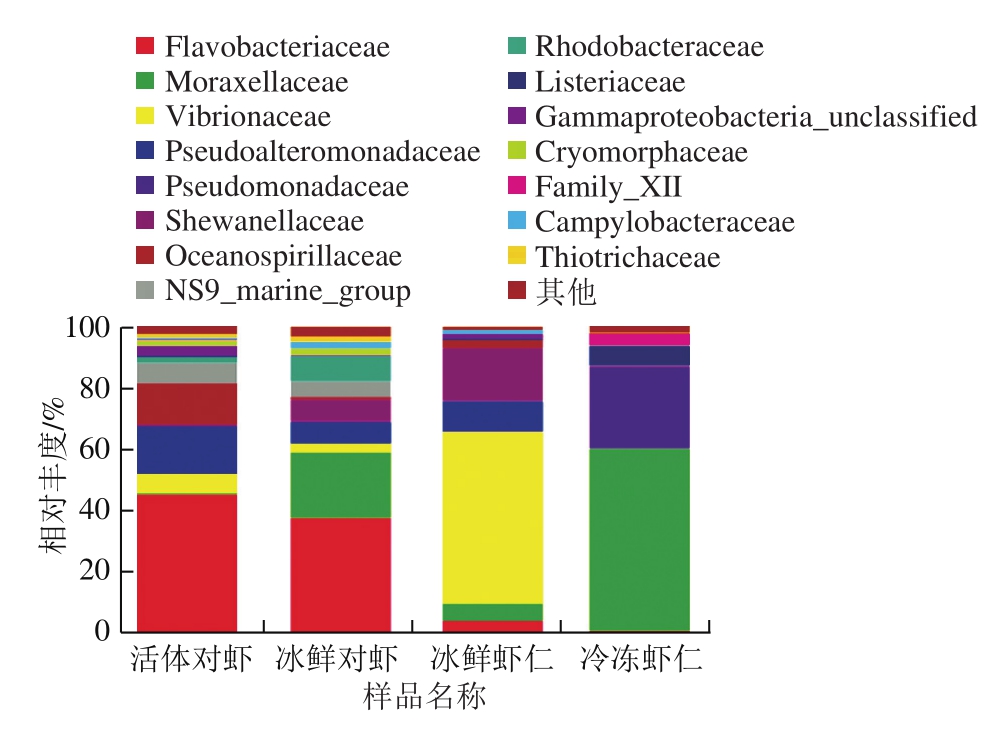
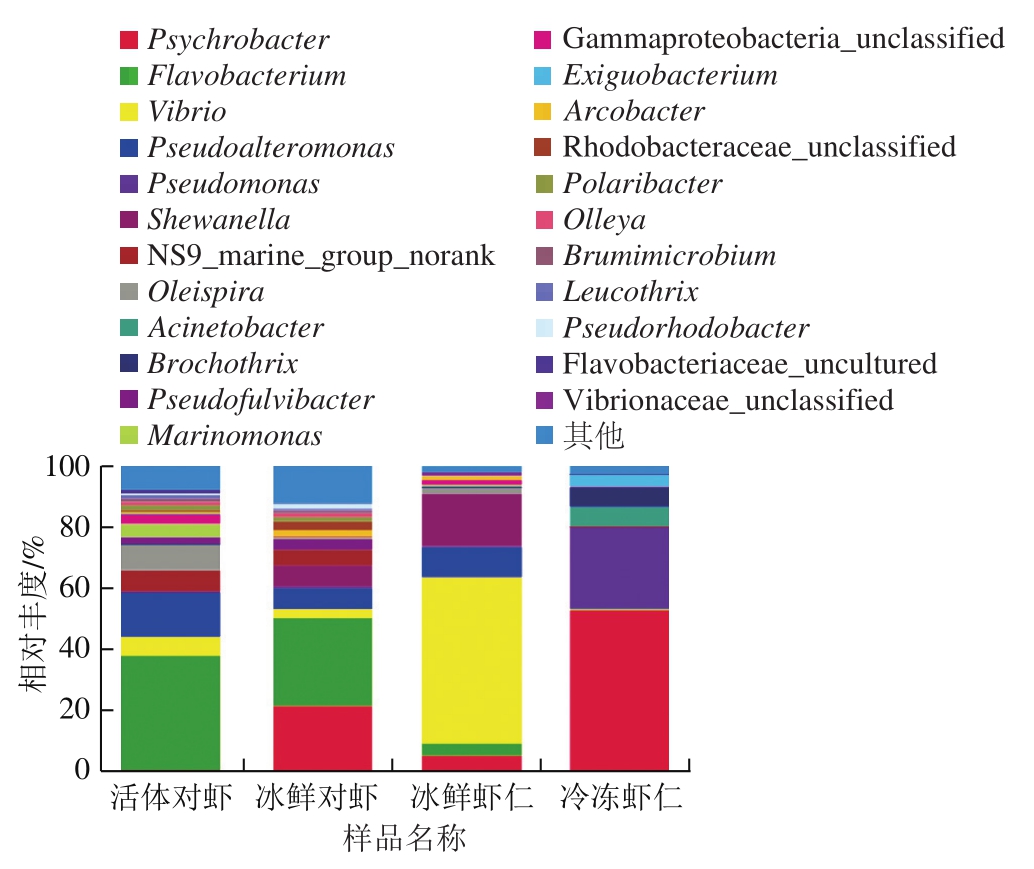
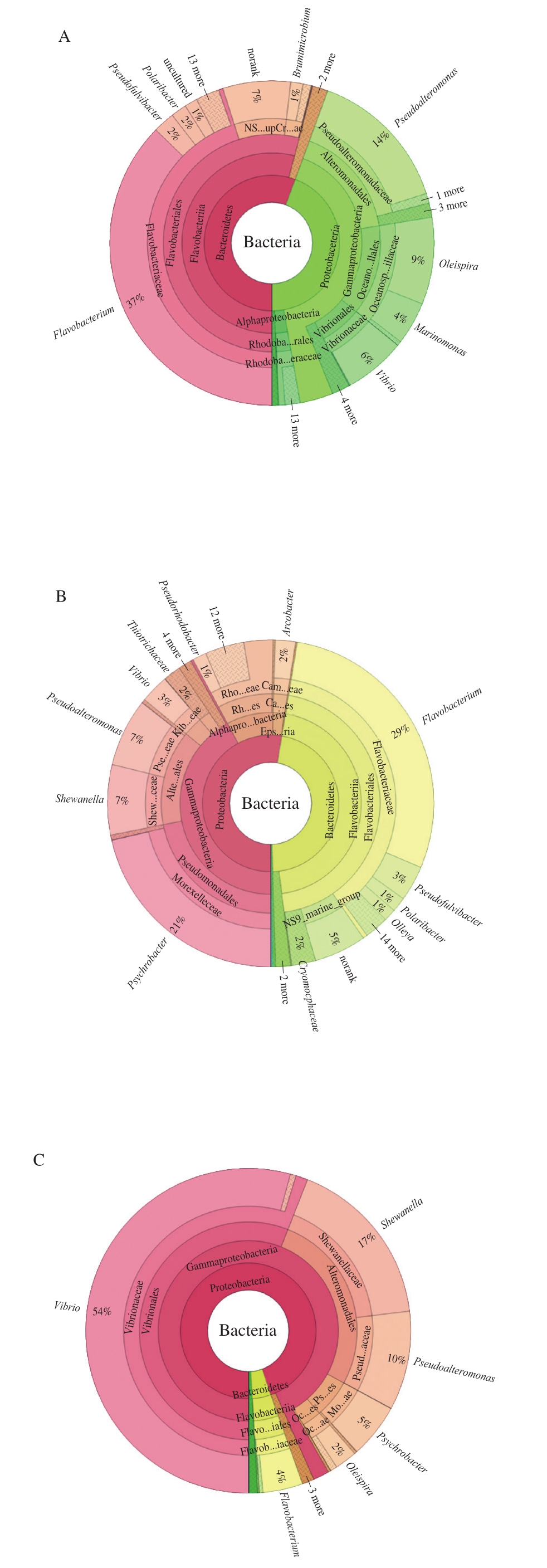
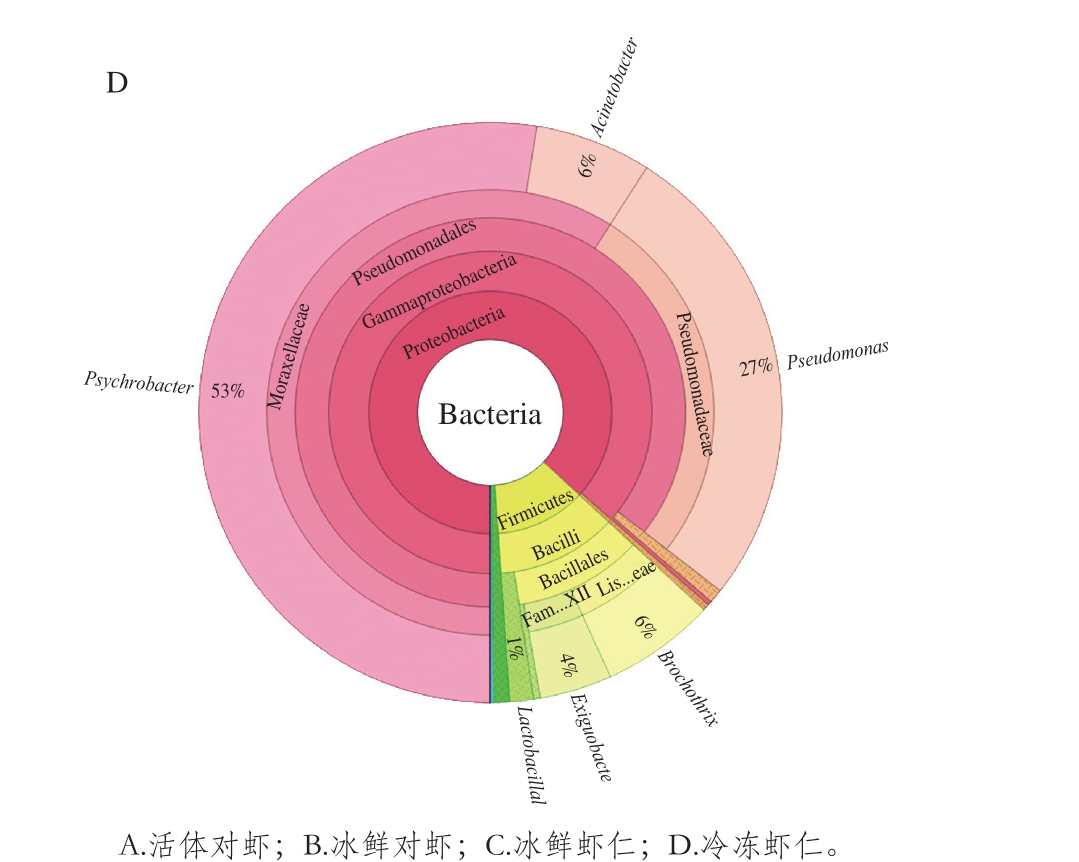
为获得每个OTU对应物种分类信息,采用RDP classifier贝叶斯算法对OTU序列进行分类学分析,并分别在科(图2)和属(图3)水平上统计各样本群落组成。
在科分类水平上(图2),活体对虾表面主要微生物为黄杆菌科(Flavobacteriaceae)、假交替单胞菌科(Pseudoalteromonadaceae)和海洋螺菌科(Oceanospirillaceae),以上3 个菌科微生物占菌群总量的73.91%。冰鲜对虾中主要微生物为黄杆菌科(Flavobacteriaceae)、莫拉氏菌科(Moraxellaceae)、红曲菌科(Rhodobacteraceae)、希瓦氏菌科(Shewanellaceae)以及假交替单胞菌科(Pseudoalteromonadaceae),占菌群总量的80.97%。相比于活虾,莫拉氏菌科相对丰度变化显著,由活虾中的0.48%升到21.39%。虾体死亡、贮藏时间不断延长,其微生物种类及数量增加、内源酶活性突显,虾仁品质逐渐下降。冰鲜对虾中莫拉氏菌科含量增加,可能与虾体死亡、微生物污染繁殖有关。曹荣等[19]研究牡蛎冷藏过程中菌落分析发现,冷藏4 d后牡蛎附着的莫拉氏菌科丰度也显著增加。冰鲜虾仁中主要为弧菌科(Vibrionaceae)、希瓦氏菌科(Shewanellaceae)、假交替单胞菌科(Pseudoalteromonadaceae)以及莫拉氏菌科(Moraxellaceae),占菌群总量的88.33%。其中,弧菌科丰度最高(55.96%)、希瓦氏菌科次之(17.1%)。相比于冰鲜对虾,二者丰度值显著增加,成为冰鲜虾仁表面的优势微生物,其来源可能是实验室环境中的微生物,以及虾仁制备过程中由加工器具等转移污染的微生物。此外,黄杆菌科(Flavobacteriaceae)和莫拉氏菌科(Moraxellaceae)的丰度明显减少,表明该两种菌科微生物可能较多的分布于虾壳表面,因此在虾仁剥壳及去头过程中丰度显著降低。冷冻虾仁中莫拉氏菌科(Moraxellaceae)为优势菌种,丰度占总菌群59.02%,其次为假单胞菌科(Pseudomonadaceae)(26.50%)、李斯特菌科(Listeriaceae)(6.30%)。研究表明,莫拉氏菌科(Moraxellaceae)和假单胞菌科(Pseudomonadaceae)为典型嗜冷菌,其为冷冻肉制品、冷冻水产品等的腐败微生物,且李斯特菌科对酸碱度适应范围广,其在生冰箱冷藏温度、低酸及高盐环境中仍可生长[20]。此外,和冰鲜虾仁相比,冷冻虾仁中弧菌科和希瓦氏菌科明显减少,主要与微生物自身低温耐受性有关,二者会随着冻藏温度的降低及冻藏时间的延长而逐渐减少。
在属分类水平上(图3),活体对虾中主要微生物为黄杆菌属(Flavobacterium)、假交替单胞菌(Pseudoalteromonas)和油螺旋菌属(Oleispira)。冰鲜对虾中主要微生物为黄杆菌属(Flavobacterium)、嗜冷菌属(Psychrobacter)、希瓦氏菌(Shewanella)和假交替单胞菌属(Pseudoalteromonas)。孙振丽等[21]研究发现,活体对虾的肠道中主要优势细菌属为假单胞菌属(Pseudomonas)、不动杆菌属(Acinetobacter)、黄杆菌属(Flavobacterium)等,该研究结果与本实验测定结果较为相似,也表明对虾表面与其肠道内细菌组成较为相近。相比于活体对虾,冰鲜对虾中嗜冷菌属(Psychrobacter)显著增加。郑振霄等[22]研究发现,冰鲜鲐鱼(-1~0 ℃)中微生物组成比较复杂,其优势种群为嗜冷菌属(Psychrobacter)和假单胞菌属(Pseudomonadaceae),且随着贮藏时间延长,鲐鱼样品中菌相逐渐变的单一。冰鲜虾仁中优势菌群为弧菌属(Vibrio),其丰度占总菌群的54.20%,次优势菌群为希瓦氏菌(Shewanella)和假交替单胞菌属(Pseudoalteromonas),分别占总菌群的17.10%和9.86%。弧菌属(Vibrio)包含有80多种,其中为病原菌,如霍乱弧菌、副溶血弧菌、创伤弧菌等[23-24]。由以上结果可知,虾仁制备过程中,为弧菌属污染主要阶段,其可能对虾类制品质量安全造成隐患。冷冻虾仁附着优势微生物主要为嗜冷菌属(Psychrobacter)、假单胞菌属(Pseudomonas)、不动杆菌属(Acinetobacter)和环丝菌属(Brochothrix),因此对于冷冻虾仁品质的调控,应重点针对以上几种菌属进行。低温贮藏环境虽可减少虾仁中微生物种类,但仍存在以上几种典型嗜冷微生物菌属,因此针对冷冻虾仁类产品,需对以上几种菌属进行重点调控。
2.4 菌群样本组成分析
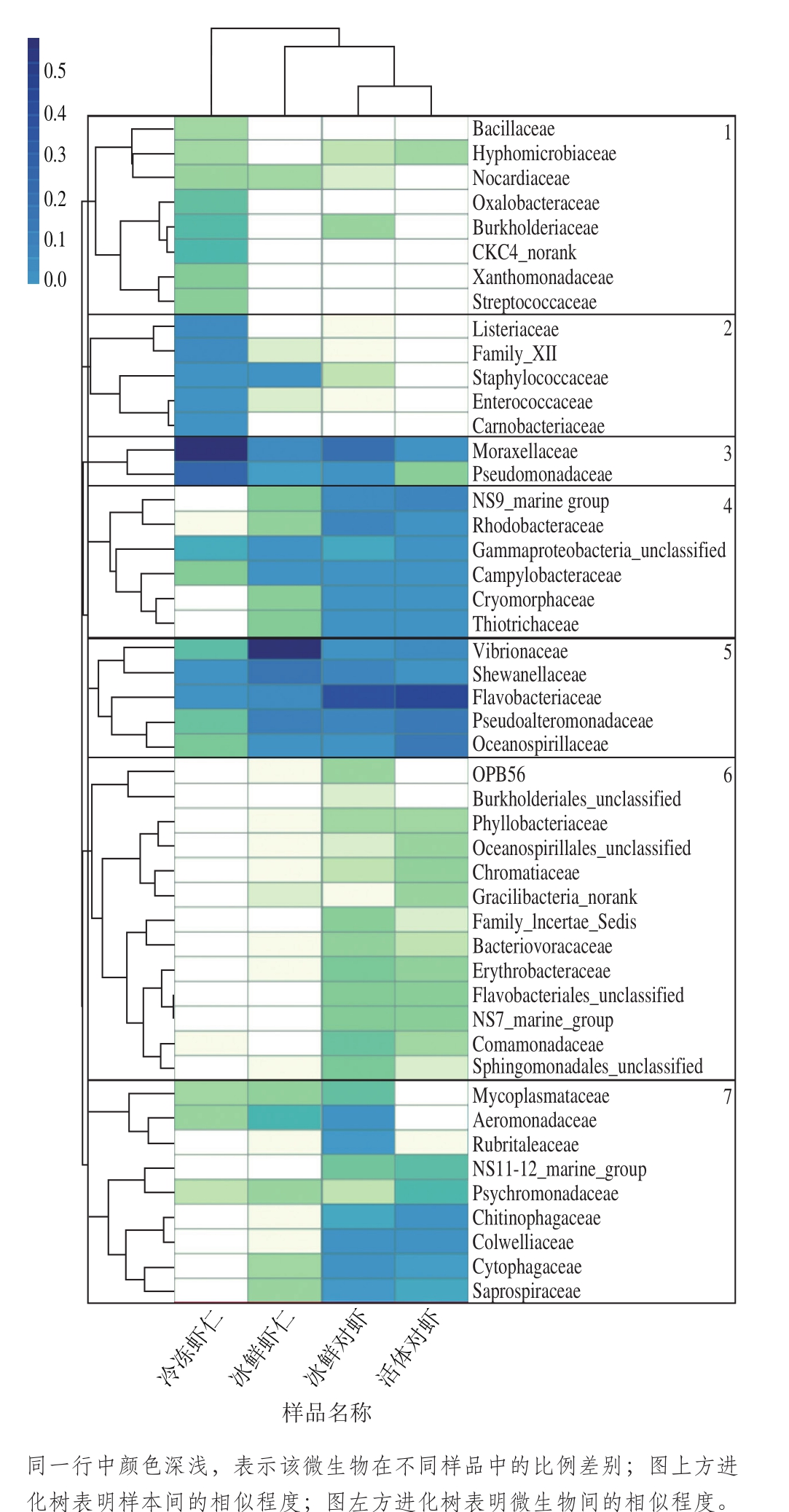
热图颜色变化反映二维矩阵或表格中数据信息,可直观地将数据值大小以定义颜色深浅表示出来[25-27]。在热图上,将高丰度和低丰度物种分块聚集,通过颜色梯度及相似程度反映多个样本群落组成相似性和差异性[28]。从物种和样本两个层面对OTU进行聚类,绘制物种丰度热图,结果如图4所示。由样本间菌群结构相似度(图4,上方进化树)分析发现,活体对虾和冰鲜对虾聚为一类,而冰鲜虾仁、冷冻虾仁各自单独聚为一类。由此,活体对虾和冰鲜对虾菌群结构整体相似度较高,其与冰鲜虾仁、冷冻虾仁相似度较差,表明虾仁制备过程、低温冻藏对于虾仁中菌群结构影响较大。该结果与以上物种分析结果相一致。
对于菌门丰度聚类分析发现,无论是活体、还是冰鲜对虾样品,在微生物组成中比例较大的均为黄杆菌科(Flavobacteriaceae)、假交替单胞菌科(Pseudoalteromonadaceae)。在活体对虾-死亡(冰鲜)对虾-冰鲜虾仁-冷冻虾仁加工及贮藏过程中,以伯克氏菌科(Burkholderiaceae)、李斯特菌科(Listeriaceae)、葡萄球菌科(Staphylococcaceae)及肠球菌科(Enterococcaceae)等为代表的菌科含量,在冷冻虾仁中明显增加,这可能是因为以上几种菌科环境适应能力较强,在冷藏过程中菌群数量逐渐增加,成为南美白对虾冷藏中的优势菌群。而以黄杆菌科(Flavobacteriaceae)、NS9_marine group、蟑螂杆状体科(Cryomorphaceae)、弧菌科(Vibrionaceae)、着色菌科(Chromatiaceae)、腐螺旋菌科(Saprospiraceae)等为代表的菌科含量明显减少,其原因主要是以上几种菌科主要附着在虾体表面及大部分不耐低温特性而造成。不同对虾样品在域、门、纲、目、科等分类学水平上的物种比例和分布,如图5所示。总体来看,对虾样品处于不同状态下,其优势微生物、菌群结构、相对丰度及相互比例,均发生了显著性变化,因此,针对于不同类型的虾类产品,需采取不同的控制条件及工艺技术,以保障虾类产品的质量与安全。
2.5 样本主成分分析(principal component analysis,PCA)比较
PCA可将原有的复杂数据降维,揭示隐藏在复杂数据背后的简单结构[29]。通过分析不同样本OTU(97%相似性)组成,可以反映样本间的差异和距离,PCA运用方差分解,将多组数据的差异反映在二维坐标图上,如样本组成越相似,反映在PCA图中相应坐标轴上的距离越近[30-33]。南美白对虾样本PCA结果,如图6所示。对于PC1、PC2,活体对虾和冰鲜对虾样本较为相近,说明在这一主成分水平上活虾和刚死亡的冰鲜虾的菌群特征较为相近。对于PC1~3分析,对于活体对虾、冰鲜对虾、冰鲜虾仁和冷冻虾仁4 组样本,PC1和PC3方面均距离相对较远,以上4 种样品在几种特定微生物组成上存在着较大的差异。对于PC2、PC3分析中,在PC2上,冰鲜对虾和冷冻虾仁区分度明显,说明虾仁的去壳制备过程对其附着微生物组成具有较大影响,该结果与物种注释分析结果一致。总体来看,在各个主成分坐标轴上,冷冻虾仁与其他组别样本均相距较远,说明冷冻虾仁中微生物种类及组成与其他样品具有较大的差异。
3 结 论
以不同状态下南美白对虾为研究对象,采用Illumina高通量测序技术分析各样品中菌群组成及变化情况。结果表明,活体对虾、冰鲜对虾、冰鲜虾仁以及冷冻虾仁中菌群组成差异较大,其中活体对虾中主要为黄杆菌科、假交替单胞菌科及海洋螺菌科;冰鲜对虾中主要为黄杆菌科、莫拉氏菌科、红曲菌科、希瓦氏菌科及假交替单胞菌科;冰鲜虾仁中主要为弧菌科、希瓦氏菌科、假交替单胞菌科及莫拉氏菌科;而冷冻虾仁中微生物主要为莫拉氏菌科、假单胞菌及李斯特菌科。需要注意的是,冰鲜虾仁中优势菌为弧菌科,冷冻虾仁中以莫拉氏菌科、假单胞菌科及李斯特菌科为优势菌科。这几个菌科对于南美白对虾质量安全与品质保障具有重要影响作用,在加工及贮藏过程中应加以重点关注。
参考文献:
[1] ZENG D G, CHEN X L, XIE D X, et al. Analysis on differential gene expression in shrimp Litopenaeus vannamei induced by IHHNV infection using high-throughput sequencing[J]. Journal of Southern Agriculture, 2013, 44(11): 1899-1903.
[2] ZHANG B, MA L K, DENG S G, et al. Shelf-life of pacific white shrimp (Litopenaeus vannamei) as affected by weakly acidic electrolyzed water ice-glazing and modified atmosphere packaging[J]. Food Control,2015, 51: 114-121. DOI:10.1016/j.foodcont.2014.11.016.
[3] 沈辉, 万夕和, 何培民, 等. 脊尾白虾肠道微生物菌群结构[J]. 微生物学通报, 2015, 42(10): 1922-1928. DOI:10.13344/j.microbiol.china.140959.
[4] DI B J, BAO Y, GLOOR G B, et al. High throughput sequencing methods and analysis for microbiome research[J]. Journal of Microbiological Methods, 2013, 95(3): 401-414. DOI:10.1016/j.mimet.2013.08.011.
[5] 陈琼, 李贵阳, 罗坤, 等. 凡纳滨对虾(Litopenaeus vannamei)亲虾繁殖期水体微生物多样性[J]. 海洋与湖沼, 2017, 48(1): 130-138.DOI:10.11693/hyhz20160700176.
[6] KRAKOVÁ L, ŠOLTYS K, BUDIŠ J, et al. Investigation of bacterial and archaeal communities: novel protocols using modern sequencing by Illumina MiSeq and traditional DGGE-cloning[J]. Extremophiles, 2016,20(5): 1-14. DOI:10.1007/s00792-016-0855-5.
[7] LU X M, LU P Z. Characterization of bacterial communities in sediments receiving various wastewater effluents with high-throughput sequencing analysis[J]. Microbial Ecology, 2014, 67(3): 612-323.
[8] MIGET R J. Microbiology of crustacean processing: shrimp, crawfish,and prawns[M]//Microbiology of marine food products. US: Springer,1991: 65-87. DOI:10.1007/978-1-4615-3926-1_4.
[9] PREIDIS G A, AJAMI N J, WONG M C, et al. Composition and function of the undernourished neonatal mouse intestinal microbiome[J].Journal of Nutritional Biochemistry, 2015, 26(10): 1050-1057.DOI:10.1016/j.jnutbio.2015.04.010.
[10] HU D X, PAN L Q, ZHAO Q, et al. Transcriptomic response to low salinity stress in gills of the Pacific white shrimp, Litopenaeus vannamei[J]. Marine Genomics, 2015, 24: 297-304. DOI:10.1016/j.margen.2015.07.003.
[11] 曲巍, 张智, 马建章, 等. 高通量测序研究益生菌对小鼠肠道菌群的影响[J]. 食品科学, 2017, 38(1): 214-219. DOI:10.7506/spkx1002-6630-201701036.
[12] 邵晖, 吴玲玲, 王伟, 等. 食品中病原微生物检测方法进展及优缺点评价[J]. 中国食品工业, 2008(4): 40-43.
[13] 原林, 丁长河. 冷藏食品中的常见微生物及其防控[J]. 粮食与食品工业, 2016, 23(6): 29-33. DOI:10.3969/j.issn.1672-5026.2016.06.008.
[14] RAHIMI E, SHAKERIAN A, RAISSY M. Prevalence of Listeria,species in fresh and frozen fish and shrimp in Iran[J]. Annals of Microbiology, 2012, 62(1): 37-40. DOI:10.1007/s13213-011-0222-9.
[15] 刘柳, 孔保华. 温度及气调包装对冷却猪肉中单核细胞增生性李斯特菌生长的影响[J]. 食品科学, 2008, 29(1): 334-337. DOI:10.3321/j.issn:1002-6630.2008.01.075.
[16] LI K, ZHUO C, TENG C, et al. Effects of Ganoderma lucidum polysaccharides on chronic pancreatitis and intestinal microbiota in mice[J]. International Journal of Biological Macromolecules, 2016, 93:904-912. DOI:10.1016/j.ijbiomac.2016.09.029.
[17] HAMADY M, LOZUPONE C, KNIGHT R. Fast UniFrac: facilitating high-throughput phylogenetic analyses of microbial communities including analysis of pyrosequencing and PhyloChip data[J]. ISME Journal, 2010, 4(1): 17-27. DOI:10.1016/0090-4295(88)90230-0.
[18] 温崇庆, 何瑶瑶, 薛明, 等. 高通量测序分析DNA提取引起的对虾肠道菌群结构偏差[J]. 微生物学报, 2016, 56(1): 130-142. DOI:10.13343/j.cnki.wsxb.20150164.
[19] 曹荣, 刘淇, 赵玲, 等. 基于高通量测序的牡蛎冷藏过程中微生物群落分析[J]. 农业工程学报, 2016, 32(20): 275-280. DOI:10.11975/j.issn.1002-6819.2016.20.036.
[20] 崔宏博, 薛勇, 宿玮, 等. 南美白对虾即食虾仁加工工艺和贮藏研究[J].食品科学, 2012, 33(4): 257-261.
[21] 孙振丽, 宣引明, 张皓, 等. 南美白对虾养殖环境及其肠道细菌多样性分析[J]. 中国水产科学, 2016, 23(3): 594-605. DOI:10.3724/SP.J.1118.2016.15255.
[22] 郑振霄, 周聃, 冯俊丽, 等. 冷海水保藏下鲐鱼(Pneumatophorus japonicus)菌相变化规律及优势腐败菌的分离鉴定[J]. 现代食品科技, 2016, 32(4): 81-86. DOI:10.13982/j.mfst.1673-9078.2016.4.014.
[23] 杨俊花, 赵志辉, 郭文博, 等. 应用Illumina-MiSeq高通量测序技术分析脱氧雪腐镰刀菌烯醇对小鼠肠道菌群的影响[J]. 动物营养学报,2017, 29(1): 158-167. DOI:10.3969/j.issn.1006-267x.2017.01.018.
[24] 金春英. 南美白对虾弧菌病病原的分离和鉴定[J]. 海洋环境科学,2013, 32(5): 647-651.
[25] 刘金昉, 刘红英, 齐凤生, 等. 纳米TiO2/壳聚糖复合保鲜剂在南美白对虾保鲜中的应用[J]. 食品科技, 2014, 39(2): 245-249.
[26] 江艳华, 姚琳, 李风铃, 等. 基于高通量测序的冷冻南极磷虾中细菌菌群结构分析[J]. 食品安全质量检测学报, 2016, 7(7): 2840-2845.
[27] 王江博. 一株莫拉氏菌新种的分离鉴定与特性研究[D]. 成都: 四川农业大学, 2016.
[28] WANG Q, GARRITY G M, TIEDJE J M, et al. Naive Bayesian classifier for rapid assignment of rRNA sequences into the new bacterial taxonomy[J]. Applied and Environmental Microbiology, 2007, 73(16):5261-5267. DOI:10.1128/AEM.00062-07.
[29] ABRAHAMSSON T, JAKOBSSON H, ANDERSSON A, et al. Low diversity of the gut microbiota in infants with atopic eczema[J]. Journal of Allergy and Clinical Immunology, 2012, 129(2): 37-41. DOI:10.1016/j.jaci.2011.10.025.
[30] 王凌燕. 南美白对虾腐败微生物分析与防黑变保鲜技术研究[D]. 青岛:青岛大学, 2012.
[31] 沈飚, 胡兴娟, 汪云泉, 等. 冷冻虾仁抑菌效果影响因素研究[J]. 食品工业科技, 2011, 32(5): 339-341. DOI:10.13386/j.issn1002-0306.2011.05.075.
[32] 田凤, 王玲. 湛江地区养殖南美白对虾冷藏期间的菌相分析及优势腐败菌的初步鉴定[J]. 食品与发酵工业, 2013, 39(2): 24-28.DOI:10.13995/j.cnki.11-1802/ts.2013.02.031.
[33] PORRAS D, NISTAL E, MARTÍNEZ-FLÓREZ S, et al. Protective effect of quercetin on high-fat diet-induced non-alcoholic fatty liver disease in mice is mediated by modulating intestinal microbiota imbalance and related gut-liver axis activation[J]. Free Radical Biology & Medicine,2017, 102: 188-202. DOI:10.1016/j.freeradbiomed.2016.11.037.
Analysis of Microbial Diversity in Shrimp (Litopenaeus vannamei) by MiSeq High-Throughput Sequencing
DENG Xiaoying, ZHANG Bin*, TANG He, HAO Guijuan, ZHANG Yangyang
(Key Laboratory of Health Risk Factors for Seafood of Zhejiang Province, College of Food Science and Pharmacy,Zhejiang Ocean University, Zhoushan 316022, China)
Abstract:Objective: To evaluate the composition of microbial communities adhered to the surface of shrimp (Litopenaeus vannamei) under different conditions. Methods: Genomic DNA was extracted from bacteria adhered to the surface of live shrimp, fresh shrimp, fresh peeled shrimp and frozen peeled shrimp and was sequenced by using high-throughput sequencing.Results: 1) A total of 30 591–42 043 valid sequences and 68–168 operational taxonomic units (OTUs) were obtained from each sample, and the number of OTUs was lowest in frozen peeled shrimp and relatively high in live shrimp and fresh shrimp.2) Among all samples investigated, frozen peeled shrimp exhibited the lowest ACE, Chao1 and Shannon indices and highest Simpson index, suggesting lower bacterial abundance and diversity. 3) At the family level, compared to live shrimp, the relative abundance of Moraxellaceae in fresh shrimp was significantly increased, which might be related to the death of shrimp and microbial contamination and growth; the abundance of Vibrionaceae in fresh peeled shrimp was significantly increased and the highest content was observed, which was caused by microbial contamination from the environment and the devices used during shrimp processing. The dominant spoilage bacteria in frozen peeled shrimp were Moraxellaceae, Pseudomonadaceae, and Listeriaceae. 4) Heatmap analysis showed that the similarity in bacterial flora between live shrimp and fresh shrimp was higher,whereas they were significantly different from fresh peeled shrimp and frozen peeled shrimp, indicating that the preparation of peeled shrimp and frozen storage had a great impact on shrimp bacterial flora. 5) Principal component analysis (PCA) indicated that there was significantly difference in microbial community composition between frozen peeled shrimp and other samples.Conclusion: There were significant differences in the dominant microorganisms, flora structure, abundance and composition among shrimp samples under different conditions. Different control processes are necessary for different types of shrimp products to ensure the quality and safety of shrimp products.
Keywords:Litopenaeus vannamei; surface microorganism; high-throughput sequencing; microbial diversity
DENG Xiaoying, ZHANG Bin, TANG He, et al. Analysis of microbial diversity in shrimp (Litopenaeus vannamei) by MiSeq high-throughput sequencing[J]. Food Science, 2018, 39(24): 149-155. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-201824023. http://www.spkx.net.cn
邓晓影, 张宾, 汤贺, 等. 基于高通量测序的南美白对虾中微生物群落分析[J]. 食品科学, 2018, 39(24): 149-155.DOI:10.7506/spkx1002-6630-2018024023. http://www.spkx.net.cn
引文格式:
文章编号:1002-6630(2018)24-0149-07
文献标志码:A
中图分类号:TS254.4
DOI:10.7506/spkx1002-6630-201824023
*通信作者简介:张宾(1981—),男,副教授,博士,研究方向为水产品加工及贮藏。E-mail:zhangbin_ouc@163.com
第一作者简介:邓晓影(1992—),女,硕士研究生,研究方向为水产品加工及贮藏。E-mail:1312790830@qq.com
基金项目:中国博士后科学基金项目(2016M590663;2017T100518);浙江省公益性项目(2016C32081);浙江省自然科学基金项目(LY18C200008)
收稿日期:2017-06-30