应用多重PCR快速鉴定自然发酵肉制品中6 种葡萄球菌
许随根,陈 曦*,李家鹏,李金春,杨君娜,熊苏玥,戚 彪,米瑞芳,黄 鑫,乔晓玲,王守伟*
(中国肉类食品综合研究中心,北京食品科学研究院,国家肉类加工工程技术研究中心,肉类加工技术北京市重点实验室,北京 100068)
摘 要:肉葡萄球菌(Staphylococcus carnosus)、木糖葡萄球菌(S. xylosus)、腐生葡萄球菌(S. saprophyticus)、表皮葡萄球菌(S. epidermidis)、马胃葡萄球菌(S. equorum)和松鼠葡萄球菌(S. sciuri)是发酵肉制品中常见的葡萄球菌,为准确、快速地检测和鉴定这些菌种,建立6 种葡萄球菌的多重聚合酶链式反应(polymerase chain reaction,PCR)方法并进行验证。以上述菌种的gap、rpoB、SodA和SNc为靶基因,设计和筛选菌种特异性引物6 对,优化PCR体系,建立6 种葡萄球菌的多重PCR鉴定方法。实验结果显示多重PCR的基因组DNA检测灵敏度可达到4 pg,活菌检测灵敏度可达到2×102CFU;采用建立的多重PCR方法对传统肉制品中分离的8 株葡萄球菌进行鉴定,结果与16S rDNA序列分析、生理生化鉴定结果一致。建立的多重PCR方法具有较高的种间特异性,可快速、准确地用于发酵肉制品中肉葡萄球菌、木糖葡萄球菌、表皮葡萄球菌、马胃葡萄球菌、松鼠葡萄球菌和腐生葡萄球菌的检测和鉴定,具有较好的应用前景。
关键词:多重PCR;葡萄球菌;发酵肉制品;快速鉴定
在自然发酵肉制品中,木糖葡萄球菌、肉葡萄球菌、腐生葡萄球菌、表皮葡萄球菌、马胃葡萄球菌和松鼠葡萄球菌通常是占据优势地位的葡萄球菌菌种[1-8],它们在发酵肉制品生产过程中能够产生脂肪酶和蛋白酶,对发酵肉制品风味品质的形成起着重要作用[9],同时还能产生硝酸还原酶和一氧化氮合酶影响发酵肉制品色泽[10],同时还产生过氧化氢酶具有抗氧化活性[11],其中,木糖葡萄球菌和肉葡萄球菌是目前商业肉品发酵剂所用菌种的重要来源[12]。快速准确地识别和鉴定自然发酵肉制品中的葡萄球菌,可大大提高优良肉品发酵剂葡萄球菌菌株的筛选鉴定效率,也有利于建立基于葡萄球菌菌群变化的发酵肉制品质量监控技术。
葡萄球菌属的许多种其菌落形态、菌体特征相似难辨,生理生化特性也存在许多相似之处。传统的生理生化实验耗时费力,Vitek 2鉴定系统、脂肪酸分析等则成本较高,不能满足发酵肉制品葡萄球菌检测中的快速、准确、低成本的需要[13]。目前,许多非培养方式鉴定技术被用于葡萄球菌的鉴定,包括种特异性聚合酶链式反应(polymerase chain reaction,PCR)、16S rDNA测序、聚合酶链式反应-变性梯度凝胶电泳(polymerase chain reaction-denaturing gradient gel electrophoresis,PCR-DGGE)[14-15]、随机扩增多态性DNA标记(random amplified polymorphic DNA,RAPD)、聚合酶链式反应-限制性片段长度多态性技术(polymerase chain reactionrestriction fragment length polymorphism,PCR-RFLP)等[16-17]、基质辅助激光解析电离飞行时间质谱(matrixassisted laser desorption ionization time-of-flight mass spectrometry,MALDI-TOF MS)[18-19]。在众多非培养方式鉴定方法中,特异性PCR技术以其快速、准确、简单及低成本等优点备受青睐,已得到了广泛应用[20]。至今,马胃葡萄球菌、木糖葡萄球菌、腐生葡萄球菌、表皮葡萄球菌、肉葡萄球菌的单菌种特异性PCR检测方法已经先后被建立[21-26]。
本研究以发酵肉制品中常见的肉葡萄球菌、木糖葡萄球菌、腐生葡萄球菌、表皮葡萄球菌、马胃葡萄球菌和松鼠葡萄球菌为研究对象,设计和筛选菌种特异性引物,优化PCR体系,建立6 种葡萄球菌的多重PCR鉴定方法,应用于发酵肉制品微生物检测过程中的葡萄球菌快速识别与鉴定。
1 材料与方法
1.1 材料、试剂与仪器
1.1.1 菌株
建立多重PCR方法体系所用菌株:1 株为本实验室筛选,保藏于中国普通微生物菌种保藏管理中心;2 株购自丹麦科汉森公司,17 株购自中国工业微生物菌种保藏管理中心(CICC),详见表1。多重PCR方法体系验证所用菌株:本实验室从传统肉制品中分离得到,并采用Vitek 2微生物生理生化鉴定仪对菌株进行种属鉴定。
表1 实验菌株
Table 1 Bacterial strains used in this study

菌株名称 菌株编号 菌株来源模仿葡萄球菌(Staphylococcus simulans) CICC24064 购买于中国工业微生物菌种保藏管理中心缓慢葡萄球菌(S. lentus) CICC10897 购买于中国工业微生物菌种保藏管理中心金黄色葡萄球菌(S. aureus) CICC10384 购买于中国工业微生物菌种保藏管理中心腐生葡萄球菌(S. saprophyticus) CICC22941 购买于中国工业微生物菌种保藏管理中心肉葡萄球菌(S. carnosus) CMRC SC4 购买于丹麦科汉森股份有限公司马胃葡萄球菌(S. equorum) CGMCC11349 保藏于中国普通微生物菌种保藏管理中心表皮葡萄球菌(S. epidermidis) CICC10294 购买于中国工业微生物菌种保藏管理中心松鼠葡萄球菌(S. sciuri) CICC23471 购买于中国工业微生物菌种保藏管理中心木糖葡萄球菌(S. xylosus) CMRC SX5 购买于丹麦科汉森股份有限公司沃氏葡萄球菌(S. warneri) CICC23992 购买于中国工业微生物菌种保藏管理中心人葡萄球菌(S. hominis) CICC23976 购买于中国工业微生物菌种保藏管理中心头葡萄球菌(S. capitis) CICC21722 购买于中国工业微生物菌种保藏管理中心小牛肉葡萄球菌(S. vitulinus) CICC10850 购买于中国工业微生物菌种保藏管理中心玫瑰色库克菌(Kocuria roseus) CICC10190 购买于中国工业微生物菌种保藏管理中心变异库克菌(K. varians) CICC21123 购买于中国工业微生物菌种保藏管理中心藤黄微球菌(Micrococcus luteus) CICC10680 购买于中国工业微生物菌种保藏管理中心弯曲乳杆菌(Lactobacillus paracasei) CICC21019 购买于中国工业微生物菌种保藏管理中心植物乳杆菌(L. plantarum) CICC6240 购买于中国工业微生物菌种保藏管理中心粪肠球菌(Enterococcus faecalis) CICC10396 购买于中国工业微生物菌种保藏管理中心屎肠球菌(E. faecium) CICC21605 购买于中国工业微生物菌种保藏管理中心
1.1.2 试剂
营养肉汤培养基、甘露醇氯化钠琼脂培养基(MSA)肉汤(MRS)、胰蛋白胨大豆肉汤、脑心浸液肉汤 北京陆桥技术股份有限公司;细菌基因组DNA提取试剂盒 北京天根生物技术有限公司;Premix TaqTM酶混合液 大连宝生物工程有限公司;SYBR Green I染料 北京康普生物技术有限公司;无水乙醇 北京化工集团;MCE-202 MultiNA微芯片电泳试剂盒 岛津企业管理(中国)股份有限公司。
1.1.3 仪器与设备
PB-10 pH计 德国赛多利斯公司;GI54DWS全自动高压灭菌器 美国致微(厦门)仪器有限公司;ESCO二级生物安全柜 新加坡艺思高科技有限公司;T100TMThermal Cycler型PCR仪美国伯乐公司;5417R冷冻离心机 德国艾本德公司;B-260恒温水浴锅 上海亚荣生化仪器厂;MCE-202 MultiNA微芯片电泳系统 岛津企业管理(中国)股份有限公司;Vitek 2 Compact全自动微生物鉴定分析系统法国生物梅里埃公司。
1.2 方法
1.2.1 实验菌株的培养
将表1中实验菌株分别接种于100 mL培养基中,37 ℃振荡培养。其中腐生葡萄球菌、肉葡萄球菌、马胃葡萄球菌、表皮葡萄球菌、松鼠葡萄球菌、木糖葡萄球菌分别取培养2、4、6、8、12、24、48 h菌液,用无菌生理盐水10 倍系列稀释后,合适的稀释度取1 mL倾注于MSA平板,37 ℃培养48 h后计数。
1.2.2 细菌基因组DNA的制备
根据细菌基因组DNA试剂盒说明书进行。
1.2.3 特异性引物设计
选择表1中菌的rpoB、gap、hsp60、tuf、sodA基因作为目的基因,用BioEdit比较其gap、rpoB、hsp60、tuf、sodA基因的基因序列,之后用Oligo7软件设计3 对特异性引物,引物确定后,使用BLAST程序通过GenBank对各引物及扩增产物同源性进行比对,最终确定利用肉葡萄球菌的gap基因、腐生葡萄球菌和木糖葡萄球菌的rpoB基因设计菌种特异性引物。马胃葡萄球菌、表皮葡萄球菌、松鼠葡萄球菌则利用已经报道的引物,所有引物由上海英骏生物技术有限公司合成(表2)。
表2 用于多重PCR的特异性引物
Table 2 Specific primer sequences for multiplex PCR

菌种 靶基因 引物名称 引物序列(5′→3′) 产物长度/bp腐生葡萄球菌 rpoB Ssa-113-F Ssa-113-R AAAGTGGATCTTGAAACGAATTCA CGGCCATTTTCGTCTAATGTT 113肉葡萄球菌 gap Sc-157-F Sc-157-R AATGTACAGGTTTCTATACAAGTGA GAAACGATTACGTCAGAGCT 157马胃葡萄球菌 SodA Seq-193-F Seq-193-R GTGGAGGACACTTAAACCATTC CAATTTACCATCGTTTACAACTAG 193[27]表皮葡萄球菌 SNc Se-251-F Se-251-R TTGTAAACCATTCTGGACCG ATGCGTGAGATACTTCTTCG 251[28]松鼠葡萄球菌 gap Ssc-306-F Ssc-306-R GATTCCGCGTAAACGGTAGAG CATCATTTAATACTTTAGCCATTG 306[29]木糖葡萄球菌 rpoB Sx-453-F Sx-453-R AGACCTTGAAACAAACAGCATT TGGCCATCTTCTTCAATAAGTT 453
1.2.4 引物的特异性检测
提取表1中实验菌株的基因组DNA作为模板,然后进行PCR扩增。针对引物添加量、退火温度、循环数等进行优化,确定最适反应体系。采用25 μL的PCR体系,其中Premix Taq™酶混合液12.5 μL,上下游引物(10 μmol/L)各0.5 μL,模板DNA 2 μL,加无菌重蒸水补足至25 μL。PCR扩增程序:95 ℃预变性5 min;95 ℃变性15 s,60 ℃退火10 s,72 ℃延伸30 s,进行32 个循环;循环结束后72 ℃延伸10 min,冷却到4 ℃,产物4 ℃保存备用。取10 μL扩增产物进行微芯片电泳检测。
1.2.5 多重PCR体系建立
将表2的6 对引物进行混合,以各目的菌株的DNA作为模板检测多重PCR特异性,针对引物添加量、退火温度、循环数进行优化,根据文献报道各引物对在多重PCR过程中呈相互抑制的关系,适当的调节引物浓度配比可以有效避免目标条带漏检的现象,是影响多重PCR有效扩增目标条带的关键因素[30],首先设计不同引物浓度梯度的组合体系(表3中组合体系1~5),之后根据各引物所产生的条带亮度调整不同菌种引物浓度(表3中组合体系6~21),以扩增出均一、清晰、明亮且特异性良好的目的条带为标准,最终确定多重PCR体系为50 μL,包括Premix Taq™酶混合液25 μL,引物F(10 μmol/L)、引物R(10 μmol/L)各6.1 μL(引物F、引物R为6 种菌特异性引物按比例混合组成),每种菌模板DNA各1.5 μL,加无菌重蒸水补足至50 μL。PCR扩增程序:95 ℃预变性5 min;95 ℃变性15 s,60 ℃退火10 s,72 ℃延伸1 min,进行32 个循环;最后72 ℃延伸10 min,4 ℃保存。产物经微芯片电泳检测。
表3 引物添加量及组合体系
Table 3 Primer addition and combination systems

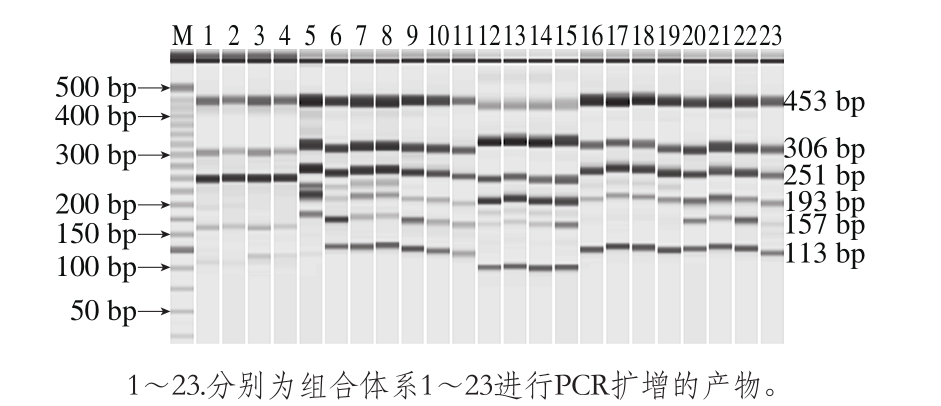
引物名称 组合体系中引物添加量/μL 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 23 Sx-453-F/R 0.20.2 0.3 0.31.50.50.81.00.80.80.40.20.20.20.2 1.0 1.51.20.60.60.80.80.6 Ssc-306-F/R 0.20.20.20.2 1.5 1.01.51.51.01.01.01.51.51.51.50.50.5 0.5 0.51.01.21.21.0 Se-251-F/R 1.01.01.01.01.51.0 1.2 1.21.01.01.00.80.80.80.81.01.01.01.0 1.0 1.01.00.5 Seq-193-F/R 0.20.20.20.21.50.51.51.5 0.8 0.80.81.51.51.51.50.80.81.01.01.51.5 1.0 1.0 Sc-157-F/R 0.50.50.50.51.51.50.50.51.20.50.50.5 0.5 0.5 1.00.20.20.20.21.01.01.20.5 Ssa-113-F/R 0.20.2 0.4 0.41.51.01.01.01.01.00.50.80.81.01.01.01.01.01.01.01.01.51.5模板 各2 各1 各2 各1 各1 各2 各2各1.0各2各1.5各1 各2 各1 各1 各2 各2 各1 各2 各1各1.5各1.5各1.5各1.5水 8.414.47.813.8 1 2 0 5.61.45.810.62.48.4 8 2 2 9 3.210.43.8 3 2.65.8
1.2.6 多重PCR特异性检测
采用优化的多重PCR体系,分别以腐生葡萄球菌、肉葡萄球菌、马胃葡萄球菌、表皮葡萄球菌、松鼠葡萄球菌、木糖葡萄球菌的DNA以及6 种菌的DNA混合体为模板进行多重PCR特异性检测实验。
1.2.7 多重PCR灵敏度检测
将6 种菌分别培养至对数生长期,用无菌生理盐水进行10 倍系列稀释后,平板计数;同时提取各稀释度的菌体DNA,利用多重PCR方法进行检测。
提取6 种菌对数生长期的菌体DNA,检测浓度后分别进行10 倍系列梯度稀释,以各稀释度DNA作为模板进行多重PCR检测。
1.2.8 多重PCR准确性验证
采用建立的多重PCR方法对传统肉制品中分离的8 株葡萄球菌进行鉴定,其结果与8 株菌利用16S rDNA序列分析、生理生化鉴定结果对比,验证本实验建立的鉴定方法的准确性。
1.2.9 PCR产物的微芯片电泳检测
本实验使用MCE-202 MultiNA 微芯片电泳系统,具体操作参照其使用说明书。
1.3 数据分析及图像处理
利用Excel 2010、SPSS19软件进行数据处理和分析;微芯片电泳图像利用MultiNA viewer软件进行图像处理。
2 结果与分析
2.1 引物的特异性检测结果
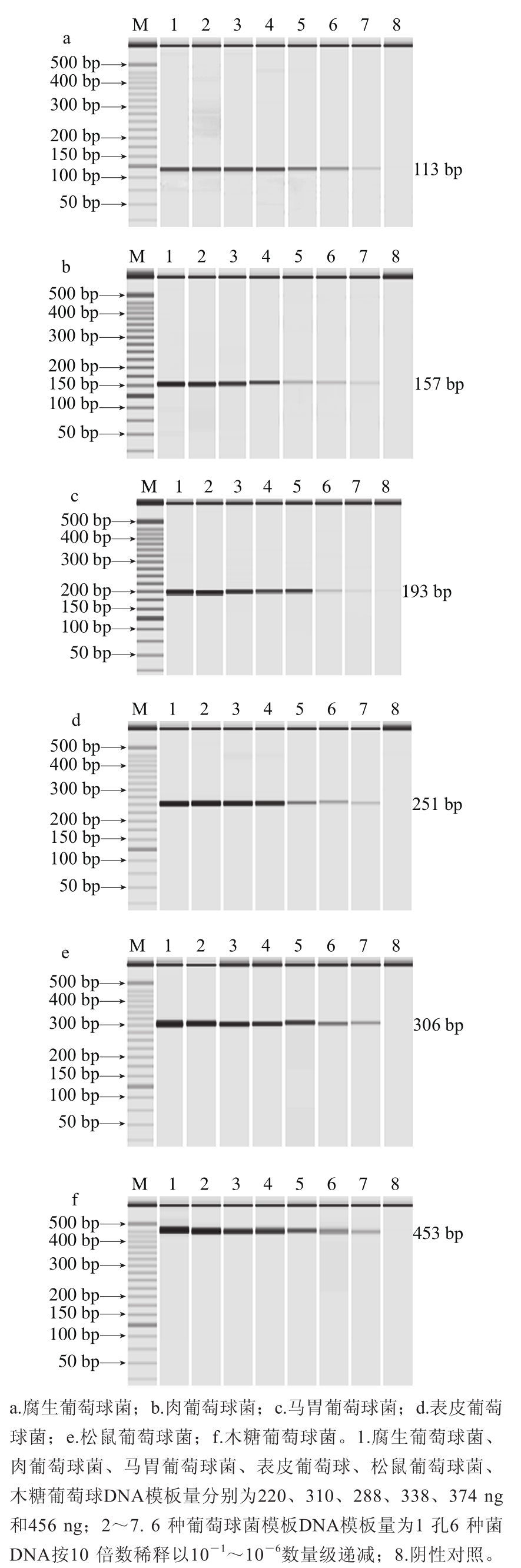
分别利用菌种特异性引物对表1中菌株进行PCR检测,结果如图1所示。所有目标菌株均产生预期条带,且没有非特异性条带出现,非目标菌株则都没有扩增出任何条带。表明实验所用的6 对引物具有很好的特异性,能够应用于这6 种葡萄球菌的种属鉴定。
2.2 多重PCR体系建立
多重PCR扩增结果受dNTP浓度、引物、Mg2+浓度、Taq聚合酶、扩增条件等诸多因素影响[31]。根据文献与聚合酶预混液试剂盒说明书,首先确定多重PCR体系总体积、Taq聚合酶预混液添加量和模板DNA添加量。针对引物添加量进行优化,多重PCR引物浓度优化结果如图2所示,不同比例的引物用量扩增的结果有所不同,引物用量较低时,会出现目标条带未被扩增的现象。而当引物用量过高时,会导致单一目标条带过于明亮且弥散。当6 种目标菌的引物浓度配比差异过大,易出现杂带扩增的现象,图2中1~4泳道,由于腐生葡萄球菌、马胃葡萄球菌、松鼠葡萄球菌、木糖葡萄球菌引物浓度含量较低(4 种引物用量均低于0.4 μL),因此,4 种菌的相应条带较暗或者未被扩增,16~19泳道,由于肉葡萄球菌引物浓度过低(引物用量0.2 μL),出现其目标条带未被扩增现象,5泳道,当6 种菌引物用量过高(6 种菌引物用量1.5 μL),又导致目标条带过于明亮且弥散,同时出现非特异性扩增,6~8和12~15泳道,均有杂带扩增现象,20~22泳道,均可扩增出6 条清晰、明亮且特异性良好的条带。考虑到条带明亮度的均一性及最大限度地提高扩增效率,最终,确定多重PCR体系中,木糖葡萄球菌、松鼠葡萄球菌、表皮葡萄球菌、马胃葡萄球菌、肉葡萄球菌和腐生葡萄球菌的引物添加量分别为0.6、1、1、1.5、1、1 μL(引物组合体系20)。
2.3 多重PCR特异性检测结果
利用上述建立的多重PCR体系,分别以腐生葡萄球菌、肉葡萄球菌、马胃葡萄球菌、表皮葡萄球菌、松鼠葡萄球菌、木糖葡萄球菌DNA作为模板,进行多重PCR特异性验证。如图3所示,目标菌株均扩增出预期大小的条带,无非特异性扩增条带出现,阴性对照未扩增出任何条带,表明该体系可同时扩增出6 种葡萄球菌,引物间未发生交叉错配现象,结果表明建立的多重PCR体系具有很好的种间特异性。因此,可利用该方法检测自然发酵肉制品中葡萄球菌群落,或对已分离纯化的葡萄球菌菌株进行快速种属鉴定。
2.4 多重PCR的灵敏度检测结果
2.4.1 多重PCR检测单菌种的菌落数检出限
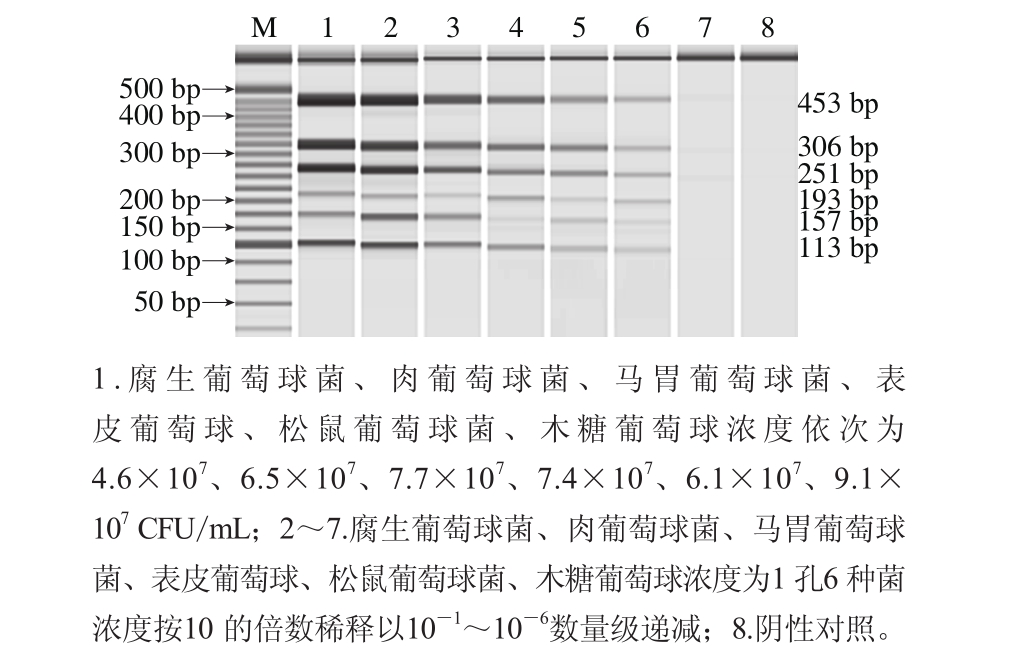
利用平板计数方法,确定腐生葡萄球菌、肉葡萄球菌、马胃葡萄球菌、表皮葡萄球菌、松鼠葡萄球菌、木糖葡萄球菌的菌液初始浓度分别为4.6×107、6.5×107、7.7×107、7.4×107、6.1×107、9.1×107CFU/mL。每种葡萄球菌的培养液经10 倍梯度稀释后,提取DNA进行多重PCR,结果如图4所示,腐生葡萄球菌、肉葡萄球菌、马胃葡萄球菌、表皮葡萄球菌、松鼠葡萄球菌、木糖葡萄球菌的检出限分别为4.6×101、6.5×102、7.7×101、7.4×101、6.1×101、9.1×101CFU/mL。目前肉制品中这6 种葡萄球菌检出限相关报道较少。Shome等[29]应用多重PCR方法检测了牛奶中的松鼠葡萄球菌和表皮葡萄球菌,检出限分别为103CFU/mL和102CFU/mL,与之相比本实验多重PCR方法的灵敏度更好;姜彦君等32]报道了食品中3 种致病菌的多重PCR鉴定方法,其中金黄色葡萄球菌检出限为101CFU/mL,与本实验结果一致。
2.4.2 多重PCR检测单菌种的DNA含量检出限
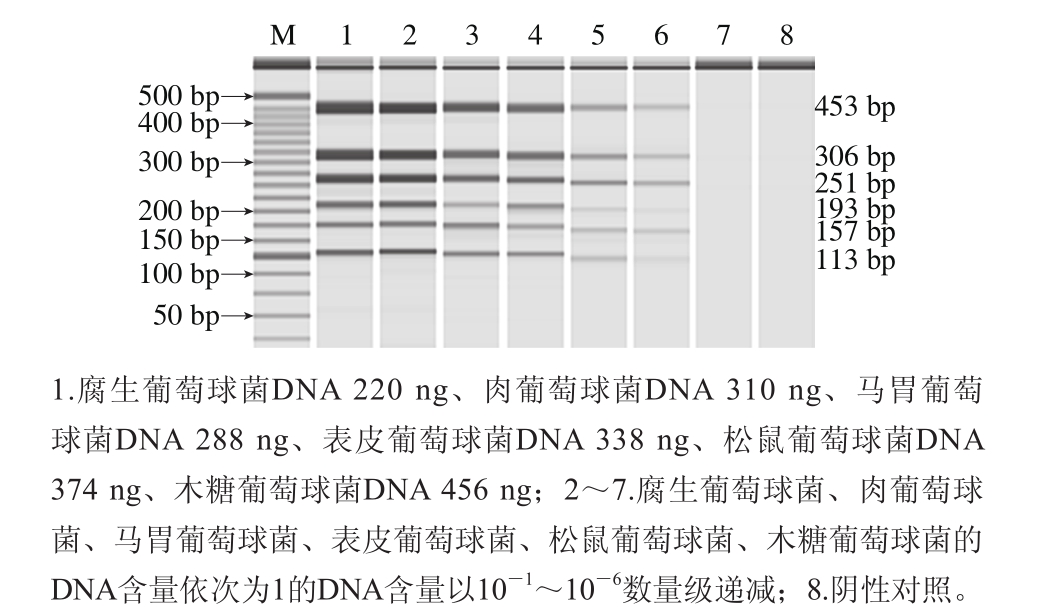
测定腐生葡萄球菌、肉葡萄球菌、马胃葡萄球菌、表皮葡萄球菌、松鼠葡萄球菌、木糖葡萄球菌的DNA的初始质量浓度分别为110、155、144、169、187、228 μg/mL,10 倍系列梯度稀释后,以各稀释度DNA作为模板进行PCR检测。图5各图分别显示其1~7泳道都扩增出目的条带,阴性对照均未扩增出条带,分析结果可以知道腐生葡萄球菌、肉葡萄球菌、马胃葡萄球菌、表皮葡萄球菌、松鼠葡萄球菌、木糖葡萄球菌的DNA的多重PCR检出限分别为0.22、0.31、0.288、0.338、0.374、0.456 pg。
2.4.3 多重PCR检测多菌种的菌落数检出限
分别提取6 种菌的各稀释度菌体DNA,并将同一稀释度的6 种菌体DNA同比例混合后作为模板进行多重PCR,扩增产物利用微芯片电泳进行检测。如图6所示,腐生葡萄球菌、肉葡萄球菌、马胃葡萄球菌、表皮葡萄球菌、松鼠葡萄球菌、木糖葡萄球菌的多重PCR检出限分别为4.6×102、6.5×102、7.7×102、7.4×102、6.1×102、9.1×102CFU/mL。检测结果表明实验建立的多重PCR方法具有较好的灵敏度。刘红玉等[33]报道了多重PCR同时检测肉中4 种致病菌,检出限与本实验结果一致。
2.4.4 多重PCR检测多菌种的DNA含量检出限
将6 种葡萄球菌的DNA进行等量混合,10 倍系列梯度稀释后进行多重PCR检测,如图7所示,腐生葡萄球菌、肉葡萄球菌、马胃葡萄球菌、表皮葡萄球菌、松鼠葡萄球菌、木糖葡萄球菌的DNA含量检出限分别为2.2、3.1、2.88、3.38、3.74、4.56 pg。钱志伟等[34]建立了乳制品中3 种致病菌多重PCR检测体系,金黄色葡萄球菌检出限为3.8 pg;胡冰雪等[35]建立肉制品中荧光假单胞菌、沙门菌和单核细胞性李斯特菌的多重PCR检测方法,DNA检测限为1 pg,与本实验结果一致。
2.5 多重PCR准确性验证
从贵州地区采集了3 种不同工艺的自然发酵酸肉,提取了酸肉微生物群落总DNA。利用本实验建立的多重PCR鉴定方法,从酸肉微生物群落中检测到了这6 种葡萄球菌。为进一步验证多重PCR检测的准确性,利用传统分离培养方法,从酸肉中分离纯化了若干葡萄球菌,其中8 株葡萄球菌经过16S rDNA序列分析、生理生化鉴定为松鼠葡萄球菌、马胃葡萄球菌、木糖葡萄球菌和腐生葡萄球菌。同时利用多重PCR对这8 株菌进行鉴定,结果见表4,可知不同方法对8 株菌的鉴定结果一致,因此,本实验建立的多重PCR方法具有较高的种间特异性,可快速、准确地用于发酵肉制品中肉葡萄球菌、木糖葡萄球菌、表皮葡萄球菌、马胃葡萄球菌、松鼠葡萄球菌和腐生葡萄球菌的检测和鉴定,具有较好的应用前景。
表4 两种鉴定方法检测结果对照
Table 4 Comparison of multiplex PCR and automated microbial Identification system for the detection of Staphylococcus
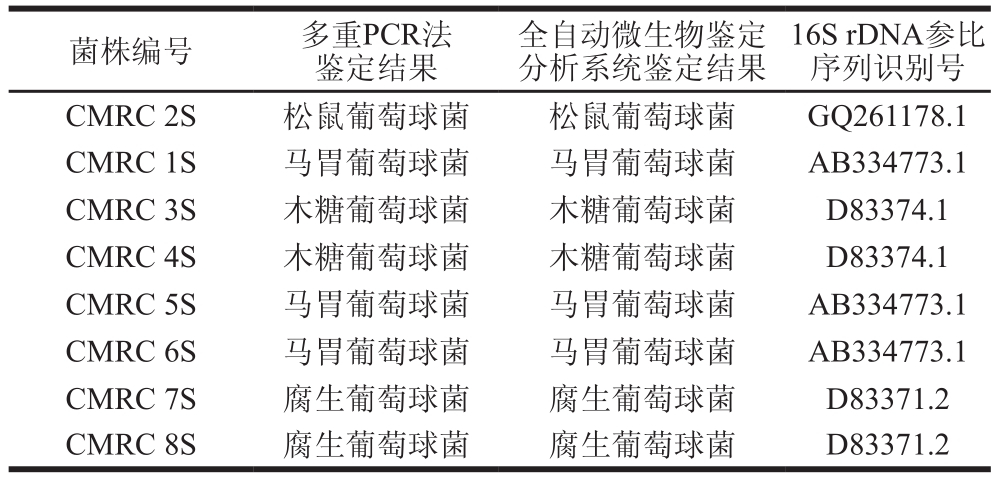
16S rDNA参比序列识别号CMRC 2S 松鼠葡萄球菌 松鼠葡萄球菌 GQ261178.1 CMRC 1S 马胃葡萄球菌 马胃葡萄球菌 AB334773.1 CMRC 3S 木糖葡萄球菌 木糖葡萄球菌 D83374.1 CMRC 4S 木糖葡萄球菌 木糖葡萄球菌 D83374.1 CMRC 5S 马胃葡萄球菌 马胃葡萄球菌 AB334773.1 CMRC 6S 马胃葡萄球菌 马胃葡萄球菌 AB334773.1 CMRC 7S 腐生葡萄球菌 腐生葡萄球菌 D83371.2 CMRC 8S 腐生葡萄球菌 腐生葡萄球菌 D83371.2菌株编号 多重PCR法鉴定结果全自动微生物鉴定分析系统鉴定结果
3 结 论
本实验设计、筛选6 种凝固酶阴性葡萄球菌的特异性引物,通过优化引物浓度、PCR扩增体系、扩增条件、检测系统等建立特异性好且稳定的多重PCR快速鉴定葡萄球菌的体系,此体系将传统方法中大量的生化试验转化为一次性PCR扩增,具有操作简便、快速、灵敏度高等特点,便于批量检测,而且相对传统鉴定方法检测成本大大降低。PCR产物利用微芯片电泳检测系统,此检测系统灵敏度高、检测时间短[36],相比传统电泳系统大大缩短检测时间。本实验建立的多重PCR体系检出限达到2×102CFU/mL,DNA含量检出限达到4 pg,具有较好的灵敏度和特异性,利用实验建立的多重PCR鉴定体系3 h就可以判定出结果,相比传统的生理生化实验省时省力,多重PCR法相比Vitek 2鉴定系统、脂肪酸分析等则成本较低,能够满足不具备Vitek 2鉴定系统科研单位和科研工作者的鉴定需求,具有较好的应用前景。
参考文献:
[1] 龙强, 聂乾忠, 刘成国. 发酵肉制品功能性发酵剂研究现状[J].食品科学, 2016, 37(17): 263-269. DOI:10.7506/spkx1002-6630-201617044.
[2] 夏秀芳, 张金铎, 孔保华. 木糖葡萄球菌添加量对发酵牛肉串品质特性的影响[J]. 食品科学, 2013, 34(7): 47-50. DOI:10.7506/spkx1002-6630-201307011.
[3] 张晓琼, 桂萌, 靳晓娇, 等. 不同发酵肉中优良葡萄球菌的筛选鉴定[J].肉类研究, 2011, 25(8): 17-21. DOI:10.3969/j.issn.1001-8123.2011.08.004.
[4] COTON E, DESMONTS M-H, LEROY S, et al. Biodiversity of coagulase-negative staphylococci in French cheeses, dry fermented sausages, processing environments and clinical samples[J].International Journal of Food Microbiology, 2010, 137(1/2/3): 221-229. DOI:10.1016/j.ijfoodmicro.2009.11.023.
[5] 孙二娜, 陈历水, 刘松玲, 等. 传统发酵肉制品中葡萄球菌的分离筛选及鉴定[C]//第9届乳酸菌与健康国际研讨会. 天津: 中国食品科学技术学会, 2014: 80.
[6] SEMEDO-LEMSADDEK T, CARVALHO L, TEMPERA C, et al.Characterization and technological features of autochthonous coagulase-negative staphylococci as potential starters for portuguese dry fermented sausages[J]. Journal of Food Science, 2016, 81(5):M1197-M1202. DOI:10.1111/1750-3841.13311.
[7] 马宇霞, 卢士玲, 李开雄, 等. 熏马肠中生物胺氧化酶菌株的筛选与鉴定[J]. 现代食品科技, 2014, 30(5): 49-55.
[8] LANDETA G, CURIEL J A, CARRASCOSA A V, et al.Characterization of coagulase-negative staphylococci isolated from Spanish dry cured meat products[J]. Meat Science, 2013, 93(3):387-396. DOI:10.1016/j.meatsci.2012.09.019.
[9] SEITTER M. Safety assessment of coagulase-negative staphylococci used in food production[D]. Stuttgart: Universität Hohenheim, 2015.
[10] MAINAR M S, LEROY F. Process-driven bacterial community dynamics are key to cured meat colour formation by coagulasenegative staphylococci via nitrate reductase ornitric oxide synthase activities[J]. International Journal of Food Microbiology, 2015, 212(6):60-66. DOI:10.1016/j.ijfoodmicro.2015.03.009.
[11] SANCHEZ M M, STAVROPOULOU D A, LEROY F. Exploring the metabolic heterogeneity of coagulase-negative staphylococci to improve the quality and safety of fermented meats: a review[J].International Journal of Food Microbiology, 2017, 247(1): 24-37.DOI:10.1016/j.ijfoodmicro.2016.05.021.
[12] MARTY E, BODENMANN C, BUCHS J, et al. Prevalence of antibiotic resistance in coagulase-negative staphylococci from spontaneously fermented meat products and safety assessment for new starters[J]. International Journal of Food Microbiology, 2012, 159(2):74-83. DOI:10.1016/j.ijfoodmicro.2012.07.025.
[13] RANTSIOU K, COCOLIN L. New developments in the study of the microbiota of naturally fermented sausages as determined by molecular methods: a review[J]. International Journal of Food Microbiology,2006, 108(2): 255-267. DOI:10.1016/j.ijfoodmicro.2005.11.013.
[14] COCOLIN L, MANZANO M, CANTONI C, et al. Denaturing gradient gel electrophoresis analysis of the 16S RNA gene v1 region to monitor dynamic changes in the bacterial population during fermentation of italian sausages[J]. Applied and Environmental Microbiology, 2001,67(11): 5113-5121. DOI:10.1128/AEM.67.11.5113-5121.2001.
[15] FONTANA C, VIGNOLO G, COCCONCELLI P. PCR-DGGE analysis for the identification of microbial populations from Argentinean dry fermented sausages[J]. Journal of Microbiological Methods, 2005, 63(3): 254-263. DOI:10.1016/j.mimet.2005.03.010.
[16] MARTY E, BUCHS J, EUGSTER-MEIER E, et al. Identification of staphylococci and dominant lactic acid bacteria in spontaneously fermented Swiss meat products using PCR-RFLP[J]. Food Microbiology, 2012, 29(2): 157-166. DOI:10.1016/j.fm.2011.09.011.
[17] SPIEGELMAN D, WHISSELL G, GREER C W. A survey of the methods for the characterization of microbial consortia and communities[J]. Canadian Journal of Microbiology, 2005, 51(5): 355-386. DOI:10.1139/w05-003.
[18] HAN H W, CHANG H C, HUNAG A H, et al. Optimization of the score cutoff value for routine identifcation of Staphylococcus species by matrix-assisted laser desorption ionization-timeoffight mass spectrometry[J]. Diagnostic Microbiology &Infectious Disease, 2015, 83(4): 349-354. DOI:10.1016/j.diagmicrobio.2015.08.003.
[19] TOMAZI T, GONCALVES J L, BARREIRO J R. Identifcation of coagulase-negative staphylococci from bovine intramammary infection by matrix-assisted laser desorption ionization-time of fight mass spectrometry[J]. Journal of Clinical Microbiology, 2014, 52(5): 1658-1663. DOI:10.1128/JCM.03032-13.
[20] 王稳航, 徐倩倩, 滕安国, 等. 发酵肉制品菌群的非培养鉴定技术研究进展[J]. 肉类研究, 2014, 28(9): 17-21.
[21] KIM J, HONG J, LIM J A, et al. Improved multiplex PCR primers for rapid identification of coagulase-negative staphylococci[J]. Archives of Microbiology, 2017, 200(1): 73-83. DOI:10.1007/s00203-017-1415-9.
[22] LANDETA G, REVERON I, CARRASCOSA A V, et al.Use of recA gene sequence analysis for the identification of Staphylococcus equorum strains predominant on dry-cured hams[J].Food Microbiology, 2011, 28(6): 1205-1210. DOI:10.1016/j.fm.2011.04.006.
[23] MOROT-BIZOT S, TALON R, LEROY-SETRINB S. Development of specific PCR primers for a rapid and accurate identification of Staphylococcus xylosus, a species used in food fermentation[J]. Journal of Microbiological Methods, 2003, 55(1): 279-286. DOI:10.1016/S0167-7012(03)00159-3.
[24] MOROT-BIZOT C S, TALON R, LEROY S. Development of a multiplex PCR for the identification of Staphylococcus genus and four staphylococcal species isolated from food[J]. Journal of Applied Microbiology, 2004, 97(5): 1087-1094. DOI:10.1111/j.1365-2672.2004.02399.x.
[25] AYMERICH T, MARTIN B, GARRIGA M, et al. Microbial quality and direct PCR identification of lactic acid bacteria and nonpathogenic staphylococci from artisanal low-acid sausages[J]. Applied and Environmental Microbiology, 2003, 69(8): 4583-4594. DOI:10.1128/AEM.69.8.4583-4594.2003.
[26] GANDRA E A, FERNANDEZ M A, SILVA J A, et al. Detection by multiplex PCR of Staphylococcus aureus, S. intermedius and S. hyicus in artificially contaminated milk[J]. Ciencia Rural, 2016 , 46(1): 1418-1423. DOI:10.1590/0103-8478cr20151391.
[27] BLAIOTTA G, ERCOLINI D, MAURIELLO G, et al. Rapid and reliable identification of Staphylococcus equorum by a species specific PCR assay targeting the sodA gene[J].Systematic & Applied Microbiology, 2004, 27(6): 696-702.DOI:10.1078/0723202042369901.
[28] HIROTAKI S, SASAKI T, KUWAHARA-ARAI K, et al. Rapid and accurate identification of human-associated Staphylococci by use of multiplex PCR[J]. Journal of Clinical Microbiology, 2011, 49(10):3627-3631. DOI:10.1128/JCM.00488-11.
[29] SHOME B R, MITRAL S D, BHUVANAL M, et al. Multiplex PCR assay for species identification of bovine mastitis pathogens[J]. Journal of Applied Microbiology, 2011, 111(6): 1349-1356. DOI:10.1111/j.1365-2672.2011.05169.x.
[30] 孙鸿燕. 四种食源性致病菌多重PCR检测方法的建立[D]. 长春:吉林大学, 2011: 37.
[31] 周明东, 张臣伟, 张海滨, 等. 食源性致病菌快速检测方法的建立与应用[J]. 中国食品学报, 2012, 12(10): 169-176.
[32] 姜彦君, 都启晶, 范颖华, 等. 食源性致病菌快速检测试剂盒的研制与应用[J]. 中国食品学报, 2014, 14(1): 224-231.
[33] 刘红玉, 李岩, 崔洪斌. 肉中4 种致病菌的PCR快速检测方法的建立[J].食品科学, 2011 , 32(6): 213-216.
[34] 钱志伟, 孙新城. 食品中3 种致病菌多重PCR检测体系的建立及初步应用[J]. 食品科学, 2011, 32(16): 236-239.
[35] 胡冰雪, 舒沿沿, 潘道东, 等. 荧光假单胞菌、沙门氏菌和单增李斯特菌多重PCR检测方法的建立[J]. 食品科学, 2016, 37(20): 209-214.DOI:10.7506/spkx1002-6630-201620036.
[36] 张莎莎, 郭新梅, 赵美爱, 等. 微芯片电泳与聚丙烯酰胺凝胶电泳的比较分析[J]. 中国农业科技导报, 2013, 15(2): 173-178.
Rapid Identification of Six Staphylococcus Species in Fermented Meat Products Using Multiplex
XU Suigen, CHEN Xi*, LI Jiapeng, LI Jinchun, YANG Junna, XIONG Suyue, QI Biao, MI Ruifang,HUANG Xin, QIAO Xiaoling, WANG Shouwei*
(Beijing Key Laboratory of Meat Processing Technology, China Meat Processing and Engineering Center,Beijing Academy of Food Sciences, China Meat Research Center, Beijing 100068, China)
Abstract:A multiplex polymerase chain reaction (mPCR) assay was developed and validated for simultaneous detection of Staphylococcus carnosus, S. xylosus, S. saprophyticus, S. epidermidis, S. equorum and S. sciuri, which are commonly found in fermented meat products. In this study, six pairs of species-specific primers were designed targeting the gap, rpoB, SodA and SNc genes, and the PCR reaction system was optimized. The results showed that The sensitivity of the mPCR system was 4 pg for bacterial genomic DNA and 2 × 102CFU for living cultures. Meanwhile, eight Staphylococcus strains from traditional fermented meat products were consistently identified by this mPCR assay and 16S rDNA sequence and biochemical analysis.This mPCR method was species-specific and effective, and could be used in the rapid and accurate identification of S. carnosus, S.xylosus, S. epidermidis, S. equorum, S. sciuri and S. saprophyticus. Thus, it has great application prospects.
Keywords:multiplex PCR; Staphylococcus; fermented meat products; rapid identification
XU Suigen, CHEN Xi, LI Jiapeng, et al. Rapid identification of six Staphylococcus species in fermented meat products using multiplex[J]. Food Science, 2018, 39(24): 303-310. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-201824045. http://www.spkx.net.cn
许随根, 陈曦, 李家鹏, 等. 应用多重PCR快速鉴定自然发酵肉制品中6 种葡萄球菌[J]. 食品科学, 2018, 39(24): 303-310.DOI:10.7506/spkx1002-6630-201824045. http://www.spkx.net.cn
引文格式:
文章编号:1002-6630(2018)24-0303-08
文献标志码:A
中图分类号:TS201.3
DOI:10.7506/spkx1002-6630-201824045
*通信作者简介:陈曦(1983—),女,高级工程师,博士,研究方向为食品生物技术。E-mail:961983@163.com王守伟(1961—),男,教授级高级工程师,硕士,研究方向为肉类食品科学与食品安全。E-mail:cmrcwsw@126.com
第一作者简介:许随根(1987—),男,工程师,硕士,研究方向为食品生物技术。E-mail:xusuigen@126.com
基金项目:“十三五”国家重点研发计划重点专项(2018YFD0401200);北京市优秀人才项目(2017754154700G098)
收稿日期:2018-08-22