乳酸菌是一类能够发酵碳水化合物并产生乳酸的革兰氏阳性细菌的总称。乳酸菌的发酵作用可以改善产品风味,延长产品的货架期等。因此乳酸菌作为一种重要的工业微生物,广泛应用于食品发酵行业[1]。由于高浓度的盐能够抑制一些导致食品腐败的微生物和一些病原微生物的生长,同时根据产品加工工艺要求,在酱油、熏肉、泡菜等发酵食品制备过程中通常会加入大量的食用盐,既满足了产品的风味要求,又可以延长食品的保质期。但高浓度盐是乳酸菌发酵过程的不利因素,其会改变细胞外界渗透压,引起菌体细胞体积缩小,导致胞内各种生理代谢活动紊乱甚至死亡。因此乳酸菌在高盐环境下的生存能力对发酵食品的品质起着关键作用。为了开发耐盐乳酸菌,或寻找合适的盐胁迫保护物质,需要更深入地了解乳酸菌的耐盐机制。本文对盐胁迫下乳酸菌调节胞内渗透压的机制进行了详细阐释,旨在寻找与乳酸菌盐胁迫适应相关的代谢通路以及相应具有调节作用的保护物质,以期为采用基因工程、发酵工程和酶工程等手段提高盐胁迫环境乳酸菌的存活率提供理论依据,进而为更有效地利用乳酸菌提供参考[2]。
1 国内外乳酸菌耐盐文献总结
在Web of Science分别以Lactobacillus salt tolerance(乳酸菌耐盐)以及salt tolerance(耐盐)为主题进行检索(截止到2017年7月10日),文献数如图1所示。近20 年来,研究者对耐盐相关的研究快速增多,自2011年以来,每年发表论文超过2 000 篇,生物耐盐方向已逐渐成为研究热点;乳酸菌耐盐的报道相对较少,但是相关研究也受到越来越多人的关注,每年发表论文量也呈现递增趋势。目前乳酸菌耐盐研究主要集中在耐盐菌种和保护剂筛选,以及微生物耐盐机制的研究。总体来说,关于乳酸菌耐盐的报道只占细胞耐盐机制研究总体中极少的一部分,而且研究还不够深入,导致对于乳酸菌耐盐机制的了解不够。目前开发耐盐乳酸菌的工作仍以传统的从高盐环境筛选耐盐菌种为主,工作量大而繁琐;而采用基因工程等新技术手段开发耐盐乳酸菌具有效率高、周期短等优点,因此研究乳酸菌的耐盐分子机制具有十分重要的价值。
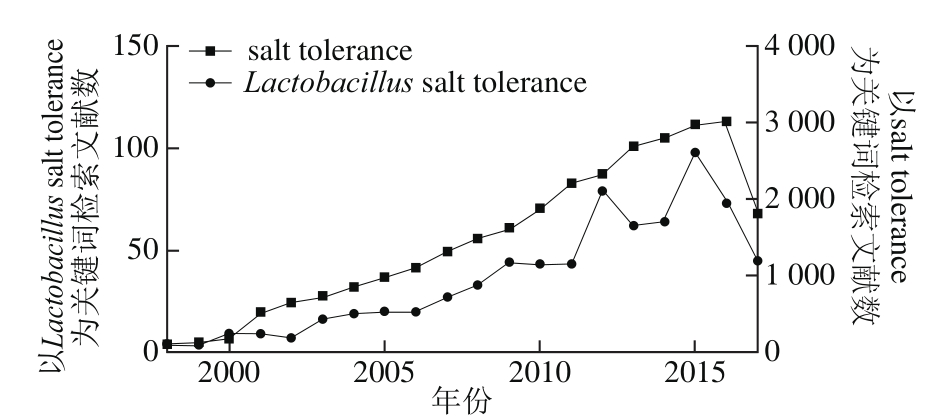
图1 Web of Science分别以Lactobacillus salt tolerance以及salt tolerance为主题检索到的文献数
Fig. 1 Number of publications searched in Web of Science database on Lactobacillus salt tolerance and salt tolerance, respectively
2 乳酸菌耐盐机制
目前研究乳酸菌的耐盐分子机制涉及到细胞内的许多调控系统,现将已报道的各种乳酸菌对应涉及到的各类耐盐调控机制总结如表1所示,并围绕此将国内外研究综述如下。
表1 乳酸菌耐盐机制相关研究
Table 1 Summary of studies regarding salt tolerance mechanism of LAB

注:QacT.季铵盐类化合物转运系统(quaternary ammonium compound transport system);OpuA.渗透保护物质转运系统(osmoprotectant uptake system);Ktr. K+转运系统(K+transport system);Kch. K+通道蛋白(K+chanel);Nha. Na+/H+反向转运蛋白(Na+/H+antiporter)。
2.1 相容性溶质调控系统
自Brown等[22]在1972年首次提出相容性溶质的概念以来,研究者已经发现了许多种类的相容性物质,如甘氨酸甜菜碱、海藻糖、脯氨酸、脯氨酸甜菜碱、四氢嘧啶、肉毒碱等[23-24]。这些溶质通常是水溶性物质,结构类似,存在较多的氢键,能够取代水与蛋白质等胞内生物大分子结合,减少细胞失水从而稳定这些大分子的天然构象[12]。当细胞外渗透压升高时,乳酸菌能够通过积累相容性溶质来维持细胞正常的渗透压;当胞外的渗透压降低时,这些相容性物质又能够迅速地释放出来。尽管大部分乳酸菌会采用吸收相容性物质这种机制来维持细胞的正常渗透压,而且它们所采用的结合底物的能量耦合机制基本相同,但是不同乳酸菌积累的相容性分子类型以及积累机制是不同的[25-28]。已报道的主要涉及植物乳杆菌(Lactobacillus plantarum)、乳酸乳球菌(Lactobacillus lactis)、戊糖片球菌(Pediococcus pentosaceus)等菌株。
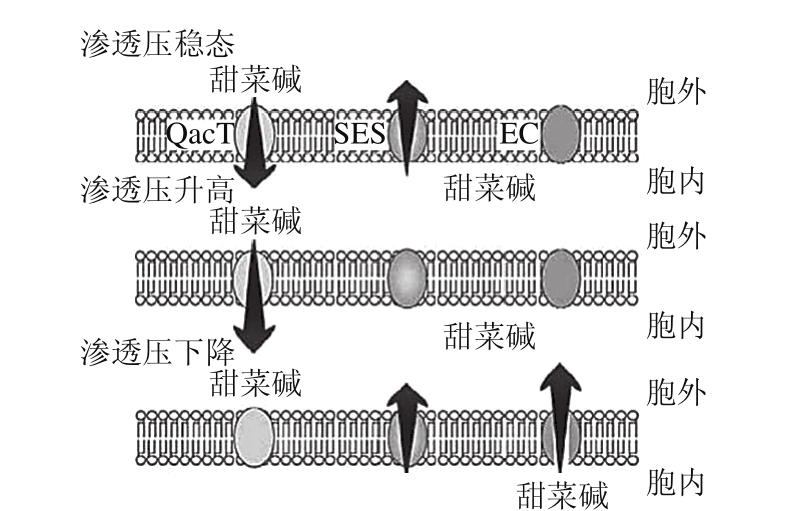
图2 Lactobacillus plantarum的渗透调节模式[27]
Fig. 2 Osmoregulation modes of Lactobacillus plantarum[27]
SES.独立外排系统(separate eラux system);EC.外排载体(eラux carrier)。
在高渗透压条件下,L. plantarum细胞质中积累的主要相容性物质是甘氨酸甜菜碱。L. plantarum自身不能合成或代谢产生甘氨酸甜菜碱,因此细胞对甘氨酸甜菜碱吸收和外排的相对速率决定了其胞内甘氨酸甜菜碱的最终积累水平。甘氨酸甜菜碱的吸收和外排由多个系统介导[3]。QacT是其中最重要的一个转运系统,属于ATP结合盒(ATP binding cassette,ABC)转运子超家族的一员。该系统由ATP驱动,对甘氨酸甜菜碱的亲和性较强,对脯氨酸和其他类似物的亲和性较弱[29]。如图2所示,在正常渗透压状态下,L. plantarum通过QacT摄取和SES排出甘氨酸甜菜碱,二者之间维持在一个平衡状态。当细胞处于高渗透压情况下,甘氨酸甜菜碱摄入量增加,而外排则受到抑制。当细胞外界渗透压低于正常细胞渗透压时,QacT转运系统被抑制,而外排系统被激活,甘氨酸甜菜碱首先通过一个机械力敏感通道,即SES,以较快的速度被排出胞外;接着通过一个单向EC,以缓慢速度外排,用于调整细胞膨压;当渗透压低于一定程度时这些通道被关闭[4,30]。这些与相容性物质运输相关的调控系统在L. plantarum盐胁迫应答中起到了重要作用。
研究表明L. lactis在面临盐胁迫时采用甜菜碱转运系统(betaine uptake system,BusA),即OpuA,运输甘氨酸甜菜碱,维持细胞正常渗透压[31]。BusA作为一个操纵子编码BusA转运蛋白,属于ABC转运系统家族的一员,由2 个亚基组成,其中一个亚基由2 个相同的核苷酸结合位点(nucleotide binding domain,NBD)结合在β-胱硫醚合成酶(beta-cystathionine synthase,CBS)上形成;另外一个亚基由镶嵌在跨膜区域的两个相同的底物结合域组成,两个亚基分别为OpuAA和OpuABC[5,32-33]。Patzlaff等[34]通过酶动力学方法研究发现在OpuA转运系统中,甘氨酸甜菜碱首先结合到底物结合域,然后通过两步ATP与ATP酶亚基的结合反应,实现对甘氨酸甜菜碱的转运吸收。其中一个ATP水解释放能量将OpuA从顺式变成反式构象,从而允许底物在细胞质膜内表面释放;第二个ATP用于重置系统,即将OpuA从反式转变回顺式状态,从而构成一个转运循环。当细胞外渗透压变化时,BusA转运甘氨酸甜菜碱的能力被激活,且其转运活力受到细胞质膜两侧的离子强度和膜的物理性质变化的影响。Bouvier等[6]研究表明BusA转运蛋白表达受到一个由busR基因编码的调控子的调节,busR能够与busA启动子的一个序列重复区域结合,抑制busA基因的转录,进而影响细胞对甘氨酸甜菜碱的吸收;同时busR基因能够增强细胞对渗透压变化的敏感性。在细胞面临盐胁迫同时培养基中甘氨酸甜菜碱匮乏的情况下,L. lacits会通过积累BusA转运蛋白在细胞质中迅速积累甘氨酸甜菜碱,但是过多的甘氨酸甜菜碱又会抑制busA基因的转录;当胞外甘氨酸甜菜碱浓度饱和时,busA基因的转录水平迅速提高,使细胞内BusA转运蛋白的合成水平与细胞质内积累的甘氨酸甜菜碱水平相一致;这也说明了busA操纵子能够调节外界渗透压,同时也能够感受外界渗透压的变化。这种调节机制与大肠杆菌中proU操纵子的调节机制类似。研究还发现,细胞外渗透压升高会使busA启动子上的BusR蛋白被取代,缓解BusR蛋白对busA基因转录的抑制,从而增强细胞对甘氨酸甜菜碱的转运能力[7,35-37]。
对P. pentosaceus渗透保护研究只涉及到不同相容性物质保护效果考察,而没有涉及到转运系统调节等方面的深入研究。Baliarda等[8]研究发现,在含有0.8 mol/L NaCl的特定培养基中分别添加甘氨酸甜菜碱、二甲基乙酸噻亭(硫代甜菜碱)、胆碱、脯氨酸或肉毒碱,均能够缓解盐胁迫P. pentosaceus生长的抑制。在细胞转运这5 种相容性物质时,其他4 种化合物和甘氨酸甜菜碱之间能够产生转运竞争,因此推测P. pentosaceus中可能存在一种转运蛋白能够同时转运这5 种物质。研究还发现P. pentosaceus和L. plantarum在特定培养基中的耐盐能力相差不大,且二者在盐胁迫下细胞内积累的相容性溶质种类也相似,不同的是肉毒碱对P. pentosaceus在盐胁迫下生长的影响相对L. plantarum较弱。对P. pentosaceus相容性物质的转运机制有待更深一步的研究。
2.2 糖酵解关键酶调控系统
乳酸菌主要通过相对简单的代谢途径使底物磷酸化产生细胞代谢所需要的能量(ATP):糖酵解(Embden-Meyerhof-Parnas,EMP)途径和磷酸酮解酶(phosphoketolase,PK)途径。对于乳酸杆菌属,根据其不同的代谢发酵方式,可以将其分为3 种类型。Ⅰ型乳酸杆菌是专一性的同型乳酸发酵,通过EMP途径将己糖(如葡萄糖)发酵形成乳酸盐,但不能代谢戊糖和葡萄糖酸盐。Ⅱ型乳酸杆菌是兼性异型乳酸发酵,可通过EMP将己糖转化为乳酸,也可被诱导产生磷酸酮酶,催化戊糖产生乳酸和乙酸。Ⅲ型乳酸杆菌是专一性的异型乳酸发酵,通过PK途径将己糖代谢形成乳酸、乙醇或乙酸和二氧化碳。EMP反应是这些代谢途径中最重要的第一步反应[38]。代谢途径中的关键酶为磷酸果糖激酶(phosphofructokinase,PFK)、果糖二磷酸醛缩酶(fructosediphosphate aldolase,FBA)、磷酸甘油酸激酶(phosphoglyceric kinase,PGK)、3-磷酸甘油醛脱氢酶(glyceraldehyde-3-phosphate dehydrogenase,GAPDH)、乳酸脱氢酶(lactic dehydrogenase,LDH)[9]。另外,乳酸菌从胞外吸收糖分子,进行EMP时,糖类物质必须经过磷酸烯醇式丙酮酸磷酸转移酶系统(phosphoenolpyruvate phosphotransferase system,PTS),被磷酸化后才能被菌体转运到细胞质中参与糖代谢,其中的某些组分在细胞面临渗透胁迫、调节胞内代谢、减少细胞损伤等过程中发挥了重要作用[10,39]。
EMP是乳酸菌生成乳酸的主要途径,基因pfk、fba、pgk、ldh对应所编码的蛋白酶均是乳酸菌EMP途径中的关键酶。有研究表明基因pfk、fba、pgk、ldh均受培养基中NaCl的诱导而表达上调,并且NaCl浓度越高,基因受诱导表达上调越显著。乳酸菌培养基(de Man,Rogosa and Sharpe broth,MRS)中的NaCl造成菌体外部渗透压变大,这会对基因pfk、fba、pgk、ldh编码的蛋白分子结构有一定破坏作用[9]。为保护自身不受破坏,乳酸菌中与EMP相关的一系列关键酶受到高渗透压胁迫的诱导,相应基因表达上调,保证了EMP途径高效进行,以适应不利环境[11]。Prasad等[12]对高渗条件处理后的细胞提取物进行液质分析,与对照组对比发现,细胞内的甘油量较少,而单糖、二糖、三糖等小分子糖的量较多,说明细胞内的许多大分子的还原性末端被甘油基团修饰。在脱水条件下,甘油分子修饰多糖将增加糖类等大分子中可用的羟基数,增强细胞大分子之间(通过氢键)的相互作用进而保护大分子,可提高高渗条件下菌体细胞的稳定性[40]。
2.3 热休克蛋白调控系统
热休克蛋白(heat shock protein,HSP)是细胞中普遍存在的ATP依赖性分子伴侣,在细胞中的蛋白质质量控制中起关键作用。HSP能够防止各种非天然底物蛋白质的聚集或失活,通过自身或在其他ATP依赖性分子伴侣的帮助重新折叠这些底物,修复受损的蛋白质分子,在细胞胁迫应答中扮演了重要角色。在Lactobacillus rhamnosus中应激蛋白GroEL和DnaK可作为分子伴侣,以短暂的非共价方式与蛋白底物结合,阻止蛋白前体折叠,促进体内蛋白正确合成[41-42]。同时GroEL能够作为RNA分子伴侣保护mRNA,防止其被核酸酶降解[43-44];GroEL和DnaK上调后能够保护细胞内蛋白和其他大分子,防止由环境胁迫诱导造成的细胞大分子损伤和降解[13-14]。
在L. lactis中,编码DnaK的结构基因位于orf1-grpE-dnaK操纵子上[16]。DnaK操纵子的启动子区域中存在两个与分子伴侣表达相关的反向重复序列(controling inverted repeat of chaperone expression,CIRCE)调控元件,其中一个CIRCE元件位于GroEL操纵子的启动子区域中[15]。在各种革兰氏阳性细菌和蓝细菌的热休克基因的启动子区域中已经发现CIRCE元件,例如DnaK、GroEL和GroES[45]。研究已经发现,枯草芽孢杆菌的orf39-grpE-dnaK操纵子中的orf39编码阻遏蛋白,结合CIRCE元件,并且负责基因的热休克控制[46-47]。目前尚不清楚阻遏物的DNA结合活性如何随温度的升高而改变。负责CIRCE元件的热应激调控基因HrcA与L. lacits的DnaK操纵子中orf1基因同源[16]。因此,研究者推测在L. lactis中的DnaK,GroEL和GroES的表达会受到由orf1基因编码阻遏物的控制。在乳球菌的盐胁迫应答反应过程中,高渗透压会使细胞质壁分离,水分活度降低,从而导致变性蛋白的积累,进而诱导HSP调控系统来保护细胞内蛋白和其他大分子,防止由盐胁迫造成的细胞损伤[48]。
2.4 胞内离子平衡
高渗透压条件下,细菌内Na+的调节主要依赖于Nha。该蛋白属于次级转运子,位于原核生物的细胞质膜上,能够促进Na+的排出和质子的进入,同时参与维持胞内正常渗透压和酸碱环境。从反向转运蛋白结构上分,Na+转运可以分为2 类系统:一类为由6 个或7 个基因编码的异源低聚体,另一类为单基因编码形成的转运蛋白。NhaA即为大肠杆菌中主要的Na+/H+转运蛋白,由单基因编码,负责Na+和Li+的运输,对于pH值变化极其敏感,每2 个质子可以交换转运2个Na+或Li+,能够提高细胞的高渗透压耐性[49-51]。NhaB是另外一个对细胞生长非常重要的反向转运蛋白,每3 个质子可以交换2 个Na+。NhaC和NhcD是另外2 个单基因产物家族的反向转运蛋白,其初级结构与NhaA和NhaB相差较大,相关生理功能还有待进一步研究[52]。另外有研究发现在海洋红嗜热盐球菌(Rhodothermus marinus)中存在一个新的Na+/H+反向转运蛋白NhaE,位于烟酰胺腺嘌呤二核苷酸(nicotinamide adenine dinucleotide,NADH)-醌氧化还原酶(NADH-quinone oxidoreductase,Nqo)复合物中,是呼吸链的一部分,在Nqo参与呼吸作用时可将胞内的Na+转运至胞外[53]。
当细胞受到盐胁迫时,除了外排Na+,细胞通常还会增加对K+的吸收,恢复Na+/K+比,从而恢复胞内水和离子平衡。K+跨细胞膜运动涉及到维持细胞渗透压和调节细胞pH值,可以激活细胞生理代谢过程中的各种酶类,参与蛋白质合成代谢过程[54]。绿脓杆菌中存在的Ktr依赖于两种K+转运蛋白。一种是由PA5021基因编码的PA5021蛋白,在高盐环境下细胞向外转运Na+和K+过程中起到了重要作用,同时K+的转运可能也与依赖于呼吸作用的Na+转运相关;另一种是由PA1207基因编码的PA1207蛋白,参与细胞对K+的吸收过程[55]。同时,Nakamura等[56]发现溶藻弧菌(Vibrio alginolyticus)中存在Ktr,KtrA是一个结合在细胞质膜外周的一个膜亚基结构单位,KtrB是一个完整的膜亚基结构单位,参与K+的运输。而之后又在枯草芽孢杆菌中发现了分别与KtrA和KtrB同源的蛋白KtrA、KtrC和KtrB、KtrD,相应地组成了与K+亲和性比较高的KtrAB和与K+亲和性相对较低的KtrCD转运系统,在二者协同作用下帮助细胞抵抗外界渗透压[57]。
目前与乳酸菌中K+转运相关的研究罕有报道。在海氏肠球菌(Enterococcus hirae)中报道的K+转运机制有KtrⅠ和KtrⅡ两种。KtrⅠ能够结合并转运K+和Rb+,但是二者的运输都需要跨细胞膜形成的质子势和ATP驱动。而KtrⅡ不依赖于质子势,只在高浓度Na+存在下被激活。研究发现KtrⅡ转运系统中,K+的吸收与Na+-ATP酶形成Na+电化学势能有关,能够特异性的转运K+[17,58]。这些Na+、K+转运系统能够使乳酸菌在渗透胁迫时迅速恢复胞内的离子平衡,降低盐胁迫对细胞造成损伤。
2.5 其他胞内代谢途径
乳酸菌在受到盐胁迫时,菌体除了通过各种代谢途径对渗透压的变化作出反应,同时必须调节胞内代谢平衡,维持细胞的正常生长。盐胁迫条件也会对细胞造成氧化损伤,造成细胞内代谢紊乱。已知NaCl是一种有效的促氧化剂,可以诱导与非典型条件相适应的蛋白质的表达。Belベore等[19]在Lactobacillus sakei CRL1756中研究发现,涉及非典型条件(假定的类铁蛋白样DNA结合蛋白,LSA1782)以及与无机离子转运和解毒过程(含铁染料脱色过氧化物酶,LSA1831)有关的2 种蛋白质在0.1 g/mLNaCl环境中是过表达的。类铁蛋白样DNA结合蛋白是饥饿细胞DNA结合蛋白(DNA-binding protein from starved cells,Dps)家族的成员,可以形成DNA-Dps共晶体,以保护DNA免受损伤环境的影响,而染料脱色过氧化物酶参与铁转运蛋白酶以及过氧化氢解毒过程的调节[59-60]。由于腌肉等肉制品是富含铁的底物,活性氧可以通过酶促反应或自发产生[18],因此L. sakei CRL1756在这些肉制品上生长过程中,类铁蛋白样DNA结合蛋白会被诱导来应对高盐对细胞造成的氧化胁迫[19]。
盐胁迫也诱导了L. sakei CRL1756中的腺苷酸琥珀酸合酶purA的合成,参与核苷酸代谢,核苷酸的积累可用于进一步合成DNA、RNA和ATP,确保细菌在胁迫条件下的存活。类似地,L. lacits中涉及嘌呤代谢的一种蛋白(bifunctional purine biosynthesis protein,PurH)在5 mol/LNaCl条件下表达上调,L. lacits在渗透胁迫条件下完成了从糖酵解到嘌呤生物合成的碳转移[20]。在L. sakei CRL1756盐胁迫还观察到基因fab1(负责编码烯酰基载体蛋白)和argS(负责编码参与脂肪酸和氨基酰-tRNA生物合成的精氨酰RNA合成酶)的下调,这些基因在L. lacits中也被报道在高渗条件下受到抑制,这些与脂肪酸代谢途径相关的基因也参与了高渗条件下细胞膜结构的调节[19,21]。
Li Yu等[61]研究发现在培养基中添加外源性的转谷氨酰胺酶,能够提高乳酸乳球菌对NaCl等许多胁迫环境的抗性,而对细胞生长没有负面影响。进一步分析发现,外源添加的转谷氨酰胺酶参与了细胞壁的合成,并且这种产生多种抗性表型的菌株是特异性的,它只发生在肽聚糖中存在谷氨酰胺与赖氨酸基团相连接的细菌中。此外,细胞面对不同的环境胁迫可能会产生交叉性应激反应,比如在酸胁迫下干酪乳杆菌中表达量显著上调的DNA修复蛋白RecO,通过重组质粒转入L. Lactis并且在细胞内过表达后,能够提高重组菌株耐盐等特性[62];Gonzalez等[63]研究报道用一些温和的高温、过氧化氢或乙醇胁迫条件预处理乳酸乳球菌后能够提高其在含有0.05 g/mL NaCl培养基中的存活率,但均未涉及到耐盐机制方面的研究。
3 结 语
乳酸菌耐盐机制的研究是目前食品科学领域研究的热点之一。迄今为止,研究者分别从相容性溶质调控系统、糖酵解关键酶调控系统、HSP调控系统、胞内离子平衡和其他一些代谢系统,详细地阐释了不同来源的乳酸菌在高盐胁迫下的调节机制,这对于开发耐盐乳酸菌和寻找合适的盐胁迫保护物质具有重要的意义,为乳酸菌能更好地应用到发酵食品工业提供了理论依据。但由于乳酸菌种类繁多,生存环境复杂多变,乳酸菌在盐胁迫下的渗透调节机制有待更全面深入的研究。随着科学技术的发展,大数据时代的来临,运用基因组学、转录组学、蛋白质组学与代谢组学相结合的方法,通过生物信息学分析,将使乳酸菌耐盐机制得到全方位的阐释,并更好地服务于发酵食品生产。
参考文献:
[1] 赵亚荣, 张文羿, 孙天松. 乳酸菌环境胁迫应答分子机制研究进展[J].中国乳品工业, 2014, 42(4): 42-45.
[2] 陈卫, 赵山山, 张秋香. 乳酸菌的耐盐机制[J]. 中国食品学报, 2013,13(10): 1-7. DOI:10.16429/j.1009-7848.2013.10.001.
[3] GLAASKER E, HEUBERGER E H, KONINGS W N, et al.Mechanism of osmotic activation of the quaternary ammonium compound transporter (QacT) of Lactobacillus plantarum[J]. Journal of Bacteriology, 1998, 180(21): 5540-5546.
[4] GLAASKER E, KONINGS W N, POOLMAN B. Glycine betaineぼuxes in Lactobacillus plantarum during osmostasis and hyper- and hypo-osmotic shock[J]. The Journal of Biological Chemistry, 1996,271(17): 10060-10065. DOI:10.1074/jbc.271.17.10060.
[5] ROMEO Y, BOUVIER J, GUTIERREZ C. Osmotic regulation of transcription in Lactococcus lactis: ionic strength-dependent binding of the BusR repressor to the busA promoter[J]. FEBS Letters, 2007,581(18): 3387-3390. DOI:10.1016/j.febslet.2007.06.037.
[6] BOUVIER J, BORDES P, ROMEO Y, et al. Characterization of OpuA,a glycine-betaine uptake system of Lactococcus lactis[J]. Journal of Molecular Microbiology and Biotechnology, 2000, 2(2): 199-205.
[7] SIRÉN N, SALONEN K, LEISOLA M, et al. A new salt inducible expression system for Lactococcus lactis[J]. Biochemical Engineering Journal, 2009, 48(1): 132-135. DOI:10.1016/j.bej.2009.08.003.
[8] BALIARDA A, ROBERT H, JEBBAR M, et al. Potential osmoprotectants for the lactic acid bacteria Pediococcus pentosaceus and Tetragenococcus halophila[J]. International Journal of Food Microbiology, 2003, 84(1): 13-20. DOI:10.1016/S0168-1605(02)00388-4.
[9] LAURET R, MOREL-DEVILLE F, BERTHIER F, et al. Carbohydrate utilization in Lactobacillus sake[J]. Applied and Environmental Microbiology, 1996, 62(6): 1922-1927.
[10] DONG S J, LIN X H, LI H. Regulation of Lactobacillus plantarum contamination on the carbohydrate and energy related metabolisms of Saccharomyces cerevisiae during bioethanol fermentation[J]. The International Journal of Biochemistry & Cell Biology, 2015, 68: 33-41.DOI:10.1016/j.biocel.2015.08.010.
[11] VEIGADACUNHA M, SANTOS H, VAN SCHAFTINGEN E.Pathway and regulation of erythritol formation in Leuconostoc oenos[J]. Journal of Bacteriology, 1993, 175(13): 3941-3948.DOI:10.1128/jb.175.13.3941-3948.1993.
[12] PRASAD J, MCJARROW P, GOPAL P. Heat and osmotic stress responses of probiotic Lactobacillus rhamnosus HN001 (DR20)in relation to viability after drying[J]. Applied and Environmental Microbiology, 2003, 69(2): 917-925. DOI:10.1128/aem.69.2.917-925.2003.
[13] WALKER D C, GIRGIS H S, KLAENHAMMER T R. The groESL chaperone operon of Lactobacillus johnsonii[J]. Applied and Environmental Microbiology, 1999, 65(7): 3033-3041.
[14] DESMOND C, FITZGERALD G F, STANTON C, et al. Improved stress tolerance of GroESL-overproducing Lactococcus lactis and probiotic Lactobacillus paracasei NFBC 338[J]. Applied and Environmental Microbiology, 2004, 70(10): 5929-5936. DOI:10.1128/aem.70.10.5929-5936.2004.
[15] KIM S G, BATT C A. Cloning and sequencing of the Lactococcus lactis subsp. lactis groESL operon[J]. Gene, 1993, 127(1): 121-126.DOI:10.1016/0378-1119(93)90626-E.
[16] EATON T, SHEARMAN C, GASSON M. Cloning and sequence analysis of the dnaK gene region of Lactococcus lactis subsp.lactis[J]. Journal of General Microbiology, 1993, 139(12): 3253-3264.DOI:10.1099/00221287-139-12-3253.
[17] KAWANO M, IGARASHI K, KAKINUMA Y. Two major potassium uptake systems, KtrⅠ and KtrⅡ, in Enterococcus hirae[J]. FEMS Microbiology Letters, 1999, 176(2): 449-453. DOI:10.1016/S0378-1097(99)00271-2.
[18] MCLEOD A, ZAGOREC M, CHAMPOMIER-VERGÈS M C, et al.Primary metabolism in Lactobacillus sakei food isolates by proteomic analysis[J]. BMC Microbiology, 2010, 10: 1-10. DOI:10.1186/1471-2180-10-120.
[19] BELFIORE C, FADDA S, RAYA R, et al. Molecular basis of the adaption of the anchovy isolate Lactobacillus sakei CRL1756 to salted environments through a proteomic approach[J]. Food Research International, 2013, 54(1): 1334-1341. DOI:10.1016/j.foodres.2012.09.009.
[20] ZHANG Y H, ZHANG Y P, ZHU Y, et al. Proteomic analyses to reveal the protective role of glutathione in resistance of Lactococcus lactis to osmotic stress[J]. Applied and Environmental Microbiology, 2010,76(10): 3177-3186. DOI:10.1128/AEM.02942-09.
[21] XIE Y, CHOU L S, CUTLER A, et al. DNA Macroarray profiling of Lactococcus lactis subsp. lactis IL1403 gene expression during environmental stresses[J]. Applied and Environmental Microbiology,2004, 70(11): 6738-6747. DOI:10.1128/aem.70.11.6738-6747.2004.
[22] BROWN A D, SIMPSON J R. Water relations of sugar-tolerant yeasts:the role of intracellular polyols[J]. Journal of General Microbiology,1972, 72(3): 589-591. DOI:10.1099/00221287-72-3-589.
[23] ROBERTS M F. Organic compatible solutes of halotolerant and halophilic microorganisms[J]. Saline Systems, 2005, 1: 5.DOI:10.1186/1746-1448-1-5.
[24] GALINSKI E A, TRUPER H G. Microbial behaviour in salt-stressed ecosystems[J]. FEMS Microbiology Reviews, 1994, 15(2/3): 95-108.DOI:10.1111/j.1574-6976.1994.tb00128.x.
[25] KEMPF B, BREMER E. Uptake and synthesis of compatible solutes as microbial stress responses to high-osmolality environments[J].Archives of Microbiology, 1998, 170(5): 319-330. DOI:10.1007/s002030050649.
[26] WOOD J M, BREMER E, CSONKA L N, et al. Osmosensing and osmoregulatory compatible solute accumulation by bacteria[J].Comparative Biochemistry and Physiology-Part A: Molecular &Integrative Physiology, 2001, 130(3): 437-460. DOI:10.1016/s1095-6433(01)00442-1.
[27] 高薇, 韩雪, 张兰威. 乳酸菌渗透胁迫相关相容性溶质及其转运机制研究进展[J]. 微生物学通报, 2013, 40(11): 2097-2106.DOI:10.13344/j.microbiol.china.2013.11.026.
[28] DINNBIER U, LIMPINSEL E, SCHMID R, et al. Transient accumulation of potassium glutamate and its replacement by trehalose during adaptation of growing cells of Escherichia coli K-12 to elevated sodium chloride concentrations[J]. Archives of Microbiology, 1988,150(4): 348-357. DOI:10.1007/BF00408306.
[29] JONES P M, GEORGE A M. Subunit interactions in ABC transporters:towards a functional architecture[J]. FEMS Microbiology Letters,1999, 179(2): 187-202. DOI:10.1111/j.1574-6968.1999.tb08727.x.
[30] CHAMPAGNE C P, RAYMOND Y, SIMON J P. Effect of water activity and protective solutes on growth and subsequent survival to air-drying of Lactobacillus and Biベdobacterium cultures[J]. Applied Microbiology and Biotechnology, 2012, 95(3): 745-756. DOI:10.1007/s00253-012-3912-8.
[31] GUL N, SCHUURMAN-WOLTERS G, KARASAWA A, et al. Functional characterization of amphipathic α-helix in the osmoregulatory ABC transporter OpuA[J]. Biochemistry, 2012,51(25): 5142-5152. DOI:10.1021/bi300451a.
[32] MAHMOOD N A, BIEMANS-OLDEHINKEL E, POOLMAN B.Engineering of ion sensing by the cystathionine beta-synthase module of the ABC transporter OpuA[J]. The Journal of Biological Chemistry,2009, 284(21): 14368-14376. DOI:10.1074/jbc.M901238200.
[33] WOLTERS J C, BERNTSSON R P, GUL N, et al. Ligand binding and crystal structures of the substrate-binding domain of the ABC transporter OpuA[J]. PLoS ONE, 2010, 5(4): e10361. DOI:10.1371/journal.pone.0010361.
[34] PATZLAFF J S, VAN DER HEIDE T, POOLMAN B. The ATP/substrate stoichiometry of the ATP-binding cassette (ABC) transporter OpuA[J]. The Journal of Biological Chemistry, 2003, 278(32): 29546-29551. DOI:10.1074/jbc.M304796200.
[35] ROMEO Y, OBIS D, BOUVIER J, et al. Osmoregulation in Lactococcus lactis: BusR, a transcriptional repressor of the glycine betaine uptake system BusA[J]. Molecular Microbiology, 2003, 47(4):1135-1147. DOI:10.1046/j.1365-2958.2003.03362.x.
[36] KARASAWA A, ERKENS G B, BERNTSSON R P, et al.Cystathionine β-synthase (CBS) domains 1 and 2 fulベll diあerent roles in ionic strength sensing of the ATP-binding cassette (ABC) transporter OpuA[J]. The Journal of Biological Chemistry, 2011, 286(43): 37280-37291. DOI:10.1074/jbc.M111.284059.
[37] HOFFMANN T, WENSING A, BROSIUS M, et al. Osmotic control of opuA expression in Bacillus subtilis and its modulation in response to intracellular glycine betaine and proline pools[J]. Journal of Bacteriology, 2013, 195(3): 510-522. DOI:10.1128/jb.01505-12.
[38] KANG T S, KORBER D R, TANAKA T. Regulation of dual glycolytic pathways for fructose metabolism in heterofermentative Lactobacillus panis PM1[J]. Applied and Environmental Microbiology, 2013, 79(24):7818-7826. DOI:10.1128/aem.02377-13.
[39] 刘倩颖. 基于RT-PCR技术对植物乳杆菌耐盐分子机理的研究[D].沈阳: 沈阳农业大学, 2014: 13-15.
[40] LESLIE S B, ISRAELI E, LIGHTHART B, et al. Trehalose and sucrose protect both membranes and proteins in intact bacteria during drying[J]. Applied and Environmental Microbiology, 1995, 61(10):3592-3597.
[41] FU X M. Chaperone function and mechanism of small heat-shock proteins[J]. Acta Biochimica et Biophysica Sinica, 2014, 46(5): 347-356. DOI:10.1093/abbs/gmt152.
[42] HENDRICK J P, HARTL F U. Molecular chaperone functions of heatshock proteins[J]. Annual Review of Biochemistry, 1993, 62: 349-384.DOI:10.1146/annurev.bi.62.070193.002025.
[43] GEORGELLIS D, SOHLBERG B, HARTL F U, et al. Identiベcation of GroEL as a constituent of an mRNA-protection complex in Escherichia coli[J]. Molecular Microbiology, 1995, 16(6): 1259-1268.DOI:10.1111/j.1365-2958.1995.tb02347.x.
[44] CHANG Y H, SHANGKUAN Y H, LIN H C, et al. PCR assay of the groEL gene for detection and diあerentiation of Bacillus cereus group cells[J]. Applied and Environmental Microbiology, 2003, 69(8): 4502-4510. DOI:10.1128/AEM.69.8.4502-4510.2003.
[45] WETZSTEIN M, VÖLKER U, DEDIO J, et al. Cloning, sequencing,and molecular analysis of the dnaK locus from Bacillus subtilis[J].Journal of Bacteriology, 1992, 174(10): 3300-3310. DOI:10.1128/jb.174.10.3300-3310.1992.
[46] SCHULZ A, SCHUMANN W. hrcA, the first gene of the Bacillus subtilisdnaK operon encodes a negative regulator of class I heat shock genes[J]. Journal of Bacteriology, 1996, 178(4): 1088-1093.
[47] YUAN G, WONG S L. Isolation and characterization of Bacillus subtilisgroE regulatory mutants: evidence for orf39 in the dnaK operon as a repressor gene in regulating the expression of both groE and dnaK[J]. Journal of Bacteriology, 1995, 177(22): 6462-6468.
[48] KILSTRUP M, JACOBSEN S, HAMMER K, et al. Induction of heat shock proteins DnaK, GroEL, and GroES by salt stress in Lactococcus lactis[J]. Applied and Environmental Microbiology, 1997, 63(5): 1826-1837.
[49] GERCHMAN Y, RIMON A, VENTURI M, et al. Oligomerization of NhaA, the Na+/H+antiporter of Escherichia coli in the membrane and its functional and structural consequences[J]. Biochemistry, 2001,40(11): 3403-3412. DOI:10.1021/bi002669o.
[50] DWIVEDI M, SUKENIK S, FRIEDLER A, et al. The Ec-NhaA antiporter switches from antagonistic to synergistic antiport upon a single point mutation[J]. Scientific Reports, 2016, 6: 23339.DOI:10.1038/srep23339.
[51] WU X H, ALTMAN R, EITEMAN M A, et al. Effect of overexpressing nhaA and nhaR on sodium tolerance and lactate production in Escherichia coli[J]. Journal of Biological Engineering,2013, 7(1): 1-7. DOI:10.1186/1754-1611-7-3.
[52] PADAN E, VENTURI M, GERCHMAN Y, et al. Na+/H+antiporters[J].Biochimica et Biophysica Acta, 2001, 1505(1): 144-157. DOI:10.1016/S0005-2728(00)00284-X.
[53] MELO A M P, FELIX N A M, CARITA J N, et al. The Na+/H+antiporter of the thermohalophilic bacterium Rhodothermus marinus[J]. Biochemical and Biophysical Research Communications,2006, 348(3): 1011-1017. DOI:10.1016/j.bbrc.2006.07.134.
[54] CALDERÓN M I, VARGAS C, ROJO F, et al. Complex regulation of the synthesis of the compatible solute ectoine in the halophilic bacterium Chromohalobacter salexigens DSM 3043T[J].Microbiology, 2004, 150(9): 3051-3063. DOI:10.1099/mic.0.27122-0.[55] UEDA A, WOOD T K. Potassium and sodium transporters of Pseudomonas aeruginosa regulate virulence to barley[J]. Applied Microbiology and Biotechnology, 2008, 79(5): 843-858. DOI:10.1007/s00253-008-1483-5.
[56] NAKAMURA T, YUDA R, UNEMOTO T, et al. KtrAB, a new type of bacterial K+-uptake system from Vibrio alginolyticus[J]. Journal of Bacteriology, 1998, 180(13): 3491-3494.
[57] HOLTMANN G, BAKKER E P, UOZUMI N, et al. KtrAB and KtrCD:two K+uptake systems in Bacillus subtilis and their role in adaptation to hypertonicity[J]. Journal of Bacteriology, 2003, 185(4): 1289-1298.DOI:10.1128/jb.185.4.1289-1298.2003.
[58] 刘礼崔. 嗜盐四联球菌CICC 10469盐胁迫应答机制的比较转录组学研究[D]. 广州: 华南理工大学, 2015: 4-5.
[59] HORSBURGH M J, CLEMENTS M O, CROSSLEY H, et al. PerR controls oxidative stress resistance and iron storage proteins and is required for virulence in Staphylococcus aureus[J]. Infection and Immunity, 2001, 69(6): 3744-3754. DOI:10.1128/iai.69.6.3744-3754.2001.
[60] ZHAO G H, CECI P, ILARI A, et al. Iron and hydrogen peroxide detoxiベcation properties of DNA-binding protein from starved cells. A ferritin-like DNA-binding protein of Escherichia coli[J]. The Joural of Biological Chemistry, 2002, 277(31): 27689-27696. DOI:10.1074/jbc.M202094200.
[61] LI Yu, KAN Zhipeng, YOU Yuanli, et al. Exogenous transglutaminase improves multiple-stress tolerance in Lactococcus lactis and other lactic acid bacteria with glutamine and lysine in the cell wall[J].Biotechnology Letters, 2015, 37(12): 2467-2474. DOI:10.1007/s10529-015-1942-x.
[62] WU C D, ZHANG J, DU G C, et al. Heterologous expression of Lactobacillus casei RecO improved the multiple-stress tolerance and lactic acid production in Lactococcus lactis NZ9000 during salt stress[J]. Bioresource Technology, 2013, 143: 238-241. DOI:10.1016/j.biortech.2013.05.050.
[63] GONZALEZ E E, OLSON D, ARYANA K. Short communication: salt tolerance of Lactococcus lactis R-604 as influenced by mild stresses from ethanol, heat, hydrogen peroxide, and UV light[J]. Journal of Dairy Science, 2017, 100(6): 4290-4293. DOI:10.3168/jds.2016-12088.