硝酸盐和亚硝酸盐主要通过蔬菜食用进入人体,亚硝酸盐是一种有毒物质,人食用后会在体内产生致癌性的亚硝胺[1-2],可导致胃癌、肠癌和肝癌等[3],且大量摄入亚硝酸盐可诱发高铁血红蛋白症[4],产生常见的亚硝酸盐急性中毒症状[5]。因此严格控制食品中亚硝酸盐含量非常重要。当前研究的反硝化细菌大多为致病微生物和土壤微生物,例如Geobacillus kaustophilus[6]、Bacillus megaterium[7]、Geobacillus thermodenitrificans[8]、Alcaligenes xylosoxidans[9]、Ralstonia pickettii[10-11],它们能够合成一种亚硝酸盐还原酶(nitrite reductase,NiR),其具有较强的降解亚硝酸盐的能力,但是这些细菌不能食用,无法应用在食品中。目前应用在食品中的反硝化细菌主要是乳杆菌[12-14],已报道的有干酪乳杆菌鼠李糖亚种(Lactobacillus casei subsp. rhamnosus)6013[15]、植物乳杆菌(L. plantarum)[16-17]、短乳杆菌(L. brevis)[18]等。研究表明,大多数乳酸菌的NiR活性对于土壤中的反硝化细菌来说是比较低的。
本研究组在豆瓣酱中分离得到一株蜡样芽孢杆菌(Bacillus cereus),命名为LJ01,可应用于食品中,前期研究发现该菌株同样可以合成具有高活性的NiR,而目前食品来源的芽孢杆菌中NiR的研究极少,且对于其中NiR的分离纯化和性质研究还鲜见科学的方法。本研究组在前期采用直接从B. cereus LJ01天然菌中分离纯化的方式无法得到大量的NiR,这为今后的研究带来困扰。为了更好地研究B. cereus LJ01中NiR的性质,必须提高NiR的提取量。本研究利用克隆表达技术,将B. cereus LJ01中NiR基因片段克隆到原核表达载体Escherichia coli BL21上,利用异丙基硫代-β-D-半乳糖苷(isopropylthio-β-D-galactoside,IPTG)对NiR进行诱导表达,从而得到大量的重组NiR,以便后续性质的研究。
1 材料与方法
1.1 材料与试剂
1.1.1 菌种与载体
B. cereus LJ01、原核载体质粒pET32a(+)、克隆宿主E. coli DH5α和表达宿主E. coli BL21(DE3)由本实验室保存于-80 ℃冰箱中。
1.1.2 试剂与培养基
核酸和蛋白电泳Marker、SalI限制性内切酶、Taq DNA聚合酶、T4 DNA连接酶、AxyPrepTMDNA Gel Extraction Kit 50-prep试剂盒 宝生物工程(大连)有限公司;柱式细菌DNAout试剂盒、NcoI限制性内切酶北京天恩泽基因科技有限公司;氨苄青霉素、IPTG、丙烯酰胺、甲叉双丙烯酰胺、Tris、过硫酸钠 广州卯林试剂有限公司。
LB培养基参照文献[19]配制;十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate-polyacrylamide gel electrophoresis,SDS-PAGE)试剂的配制参照文献[20]。
1.2 仪器与设备
立式压力蒸汽灭菌锅 上海博迅实业有限公司医疗设备厂;SW-CJ-2F洁净工作台 苏州安泰空气技术有限公司;HYG-A全温摇瓶柜 太仓市实验设备厂;WD-9403B紫外仪、DYY-6C型电泳仪 北京市六一仪器厂;聚合酶链式反应(polymerase chain reaction,PCR)仪、JW-3021 HR高速冷冻离心机 安徽嘉文仪器装备有限公司;亲和层析柱Ni Sepharose 6 Fast Flow 美国GE公司;5800 MALDI-TOF/TOF基质辅助激光解吸电离飞行时间质谱仪 德国Bruker公司;Chirascan圆二色谱仪英国应用光物理公司;电感耦合等离子体质谱仪 赛默飞世尔科技(中国)有限公司。
1.3 方法
1.3.1 引物设计与合成
在NCBI中查找蜡样芽孢杆菌中NiR蛋白序列(GenBank编号CP009628),然后根据蛋白序列的位点,在蜡样芽孢杆菌的全基因组中找到其编码基因的序列,根据找到的NiR编码序列(nir)设计引物,即上游引物 Bc-NP1:5’-CATGCCATGGATGA GTTATGAAAAAGTAT-3’,下游引物Bc-NP2:5’-ACGCGTCGACCTAAGAGCTATTACTTCT-3’,下划线为保护性碱基,分别从其中引入NcoI和SalI的酶切位点。引物由英潍捷基上海有限公司合成。
1.3.2 基因克隆和测序
用基因组DNA提取试剂盒抽提B. cereus LJ01的基因组DNA,以其为模板扩增nir片段。将PCR产物上样至1%琼脂糖凝胶中,在紫外灯下观察结果,并利用QIAquick PCR Purification kit试剂盒对PCR产物进行回收,送至生工生物工程(上海)有限公司进行测序[21]。将测得的DNA序列翻译成蛋白质序列后,与GenBank收录的NiR蛋白序列对比,将正确的NiR的DNA序列登录GenBank。
1.3.3 重组菌株pET-32a(+)-nir-BL21的构建
由于两个限制性内切酶的最适buffer不同,因此不进行双酶切,而是分别进行单酶切。将回收的目的nir片段、pET-32a(+)质粒分别进行NcoI和SalI单酶切,经1%琼脂糖凝胶电泳分析,胶回收酶切后的目的基因nir和pET-32a(+)[22-23]。利用T4 DNA连接酶将目的基因nir连接到pET载体上,构建重组质粒pET-32a(+)-nir。将连接产物转化到E. coli BL21(DE3)中,同时向另两个装有E.coli BL21感受态细胞的离心管中分别加入空质粒pET-32a(+)和无菌水(两者均作为空白对照),用移液枪轻轻吹打混匀后冰浴30 min。
将3 个离心管均放入42 ℃的水浴锅中保温90 s后,快速冷却细胞2~3 min。向每个离心管中分别加入800 μL液体LB培养基,置于37 ℃培养箱中静置培养45 min,使细菌复苏。分别取100 μL以上3 种培养液涂布于含50 μg/mL氨苄青霉素的固体LB培养基上,观察平板菌落生长情况。在重组质粒组平板上随机挑取10 个单菌落进行PCR鉴定。再将阳性菌株分别培养后,送菌液至生工生物工程(上海)有限公司进行测序。
1.3.4 重组菌NiR的表达鉴定
将得到的阳性克隆菌种按2%接种量接种至LB液体培养基(含50 µg/mL氨苄青霉素),于37 ℃、180 r/min下培养至对数生长期(OD600nm=0.6)时,加入终浓度为1 mmol/L的IPTG进行诱导,培养4 h时终止发酵,将培养液在4 ℃、8 000 r/min条件下离心收集菌体,用无菌水清洗两次,每100 mL培养液加入10 mL已4 ℃预冷的0.02 mol/L的磷酸缓冲液(pH 7.4),同时加入终质量浓度为2 µg/mL的胰蛋白酶抑制剂,冰浴超声波破碎菌体,离心取上清液,记为NiR粗酶液。同时设立对照组,一组为加IPTG诱导的含有空质粒的BL21组(阴性对照组),另一组为不加IPTG诱导的含重组质粒的BL21组(阳性对照组),按同样的方法收集粗酶液,在相同浓度下进行SDS-PAGE分析,以初步判断NiR是否成功表达。
1.3.5 重组NiR的分离纯化
利用亲和层析柱Ni Sepharose 6 Fast Flow分离纯化得到目的蛋白,具体纯化操作参照文献[16],并对相应组分进行酶活性测定。经Ni柱分离后收集到的蛋白进行透析后浓缩,利用DEAE Sepharose Fast Flow对浓缩后的蛋白进行二次分离,先用不含NaCl的50 mmol/L HEPES缓冲液(pH 7.4)将杂蛋白洗脱下来,再用含0~300 mmol/L NaCl的50 mmol/L HEPES缓冲液(pH 7.4)进行梯度洗脱,流速为1 mL/min,分部收集洗脱液,每管收集洗脱液1.5 mL,测定每管洗脱液的蛋白含量、降解亚硝酸盐的活性及NiR活性,收集和合并有NiR活性的组分,再用聚乙二醇20000浓缩至所需浓度,于-80 ℃保存备用。其中酶活性测定体系:在1.5 mL离心管中加入分别用0、100、200 mmol/L NaCl洗脱下来的洗脱液100 μL、500 mg/L的NaNO2100 μL、 0.1 mol/L亚硫酸钠100 μL,再用HEPES缓冲液定容到500 μL,盐酸萘乙二胺法测定残留的亚硝酸盐含量。NiR活性单位定义为:在37 ℃条件下,每分钟每毫克蛋白催化还原亚硝酸钠的质量(ng)为一个酶活力单位。
1.3.6 重组NiR的性质分析
Native-PAGE测定:利用样品缓冲液将上述步骤中得到的重组蛋白进行样品制备,凝胶为10 g/100 mL丙烯酰胺,电压为120 V,整个电泳过程处于冰浴中,电泳2 h后,进行考马斯亮蓝染色,再脱色后拍照。
质谱测定:通过基质辅助激光解吸电离飞行时间质谱对供试品的相对分子质量进行分析。
圆二色谱测定:利用圆二色谱仪分别对重组蛋白、经100 ℃高温处理后的重组蛋白和经高压灭菌的重组蛋白进行二级结构的测定,并用Pro-uta Viewer软件分析结果。样品分为3 组,分别为重组NiR、经100 ℃加热处理15 min后的重组NiR以及经121 ℃高压灭菌的重组NiR。
金属含量的测定:利用电感耦合等离子体质谱对重组NiR进行金属含量测定。
2 结果与分析
2.1 目的基因nir的PCR扩增
提取B. cereus LJ01的基因组DNA,并以此为模板得到的PCR扩增产物经1%琼脂糖凝胶电泳后结果如图1,扩增出的条带约1 600 bp ,条带单一,明亮清晰。该大小与预计的编码基因大小1 623 bp几乎相一致,因此可以初步判定该条带即为目的基因片段。将纯化得到的基因片段送至生工生物工程(上海)有限公司进行测序。测序结果表明NiR含有一个1 623 bp的开放阅读框,编码540 个氨基酸序列,将测得的DNA序列翻译成蛋白质序列后,与GenBank收录的相比,结果与B. thuringiensis strain XL6(Accession number:CP013000.1)的铁氧化还原蛋白亚硝酸还原酶的碱基序列相似性达到99%。在B. thuringiensis strain XL6菌株中第1 610个碱基是鸟嘌呤(G),而LJ01对应的是胸腺嘧啶(T),相应地,第537个密码子编码的氨基酸由前者的缬氨酸变成了后者的甘氨酸,将编码B. cereus LJ01中NiR蛋白的DNA序列上传至NCBI中,获得Accession number:MG839504。

图1 B. cereus LJ01中nir基因PCR扩增产物的琼脂糖凝胶电泳图
Fig. 1 Gel electrophoresis of PCR-amplified nir gene from B. cereus LJ01
2.2 重组表达载体的构建结果
实验中阴性组未长出菌落,而阳性组的菌落较多,说明感受态细胞制备成功。在重组菌平板上挑取10 个单菌落进行PCR初步鉴定,结果见图2,其中只有3号和10号未扩增出条带,其余8 株菌均能扩增出目的片段,说明挑取的10 株重组菌中,有8 株含有目标片段,将上述8 株重组菌分别培养后,送菌液至生工生物工程(上海)有限公司进行测序,测序结果均正确。
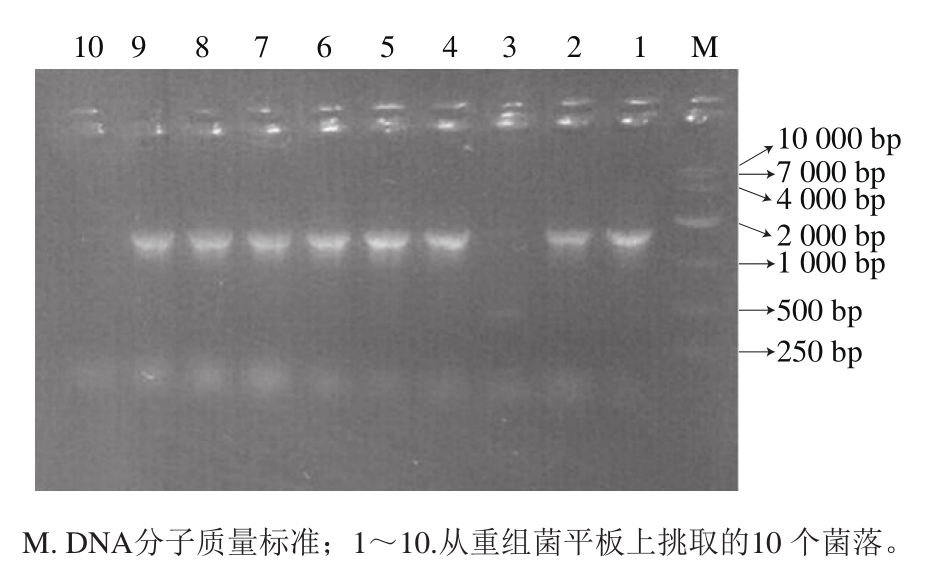
图2 重组大肠杆菌的菌落PCR鉴定
Fig. 2 PCR identification of recombinant E. coli
2.3 重组菌株中的NiR诱导表达及其分离纯化
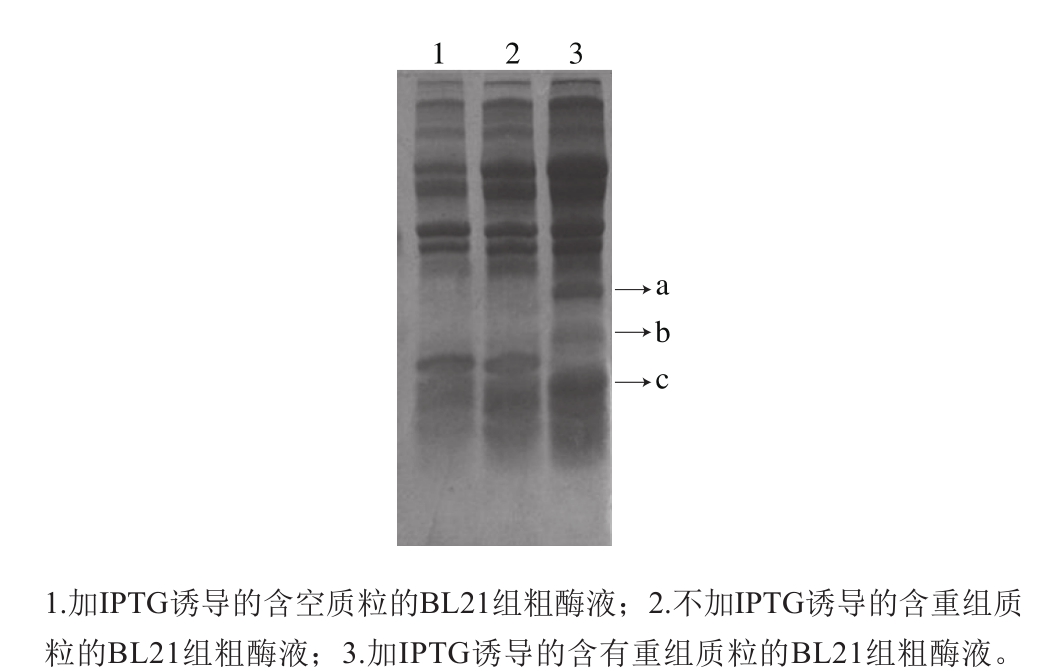
图3 大肠杆菌中粗酶液的电泳图
Fig. 3 Electrophoresis of crude enzyme from E. coli
pET-32a(+)-nir-BL21中NiR的诱导表达结果如图3所示,1、2条带的蛋白基本一致,而与前两条带相比,条带3在箭头所指a、b和c的位置多出3 处条带,说明该重组蛋白可能已经成功得到表达。
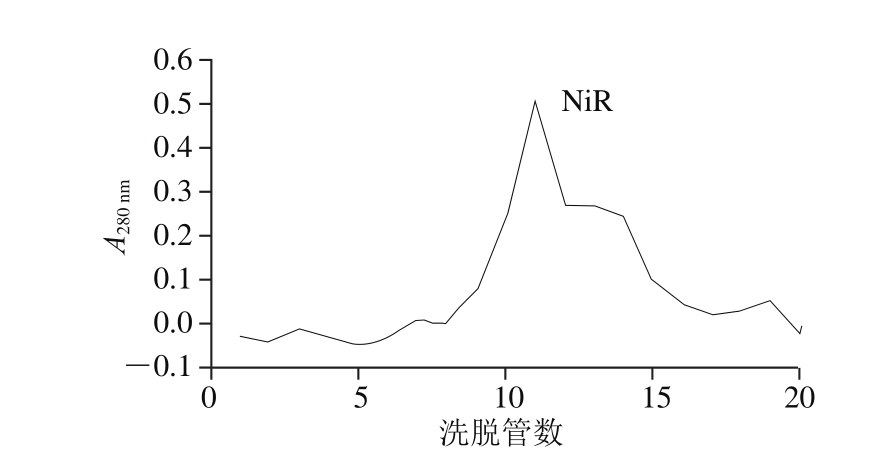
图4 重组NiR的亲和层析
Fig. 4 Affinity chromatography of recombinant NiR
将上述所得的重组蛋白粗酶液进行亲和层析纯化。重组NiR经500 mmol/L咪唑洗脱曲线如图4所示,收集后测定酶活性,经计算其产量为1 L发酵液中可得到20~30 mg重组蛋白。

图5 重组NiR的DEAE Sepharose Fast Flow层析
Fig. 5 Chromatography of recombinant NiR on DEAE Sepharose Fast Flow
将经亲和层析柱洗脱后收集的蛋白再次浓缩,取40 mL上样至DEAE Sepharose Fast Flow,其蛋白洗脱曲线如图5所示。其中第1个峰的NaCl浓度为200 mmol/L,后一个峰的NaCl浓度为1 mol/L,说明大多数的重组NiR都在200 mmol/L的盐浓度下被洗脱出来,且测得NiR活性为10 987.65 U/mg,说明经克隆表达并纯化出的是正确的NiR。洗脱出的酶液经聚乙二醇20000浓缩后进行SDSPAGE检测,其结果如图6所示,对比条带1和条带2得出箭头所指处为目的蛋白NiR,对照3、4、5条带说明图3中箭头c所指的条带为重组蛋白,同时发现其中还有少许杂带,还需要进一步浓缩来提高其纯度。

2.4 重组NiR的性质分析
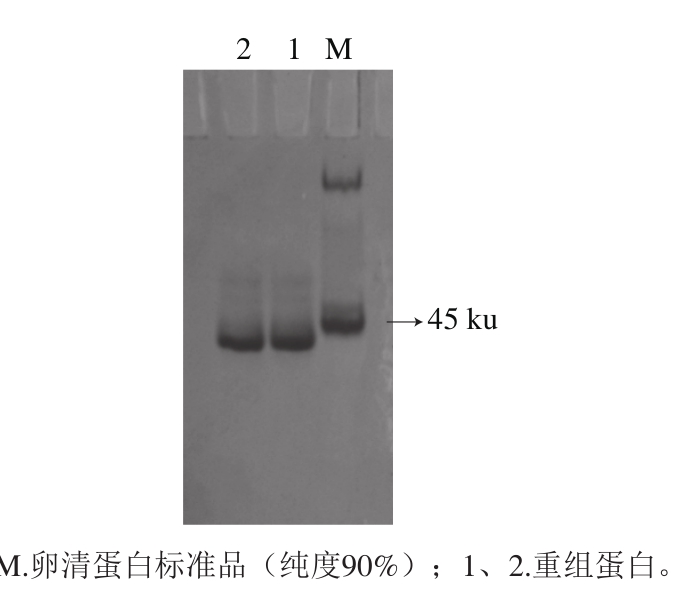
图7 重组蛋白的非变性聚丙烯酰胺电泳图
Fig. 7 Non-denaturing polyacrylamide gel electrophoresis of recombinant protein
将得到的重组蛋白进行非变性聚丙烯酰胺电泳检测,结果见图7,泳道1、2 均为单一条带,说明样品蛋白为单一蛋白,因此推测图6中1、2道中上方的条带为聚合物,同时重组蛋白较天然蛋白相比多了组氨酸头,因此导致组成NiR的两个单体分子质量有所差异。图7中按照分子质量的迁移来看,重组NiR的分子质量应略小于45 ku,约为40 ku,这和理论分子质量(约60 ku)差距较大。之所以出现这种偏差,是由于在非变性聚丙烯酰胺电泳中,蛋白质的迁移取决于许多因素,包括大小,形状和电荷[24],蛋白分子的空间结构也会影响条带的迁移,因此无法凭借标准蛋白来确定重组NiR的准确分子质量。有研究利用非变性聚丙烯酰胺电泳来分离纯化蛋白[24-25]和研究蛋白间的相互作用[26],而几乎没有利用非变性聚丙烯酰胺电泳来确定其天然分子质量的研究。

图8 不同状态下重组NiR的非变性聚丙烯酰胺电泳图
Fig. 8 Non-denaturing polyacrylamide gel electrophoresis of recombinant NiR under different conditions
为进一步确定空间结构对蛋白迁移的影响,重组NiR经不同处理后的非变性聚丙烯酰胺电泳结果见图8, 1、2、3蛋白带的位置没有明显变化,4蛋白带明显向下迁移,这可能是因为3号样品是经100 ℃加热的,其空间变化不大,而4号样品的处理条件为高温高压,空间结构发生明显变化,因此迁移量与前3组有明显区别。研究表明,高压处理对如氨基酸、维生素和风味化合物等小分子无影响[27],但是对球状蛋白质的三级和四级结构有破坏性的影响,会破坏其非共价键[28]。因此不能通过非变性聚丙烯酰胺电泳来确定重组NiR的准确分子质量,而是需要利用质谱等方法来确定。
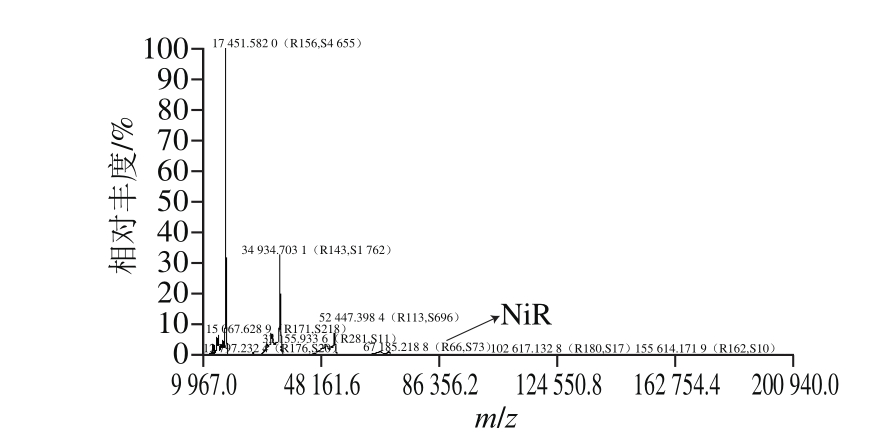
图9 重组蛋白质谱图
Fig. 9 Mass spectrometry of recombinant protein
重组NiR质谱图见图9,根据其DNA大小推测得到的分子质量约为57 ku,同时质粒pET-32a(+)上有Histags等标签,因此判定图9中大小为67.185 218 8 ku的组分为重组NiR样品的分子质量。经不同处理的重组蛋白的圆二色谱图见图10,和重组蛋白相比,100 ℃加热后的重组蛋白其α-螺旋结构含量有所降低,而经高压灭菌后的重组蛋白其α-螺旋结构含量有明显的下降。根据电感耦合等离子体质谱的测定结果,重组NiR中铁离子质量浓度为0.034 mg/L,铜离子质量浓度为0.123 mg/L,样品质量浓度为2/3 mg/mL,因此最后得到样品中铁离子含量为51 mg/kg,铜离子含量为184.5 mg/kg。据报道,无论是铁离子还是铜离子,都可能对NiR的活性产生影响。在Cu型NiR中,存在I型Cu中心和II型Cu中心,其中I型Cu中心是电子转移位点,II型Cu中心是酶催化位点[29],说明Cu原子对于Cu型NiR的催化具有促进作用,同时研究表明,亚硝酸盐与T2铜结合后可以提高该中心的氧化还原电位,从而使分子内电子转移更有利[5]。在菠菜中,NiR在铁-硫簇的轴向位置与血红素铁结合,其中铁-硫域为其活性中心[30]。而B. cereus LJ01中的NiR同时含有铁离子和铜离子,而对于两种金属离子在NiR中的作用,还要进行下一步探讨。
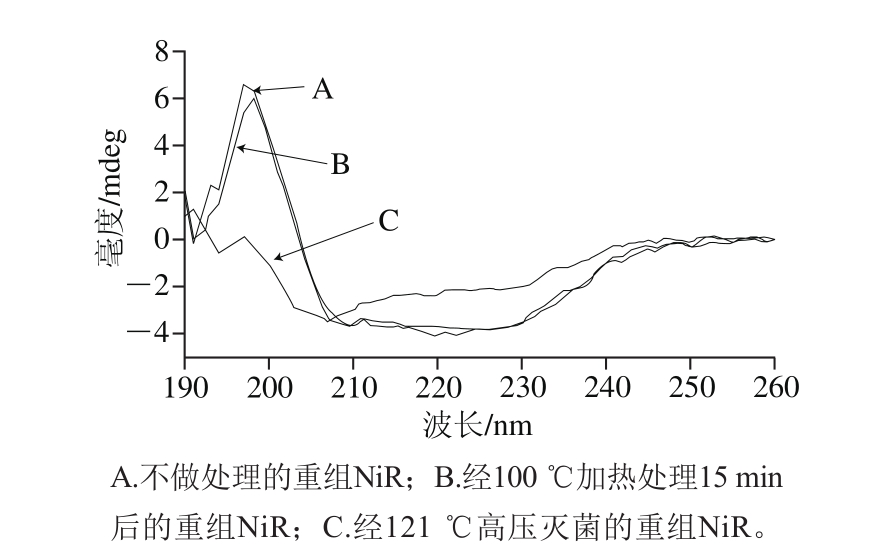
图10 重组蛋白圆二色谱图
Fig. 10 Circular dichroism spectra of recombinant protein
3 结 论
采用克隆表达技术提高NiR的量,于NCBI上查找蜡样芽孢杆菌中NiR的DNA序列,根据该序列设计引物,以B. cereus LJ01全基因为模板进行PCR扩增,将目的片段双酶切后连接于质粒pET-32a(+)上,再导入大肠杆菌BL21中,利用含50 μg/mL氨苄青霉素的LB平板对重组菌进行筛选,用菌落PCR和测序进行验证,得到含重组质粒的重组BL21。重组菌用IPTG进行诱导产酶,采用超声破碎法离心收集得到重组菌的粗酶液,利用Ni柱亲和层析和DEAE Sepharose Fast Flow交换层析对粗蛋白进行纯化,并研究了重组NiR的性质,结果表明:该NiR含有一个1 623 bp的开放阅读框,编码540 个氨基酸序列。重组NiR的分子质量约为67 ku;该酶中同时存在铁离子和铜离子,且含量分别为51.0 mg/kg和184.5 mg/kg;圆二色谱结果显示α-螺旋结构在重组NiR中所占比例最大。
参考文献:
[1] SEN N P, SEAMAN S W, BADDOO P A, et al. Formation of N-nitroso-N-methylurea in various samples of smoked/dried fish,fish sauce, seafoods, and ethnic fermented/pickled vegetables following incubation with nitrite under acidic conditions[J].Journal of Agricultural & Food Chemistry, 2001, 49(4): 2096-2103.DOI:10.1021/jf0011384.
[2] 鲁奇林, 王娜, 冯叙桥, 等. 大白菜贮藏过程中硝酸盐和亚硝酸盐含量变化分析[J]. 食品科学, 2014, 35(18): 151-155. DOI:10.7506/spkx1002-6630-201418030.
[3] GANGOLLI S D, PA V D B, FERON V J, et al. Nitrate, nitrite and N-nitroso compounds[J]. European Journal of Pharmacology, 1994,292(1): 1-38. DOI:10.1016/0926-6917(94)90022-1.
[4] SPENCER J P, WHITEMAN M, JENNER A, et al. Nitrite-induced deamination and hypochlorite-induced oxidation of DNA in intact human respiratory tract epithelial cells[J]. Free Radical Biology & Medicine,2000, 28(7): 1039-1050. DOI:10.1016/S0891-5849(00)00190-8.
[5] HABERMEYER M, ROTH A, GUTH S, et al. Nitrate and nitrite in the diet: how to assess their benefit and risk for human health[J].Molecular Nutrition & Food Research, 2015, 59(1): 106-128.DOI:10.1002/mnfr.201400286.
[6] FUKUDA Y, KOTEISHI H, YONEDA R, et al. Structural and functional characterization of the Geobacillus copper nitrite reductase:Involvement of the unique N-terminal region in the interprotein electron transfer with its redox partner[J]. Biochimica et Biophysica Acta (BBA)-Bioenergetics, 2014, 1837(3): 396-405. DOI:10.1016/j.bbabio.2014.01.004.
[7] AIKO T, RYOSUKE I, HIROYASU K, et al. Structural and mechanistic insights into the electron flow through protein for cytochrome c-tethering copper nitrite reductase[J]. Journal of Biochemistry, 2013, 154(1): 51-60. DOI:10.1093/jb/mvt023.
[8] FUKUDA Y, TSE K M, LINTULUOTO M, et al. Structural insights into the function of a thermostable copper-containing nitrite reductase[J].Journal of Biochemistry, 2014, 155(2): 123-135. DOI:10.1093/jb/mvt107.
[9] LEFERINK N G H, ANTONYUK S V, HOUWMAN J A, et al. Impact of residues remote from the catalytic centre on enzyme catalysis of copper nitrite reductase[J]. Nature Communications, 2014, 5(5): 4395.DOI:10.1038/ncomms5395.
[10] HAN C, WRIGHT G S, FISHER K, et al. Characterization of a novel copper-haem c dissimilatory nitrite reductase from Ralstonia pickettii[J]. Biochemical Journal, 2012, 444(2): 219-226. DOI:10.1042/BJ20111623.
[11] ANTONYUK S V, HAN C, EADY R R, et al. Structures of proteinprotein complexes involved in electron transfer[J]. Nature, 2013, 496:123-126. DOI:10.1038/nature11996.
[12] WANG X H, REN H Y, LIU D Y, et al. Effects of inoculating Lactobacillus sakei starter cultures on the microbiological quality and nitrite depletion of Chinese fermented sausages[J]. Food Control,2013, 32(2): 591-596. DOI:10.1016/j.foodcont.2013.01.050.
[13] 吴慧昊, 牛锋, 陈珊珊, 等. 高效降亚硝酸盐乳酸菌的驯化复筛及菌株鉴定[J]. 食品科学, 2016, 37(19): 160-165. DOI:10.7506/spkx1002-6630-201619027.
[14] 于华, 刘有晴, 黄丹, 等. 泡菜中高产酸、高效降解亚硝酸盐的乳酸菌分离鉴定及应用[J]. 中国调味品, 2016, 41(6): 34-38.DOI:10.3969/j.issn.1000-9973.2016.06.007.
[15] 张馨月. 干酪乳杆菌LCR 6013降解亚硝酸盐的途径及其亚硝酸盐还原酶的表征[D]. 广州: 华南理工大学, 2014.
[16] 王盼. 植物乳杆菌DMDL9010中亚硝酸盐还原酶的克隆表达、纯化及酶学性质[D]. 广州: 华南理工大学, 2015.
[17] 丁少南. 植物乳杆菌中亚硝酸还原酶的研究[D]. 上海: 上海师范大学, 2013.
[18] 吕玉涛, 管世敏, 龚钢明. 短乳杆菌NiRs降低食品中亚硝酸盐含量的研究[J]. 食品工业, 2013(2): 144-146.
[19] 萨姆布鲁克. 分子克隆实验指南[M]. 北京: 科学出版社, 1992: 707.
[20] 汪家政, 范明. 蛋白质技术手册[M]. 北京: 科学出版社, 2000: 77-100.
[21] 侯进慧, 王宾, 王富威. 菠萝泛菌木聚糖酶xynB的克隆表达与活性研究[J]. 生物技术, 2013(5): 28-31.
[22] 张卫卫. 弧菌琼胶酶和丝氨酸蛋白酶的筛选、分析以及免疫应用[D].青岛: 中国科学院海洋研究所, 2009: 21-23.
[23] QIAN S, YU H, ZHANG C, et al. Purification and characterization of dioscin-alpha-L-rhamnosidase from pig liver[J]. Journal of General & Applied Microbiology, 2009, 55(3): 191-199. DOI:10.1248/cpb.53.911.
[24] SMET J, DEVREESE B, BEEUMEN J V, et al. Chapter 32—Nondenaturing polyacrylamide gel electrophoresis as a method for studying protein interactions: applications in the analysis of mitochondrial oxidative phosphorylation complexes[J]. Cell Biology,2006, 4: 259-264. DOI:10.1016/B978-012164730-8/50217-3.
[25] ILÍK P, KRCHŇÁK P, TOMEK P, et al. 2-D gel densitometer for high-contrast and selective imaging of chlorophyll-containing protein complexes separated by non-denaturing polyacrylamide gel electrophoresis[J]. Journal of Biochemical & Biophysical Methods,2002, 51(3): 273-281. DOI:10.1016/j.apsusc.2006.12.100.
[26] KUEHN L, MEYER H, RUTSCHMANN M, et al. Identification of photoaきnity labeled insulin receptor proteins by linear polyacrylamide gradient gel electrophoresis under non-denaturing conditions[J]. Febs Letters, 1980, 113(2): 189-192. DOI:10.1016/0014-5793(80)80588-6.
[27] O’REILLY C E, KELLY A L, MURPHY P M, et al. High pressure treatment: applications in cheese manufacture and ripening[J]. Trends in Food Science & Technology, 2001, 12(2): 51-59. DOI:10.1016/S0924-2244(01)00060-7.
[28] PUPPO C, CHAPLEAU N, SPERONI F, et al. Physicochemical modifications of high-pressure-treated soybean protein isolates[J].Journal of Agricultural & Food Chemistry, 2004, 52(6): 1564-1571.DOI:10.1021/jf034813t.
[29] NOJIRI M. Metalloenzymes in denitrification: applications and environmental impacts[M]. USA: Isabel Moura, 2016: 91-113.DOI:10.1039/9781782623762-00091.
[30] SWAMY U, WANG M, TRIPATHY J N, et al. Structure of spinach nitrite reductase: implications for multi-electron reactions by the ironsulfur: siroheme cofactor[J]. Biochemistry, 2014, 44(49): 16054-16063. DOI:10.1021/bi050981y.