鼠伤寒沙门菌(Salmonella typhimurium)是引起急性胃肠炎的主要病原菌之一。根据2015年欧洲食品安全局的年度报告,沙门菌是导致食物中毒的第二大类最常见的肠道致病菌[1]。人类感染沙门菌会引起发烧、肠胃炎、败血症等,这些症患对社会经济和人类健康造成了很大的负面影响[2]。鼠伤寒沙门菌频繁导致食源性疾病爆发与食用家禽产品有关。食源性致病菌在食品加工过程中或感染人体时会面临各种环境胁迫,为适应环境胁迫且提升其存活能力,这些细菌已进化出复杂的响应体系[3]。
RpoS因子作为经典的响应环境应激的重要因子,被广为研究,它在肠杆菌科细菌中影响细菌毒力,参与细菌克服高渗、氧等环境应激[4]。RpoS因子促进食源性致病菌在恶劣环境中的存活率,因此也增加了食源性致病菌在食品中存在的风险[5-6]。有研究表明在大肠杆菌内,RpoS因子除了在稳定期调控基因的表达外,还能在其应对各种环境压力时进行调控,不同的压力条件可能导致特定的sRNAs的转录,诱导RpoS的翻译[7]。当大肠杆菌暴露在较高的渗透压、变化的pH值、高温和抗菌化合物等条件下时,RpoS因子会调控不同类型蛋白质的转录,这些蛋白质包括酶类、膜蛋白、调控蛋白等[8-9]。RpoS因子与沙雷氏菌对高渗透压的耐受性形成也有关[10]。微生物应对渗透压的途径之一就是通过自身合成或从环境中吸收可溶性物质[11]。RpoS因子能指导proP的转录,proP能够运输可溶性物质如甜菜碱,RpoS还可以指导海藻糖的生物合成[12-15]。Shiroda等[16]为了解在渗透压条件下沙门菌中rpoS基因对延滞期的影响,通过比较沙门菌rpoS基因缺失菌株和亲本菌株在渗透压条件下的延滞期,发现rpoS基因的缺失会导致沙门菌的延滞期延长。为了研究RpoS因子对鼠伤寒沙门菌在外界压力条件下的影响,本研究用Red同源重组技术[17]构建rpoS基因缺失菌株SL1344/ΔrpoS,通过比较亲本菌株SL1344和基因缺失菌株SL1344/ΔrpoS在3.5% NaCl(渗透压)、高温、低pH值以及在不同盐质量分数中预先处理不同时间后置于3.5% NaCl中生长情况,对RpoS因子在环境压力下的作用进行深入的了解,从而为沙门菌疾病的预防及治疗提供理论支持。
1 材料与方法
1.1 材料
1.1.1 菌株与质粒
鼠伤寒沙门菌SL1344由英国食品研究所赠送;Red同源重组系统工具pKD3、pKD46和pCP20 武汉淼灵生物科技有限公司。
pKD3含有氯霉素抗性基因,作为模板扩增含有同源臂的PCR打靶片段;pKD46是温敏型质粒,在30 ℃培养时可以正常复制,而高于37 ℃时会自动丢失,在pKD46上还含有受araB启动子调控的exo、bet、gam基因,它们通过L-阿拉伯糖诱导表达,并携带氨苄青霉素抗性基因作为筛选标记。pCP20是一种温敏性复制子,有氨苄青霉素和氯霉素两种抗性,同时包含有FLP重组酶的基因,在42 ℃可以诱导重组酶的表达,特异地识别结合于FRT位点,FRT位点自身发生同源重组,丢掉抗性基因片段,失去一个FRT位点,达到去掉引入的抗性基因片段的目的。
1.1.2 试剂
LB培养基(NaCl 10 g/L、胰蛋白胨10 g/L、酵母浸出粉5 g/L、琼脂粉1.5%,pH值调至7.0±0.2)、最低营养培养基(basic minimal medium,BMM)[18]、L-阿拉伯糖、SOC(super optimal broth)培养基配制参照文献[19],配方中的试剂均购于成都万科实业有限公司;细菌基因组DNA提取试剂盒(离心柱型) 天根科技(北京)有限公司;DNA Marker、2×Taq PCR Master Mix大连宝生物工程有限公司;聚合酶链式反应(polymerase chain reaction,PCR)产物纯化试剂盒 生工生物工程(上海)股份有限公司。
1.2 仪器与设备
Gene Pulser XcellTM电穿孔系统、T100 PCR仪、1645050电泳仪、SYSTEM GelDoc XR+凝胶成像系统 美国Bio-Rad公司;3K15离心机 美国Sigma公司;ZHP-160振荡培养箱 常州国宇仪器制造有限公司;DHP-9162B电热恒温培养箱 上海一恒科学仪器有限公司。
1.3 方法
1.3.1 引物设计与合成
根据沙门菌基因组(NC_016810.1)rpoS基因序列和pKD3质粒上氯霉素抗性基因设计引物C1/C2,扩增含有同源臂的氯霉素抗性片段。S1/S2为rpoS基因敲除后的鉴定引物。引物由成都擎科梓熙生物技术有限公司合成,引物序列(5’-3’)见表1。
表1 PCR引物序列
Table 1 PCR Primers used in this study

注:下划线部分是长度为39 bp的rpoS基因上下游同源区域序列。
1.3.2 沙门菌rpoS基因的敲除
1.3.2.1 PCR打靶片段的制备
以质粒pKD3为模板,C1和C2为引物,进行PCR扩增,反应条件为:98 ℃预变性3 min;98 ℃变性10 s,55 ℃退火15 s,72 ℃延伸60 s,30 个循环;72 ℃延伸10 min。用1.0%的琼脂糖凝胶电泳对PCR产物进行检测,然后用胶回收试剂盒对PCR产物进行纯化回收。
1.3.2.2 感受态细胞的制备和质粒pKD46的电转化
取OD600nm为0.7左右的新鲜培养的SL1344,4 ℃冰浴15min,4 000 r/min离心10min,弃上清液,用无菌ddH2O重悬洗涤菌体3 次,每次4 000 r/min离心10 min,弃上清液;再用体积分数10%的甘油洗涤2 次,每次4 000 r/min离心10 min,弃上清液;沉淀用500 μL的10%甘油重悬保种。取一支制备好的感受态细胞SL1344,加入1 μL pKD46,混匀;电转化(2.3 kV,5 ms)后加入1 mL SOC培养基,混匀后全部转移至1.5 mL离心管中,30 ℃、140 r/min振荡培养2 h,取150 μL涂布到含有氨苄青霉素的LB平板,30 ℃培养24 h,筛选阳性转化子。
1.3.2.3 PCR打靶片段的电转化
取0.5 mL新鲜培养的含有pKD46的SL1344接种于50 mL含有氨苄青霉素的LB液体培养基中,30 ℃、140 r/min振荡培养至OD600nm约为0.2,加L-阿拉伯糖(终浓度30 mmol/L),继续培养至OD600nm约为0.8,用1.3.2.2节的方法制备含有pKD46的感受态细胞SL1344/ pKD46。取一支感受态细胞,加入5 μL的1.3.2.1节中回收的PCR片段进行电转化(2.3 kV,5 ms),转化后的步骤同1.3.2.2节,将重悬菌液涂布到氯霉素的LB平板,30 ℃培养24 h,筛选阳性转化子,并且用引物S1和S2对阳性转化子进行PCR验证。挑取阳性转化子至含氯霉素的LB液体培养基中培养,30 ℃培养OD600nm约为0.8,取200 μL涂布于含氯霉素的LB平板上,42 ℃培养至长出单菌落,挑取单菌落分别划线于LB平板和含氨苄青霉素的LB平板,若在LB平板上生长而在含氨苄青霉素的LB平板上不生长,则pKD46质粒已被消除。
1.3.2.4 抗性基因的消除
用1.3.2.2节的方法制备感受态细胞SL1344/ΔrpoS::Cmlr,取100 μL感受态细胞于1.5 mL预冷的离心管中,加入1 μL的质粒pCP20进行电转化(2.3 kV,5 ms),转化后步骤同1.3.2.2节,取150 μL菌悬液涂布到含氨苄青霉素的LB平板,30 ℃培养48 h,筛选阳性转化子。然后挑取成功的转化子接种含氨苄青霉素的LB液体培养基中,30 ℃培养OD600nm约为0.8,然后将菌液涂布在不含抗生素的LB平板上,42 ℃培养至长出单菌落,将单菌落分别接种含有氨苄青霉素和氯霉素的LB平板上,在平板上均不长的为抗性基因和pCP20成功消除的菌株且rpoS基因也成功敲除。并且用引物S1/S2对rpoS基因敲除的菌株进行PCR验证。将rpoS基因成功敲除的菌株SL1344/ΔrpoS用-80 ℃甘油保存备用。
1.3.3 SL1344和SL1344/ΔrpoS在环境胁迫下的生长
将在-80 ℃甘油保存的SL1344和SL1344/ΔrpoS在LB液体培养基中活化2 次,在BMM培养基中驯化5 次后将菌株分别接种至3.5% NaCl、42℃、pH 4.0的BMM培养基中,分别对这3 种胁迫进行生长研究,初始接种量均为104CFU/mL,间隔合适的时间取1 mL菌液,稀释至适当的浓度梯度,以平板计数法(倾注法)进行生长曲线的测定。实验所得数据用Origin 8.1软件进行统计学分析,并绘制生长曲线。
1.3.4 SL1344和SL1344/ΔrpoS在不同盐质量分数处理不同时间后置于渗透压(3.5% NaCl)中的生长
将驯化好的菌株(接种量106CFU/mL)分别接种至含有0%、3.5%、5% NaCl的BMM培养基中分别刺激0.5、1、3 h后,再分别取3mL接种至含3.5% NaCl的300 mL的BMM培养基中,置于37 ℃条件下培养,间隔合适的时间取1 mL菌液,稀释至适当的浓度梯度,以平板计数法(倾注法)进行生长曲线的测定。实验所得数据用Origin 8.1软件进行统计学分析,并绘制生长曲线。
2 结果与分析
2.1 突变菌株SL1344/ΔrpoS的构建与鉴定
2.1.1 PCR打靶片段的制备
以质粒pKD3为模板,含有rpoS同源臂的C1和C2为引物,进行PCR扩增,产物两侧为39 bp的rpoS基因上、下游同源区序列,中间为氯霉素抗性基因,预期长度为1 133 bp,琼脂糖凝胶电泳鉴定结果见图1a。

图1 缺失菌株SL1344/ΔrpoS的构建与鉴定
Fig. 1 Construction and identification of SL1344/ΔrpoS
2.1.2 SL1344/ΔrpoS::Cmlr菌株的鉴定
用电转化的方式将含rpoS同源臂的氯霉素抗性PCR片段转入含有pKD46的沙门菌SL1344中,42 ℃消除pKD46质粒,再用含有氯霉素的LB平板对阳性重组菌株进行筛选,得到SL1344/ΔrpoS::Cmlr菌株。PCR产物预期长度为1 432 bp,用琼脂糖凝胶电泳进行验证,以S1和S2为引物,结果见图1b。
2.1.3 沙门菌rpoS基因缺失菌株SL1344/ΔrpoS的鉴定
以电转的方式将pCP20转入SL1344/ΔrpoS::Cmlr菌株中,先30℃培养,消除抗性基因,再42 ℃培养,消除质粒pCP20,通过抗性筛选,将遗传稳定的rpoS基因缺失的SL1344菌株保存并命名为SL1344/ΔrpoS。以S1和S2为引物对亲本菌株和基因缺失菌株进行PCR扩增,结果显示rpoS基因成功敲除,SL1344/rpoS菌株成功构建。缺失菌株和亲本菌株的PCR片段预期长度为500 bp和1 300 bp,琼脂糖凝胶电泳鉴定见图1c,泳道1为rpoS基因缺失菌株的PCR鉴定,泳道2为原始菌株的PCR鉴定。
2.2 SL1344和SL1344/ΔrpoS在环境胁迫下的生长
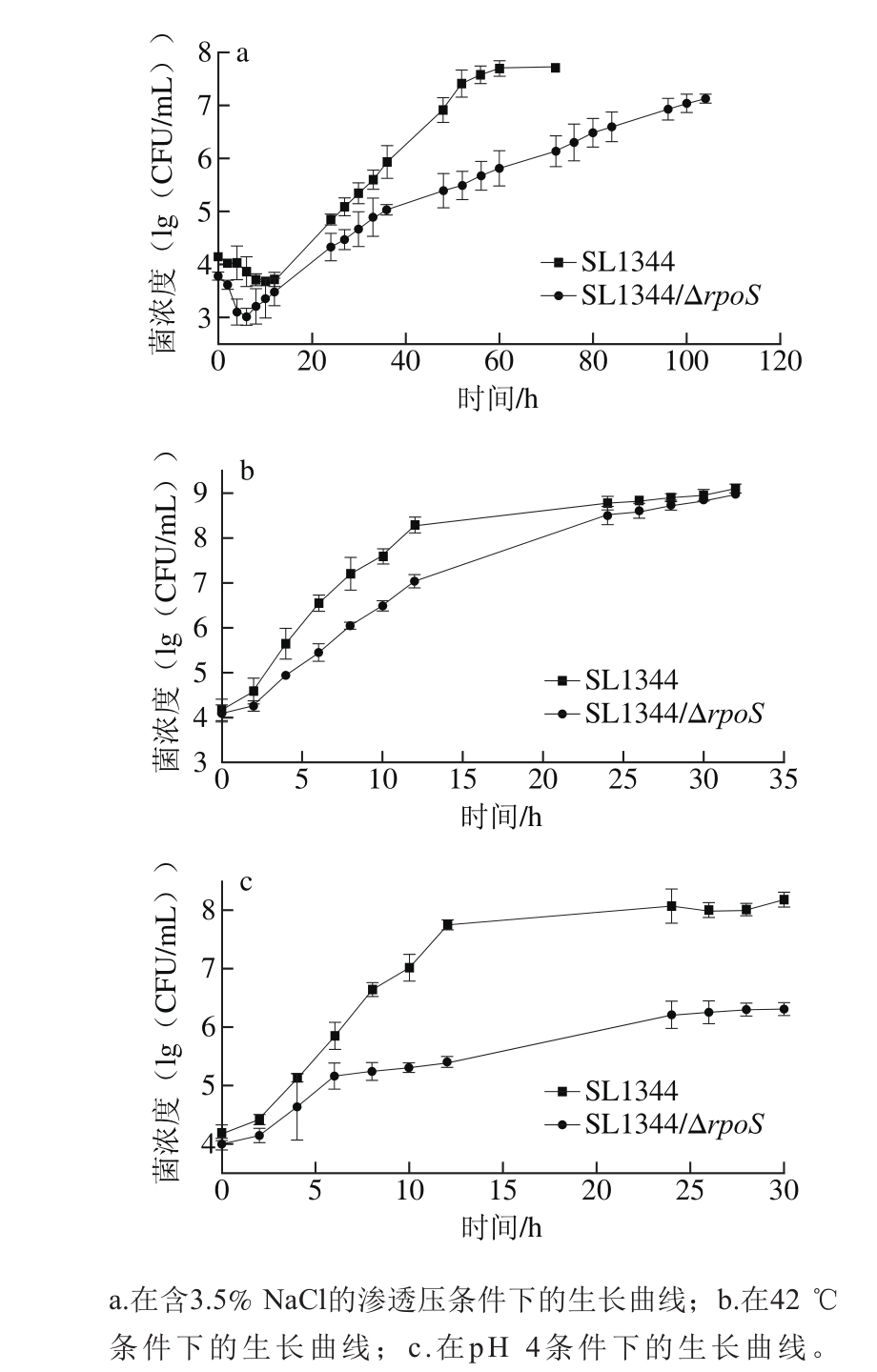
图2 SL1344和SL1344/ΔrpoS在环境胁迫下的生长曲线
Fig. 2 Growth curves of SL1344 and SL1344/ΔrpoS in stressful environments
菌株SL1344和SL1344/ΔrpoS在3.5% NaCl中的生长情况如图2a所示,二者均有延滞期,与亲本菌株SL1344相比,SL1344/ΔrpoS在延滞期的菌浓度下降较快,且在对数期相同时间点,SL1344/ΔrpoS的菌浓度均小于SL1344,SL1344先到达稳定期,通过DMFit(http://browser.combase.cc/DMFit.aspx)软件将二者的生长曲线进行拟合,得到的结果为:SL1344的最大生长速率为SL1344/ΔrpoS的2.49 倍。SL1344和SL1344/ΔrpoS在42 ℃条件下的生长曲线如图2b所示,在对数期的相同时间点,SL1344菌浓度均大于SL1344/ΔrpoS菌浓度,而在稳定期,二者的菌浓度基本相同(109CFU/mL),通过DMFit软件将二者的生长曲线进行拟合,其结果为:SL1344的最大生长速率为SL1344/ΔrpoS的1.33 倍。SL1344和SL1344/ΔrpoS在pH 4.0条件下的生长曲线如图2c所示,二者的生长情况相差较大,在整个生长周期中的相同时间点,SL1344/ΔrpoS的菌浓度一直小于SL1344,SL1344先到达稳定期,且SL1344在稳定期的菌浓度(108CFU/mL)大于SL1344/ΔrpoS菌浓度(106CFU/mL),通过DMFit软件将二者的生长曲线进行拟合,得到的结果为:SL1344的最大生长速率为SL1344/ΔrpoS的2.77 倍。与在3.5% NaCl中的生长相比,SL1344和SL1344/ΔrpoS在42 ℃和pH 4.0条件下到达稳定期的时间均较短。
2.3 SL1344和SL1344/ΔrpoS在不同盐质量分数中处理不同时间后置于3.5% NaCl中的生长
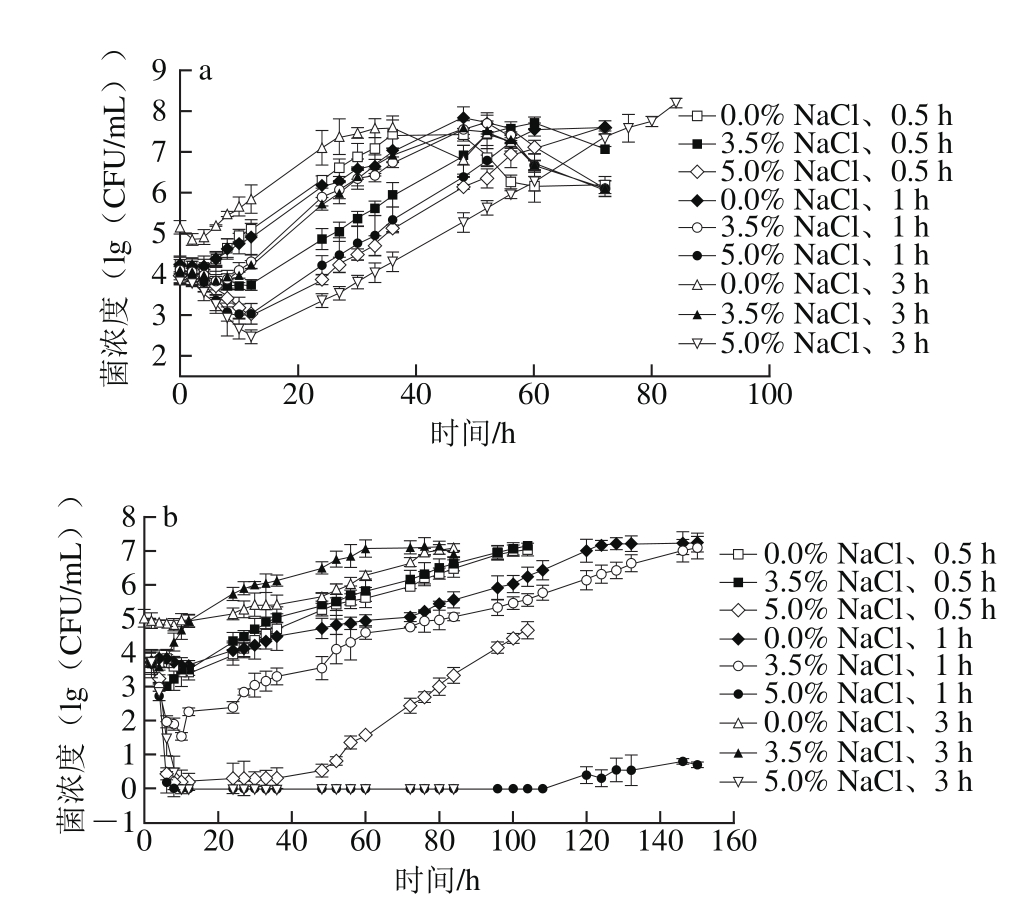
图3 SL1344(a)和SL1344/ΔrpoS(b)在不同盐质量分数中刺激不同时间后置于3.5% NaCl中培养后的生长曲线
Fig. 3 Growth curves of SL1344 (a) and SL1344/ΔrpoS (b) in BMM containing 3.5% NaCl after being stimulated by different concentrations of NaCl for different times
如图3所示,与SL1344和SL1344/ΔrpoS在不加盐的BMM中的生长情况相比,二者的生长均受到抑制,且SL1344/ΔrpoS受到的抑制作用更大。从图3a可以看出,SL1344在不同盐质量分数(0%、3.5%、5% NaCl)中刺激相同时间后的延滞期均为:0%<3.5%<5%;且用不同的盐质量分数处理后,SL1344到达稳定期时的菌浓度均为107~108CFU/mL;SL1344在0% NaCl分别处理0.5、1、3 h后,处理3 h的延滞期最短,最先达到稳定期,在5% NaCl中分别处理0.5、1、3 h后,处理3 h的延滞期最长,到达稳定期的时间也最短。从图3b可以根据培养时间看出,在相同的盐质量分数和时间的处理条件下,SL1344/ΔrpoS到达稳定期的时间均比SL1344长;SL1344/ΔrpoS在0%和3.5% NaCl中处理后,二者的生长趋势一致,在对数期的相同时间点的菌浓度大小均为1 h<0.5 h<3 h;在5% NaCl中刺激不同时间后,SL1344/ΔrpoS的菌浓度迅速下降,特别是刺激3 h之后,SL1344/ΔrpoS菌浓度降至0后基本没有恢复活性;SL1344/ΔrpoS在0%和3.5% NaCl处理0.5 h和3 h之后,在对数后期的相同时间点,3.5% NaCl处理的菌株浓度均超越0% NaCl处理的菌株浓度,而处理1 h的与之相反。
3 讨 论
鼠伤寒沙门菌是十分常见的食源性致病菌。在食品行业中,环境压力会导致微生物中很多受到RpoS因子调控的基因的表达[20]。大肠杆菌K12的转录组情况分析表明其中10%~20%的基因受到RpoS的直接或者间接调控[21-22]。RpoS因子在鼠伤寒沙门氏菌ATCC14028新陈代谢和膜功能方面起着主要的作用,除此之外,RpoS还调控大量sRNA,这些sRNA可能赋予RpoS一些功能,影响mRNA的稳定性或者蛋白稳定性[23-24]。本研究用Red同源重组技术构建rpoS基因缺失菌株SL1344/ΔrpoS,并且在不同条件下对亲本菌株SL1344和基因缺失菌株SL1344/ΔrpoS进行培养,对二者的生长情况进行比较,从而对RpoS因子的作用进行更深入的了解。
在食物中加盐可以提高食物环境的渗透压从而抑制有害微生物的生长,因此盐是保存食物的常见方法。在渗透压条件下,微生物会为了生存而逐渐适应此环境,RpoS为微生物对渗透压的适应提供了条件,通过调控相关基因的表达使其适应渗透压而存活[25-26]。根据本研究的结果,与SL1344和SL1344/ΔrpoS在不加盐的BMM中的生长情况相比,在渗透压(3.5% NaCl)条件下,二者均出现延滞期,稳定期时的菌浓度也比其在不加盐的条件下的菌浓度小(不加盐条件下稳定期菌浓度均为109CFU/mL),且SL1344/ΔrpoS在延滞期菌浓度下降幅度更大,说明渗透压对鼠伤寒沙门菌的生长有抑制,且RpoS因子能增强鼠伤寒对渗透压的适应能力。Zhang Xia等[27]将鼠伤寒沙门菌野生菌株和rpoS基因缺失菌株置于较高渗透压(NaCl终浓度为300 mmol/L)条件下检测其生长情况,结果显示基因缺失菌株的生长明显比野生菌株慢(P<0.05)。此结果与本研究得到的结果一致。鼠伤寒沙门菌的最适生长温度是37 ℃,本研究选择在42 ℃条件下比较亲本菌株和基因缺失菌株的生长情况,发现在42 ℃的高温胁迫条件下,rpoS基因的缺失对鼠伤寒沙门菌在此条件下的最高活菌数的影响较小,虽然SL1344/rpoS在对数期的生长速度比SL1344慢,但到达稳定期时二者的菌浓基本达到一致,说明在42℃条件下,RpoS对鼠伤寒沙门菌的生长影响不大。Yang Yishan等[28]通过对不同温度(10、25、37、42 ℃)条件下培养的肠炎沙门菌中rpoH表达量的检测,结果发现在42 ℃时rpoH表达量最高。在高温条件下,RpoH通过调控热击蛋白的转录来保护细胞免受胞质热应力作用的影响[29]。因此,鼠伤寒沙门菌在较高温度条件下的存活,起主要作用的可能是RpoH,而不是RpoS。鼠伤寒沙门菌生长的最适pH值为7.0,本研究在pH 4.0的条件下比较SL1344和SL1344/ΔrpoS的生长情况,发现在此条件下,处于对数期的SL1344/ΔrpoS生长能力明显弱于SL1344,稳定期时二者菌浓相差较大(分别为108CFU/mL和106CFU/mL),暗示RpoS因子在pH 4.0条件下对鼠伤寒沙门菌的适应能力起到一定的作用,且rpoS基因的缺失会导致沙门菌在此条件下生存能力下降。Burin等[30]将沙门菌置于在pH 5.0和pH 6.0条件下培养,检测rpoS基因表达量,发现沙门菌对酸性条件的适应与rpoS基因的表达有关。与鼠伤寒沙门菌SL1344在42 ℃和pH 4.0条件下的生长情况相比,在渗透压(3.5% NaCl)条件下,SL1344到达稳定期所需要的时间最长,证明在这3 种条件下,渗透压(3.5% NaCl)对其抑制作用最大。此外,国内有学者也就肠炎沙门菌rpoS基因缺陷株在不同环境中进行了综合分析[31],得到了与本研究类似的结果。
为了解鼠伤寒沙门菌在不同渗透压中处理不同时间后的生长情况,本研究将SL1344和SL1344/ΔrpoS于0%、3.5%、5% NaCl中分别处理0.5、1、3 h后置于3.5% NaCl培养(图3)。SL1344在3 种盐质量分数中处理同一特定时间后的延滞期均为:0%<3.5%<5%,表明延滞期与盐质量分数相关,随着盐质量分数增加,延滞期延长;在0% NaCl中处理后,由于生长初期没有盐的刺激,SL1344的生长不受抑制,生长较快,即使处理后置于3.5% NaCl中培养,菌浓度已上升,对渗透压环境的适应能力较强,因此延滞期较短,而在3.5% NaCl中处理后,由于SL1344的初期生长受到限制,所以生长缓慢,再置于3.5% NaCl中培养时,菌可能仍然在适应过程中,因此延滞期较长,同样,在5% NaCl中处理后,菌生长初期受到的限制作用更强,因此生长更慢,延滞期更长;rpoS基因缺失菌株SL1344/ΔrpoS在0%和3.5% NaCl中处理同一特定时间后的生长情况相似,均为处理3 h的先到达稳定期,处理0.5 h的次之,处理1 h的最后达到稳定期,到达稳定期的先后顺序并不是按照处理时间的长短依次上升或递减,表明rpoS基因的缺失会抑制SL1344/ΔrpoS的生长,同时也暗示了rpoS基因的缺失会可能会导致SL1344/ΔrpoS对渗透压的耐受时间有一定的阈值,大于或小于这个时间,SL1344/ΔrpoS受到的抑制作用都会减小。SL1344/ΔrpoS在5% NaCl中处理后,菌浓度迅速下降,与SL1344相比,其对5% NaCl的耐受性明显下降,并且随着刺激时间的延长,SL1344/ΔrpoS在3.5% NaCl中的生长能力越来越弱,说明RpoS因子在鼠伤寒沙门菌面对较高渗透压时对其自身起到一定的保护作用。
本研究通过对沙门菌亲本菌株SL1344和基因缺失菌株SL1344/ΔrpoS在渗透压(3.5% NaCl)、pH 4.0、42 ℃条件下的生长情况对比,发现在这些条件下,鼠伤寒沙门菌受到的抑制作用依次减小。在相同条件下,rpoS基因缺失菌株SL1344/ΔrpoS的生长情况均弱于亲本菌株SL1344;且SL1344和SL1344/ΔrpoS在不同盐质量分数中处理特定时间后的生长情况也存在差异。这些情况为进一步了解RpoS因子在各种环境胁迫下的作用机制提供帮助,同时也为提供更加有效的预防和控制沙门菌疾病措施提供理论支持。
参考文献:
[1] ZADERNOWSKA A, CHAJĘCKA-WIERZCHOWSKA W.Prevalence, biofilm formation and virulence markers of Salmonella sp. and Yersinia enterocolitica, in food of animal origin in Poland[J].LWT-Food Science and Technology, 2017, 75: 552-556. DOI:10.1016/j.lwt.2016.10.007.
[2] PARK H C, BAIG I A, LEE S C, et al. Development of ssDNA aptamers for the sensitive detection of Salmonella typhimurium and Salmonella enteritidis[J]. Applied Biochemistry and Biotechnology,2014, 174(2): 793-802. DOI:10.1007/s12010-014-1103-z.
[3] TAN H J, LIU S H, OLIVER J D, et al. Role of RpoS in the susceptibility of low salinity-adapted Vibrio vulnificus to environmental stresses[J]. International Journal of Food Microbiology,2010, 137(2/3): 137-142. DOI:10.1016/j.ijfoodmicro.2009.12.006.
[4] DONG T, SCHELLHORN H E. Role of RpoS in virulence of pathogens[J]. Infection & Immunity, 2010, 78(3): 887-897.DOI:10.1128/IAI.00882-09.
[5] DODD C E, ALDSWORTH T G. The importance of RpoS in the survival of bacteria through food processing[J]. International Journal of Food Microbiology, 2002, 74(3): 189-194. DOI:10.1016/S0168-1605(01)00679-1.[6] VASUDEVAN P, VENKITANARAYANAN K. Role of the rpoS gene in the survival of Vibrio parahaemolyticus in artificial seawater and fish homogenate[J]. Journal of Food Protection, 2006, 69(6):1438-1442. DOI:10.4315/0362-028X-69.6.1438.
[7] BATTESTI A, MAJDALANI N, GOTTESMAN S. The RpoS-mediated general stress response in Escherichia coli[J]. Annual Review of Microbiology, 2011, 65(65): 189-213. DOI:10.1146/annurev-micro-090110-102946.
[8] AITOUAZZOU A, CHERRAT L, ESPINA L, et al. The antimicrobial activity of hydrophobic essential oil constituents acting alone or in combined processes of food preservation[J]. Innovative Food Science &Emerging Technologies, 2011, 12(3): 320-329. DOI:10.1016/j.ifset.2011.04.004.
[9] SOMOLINOS M, GARCÍA D, MAÑAS P, et al. Effect of environmental factors and cell physiological state on pulsed electric fields resistance and repair capacity of various strains of Escherichia coli[J]. International Journal of Food Microbiology, 2008, 124(3): 260-267. DOI:10.1016/j.ijfoodmicro.2008.03.021.
[10] LIU X, WU Y, CHEN Y, et al. RpoS diあerentially aあects the general stress response and biofilm formation in the endophytic Serratia plymuthica G3[J]. Research in Microbiology, 2015, 167(3): 168-177.DOI:10.1016/j.resmic.2015.11.003.
[11] O’BYRNE C P, BOOTH I R. Osmoregulation and its importance to food-borne microorganisms[J]. International Journal of Food Microbiology, 2002, 74(3): 203-216. DOI:10.1016/S0168-1605(01)00681-X.
[12] MELLIES J, WISE A, VILLAREJO M. Two different Escherichia coli proP promoters respond to osmotic and growth phase signals[J].Journal of Bacteriology, 1995, 177(1): 144-151. DOI:10.1128/jb.177.1.144-151.1995.
[13] IBANEZRUIZ M, ROBBESAULE V, HERMANT D, et al.Identification of RpoS (sigma(S))-regulated genes in Salmonella enterica serovar Typhimurium[J]. Journal of Bacteriology, 2000,182(20): 5749-5756. DOI:10.1128/JB.182.20.5749-5756.2000.
[14] VIJAYAKUMAR S R V, KIRCHHOF M G, PATTEN C L, et al.RpoS-Regulated genes of Escherichia coli identified by random lacZ fusion mutagenesis[J]. Journal of Bacteriology, 2005, 186(24):8499-8507. DOI:10.1128/JB.186.24.8499-8507.2004.
[15] DONG T, SCHELLHORN H E. Global effect of RpoS on gene expression in pathogenic Escherichia coli, O157:H7 strain EDL933[J].BMC Genomics, 2009, 10(349): 1-17. DOI:10.1186/1471-2164-10-349.
[16] SHIRODA M, PRATT Z L, DÖPFER D, et al. RpoS impacts the lag phase of Salmonella enterica during osmotic stress[J]. Fems Microbiology Letters, 2014, 357(2): 195-200. DOI:10.1111/1574-6968.12523.
[17] 吕沈聪, 赵颖颖, 钟卫鸿. Red同源重组在大肠杆菌基因敲除中的应用[J]. 化学与生物工程, 2013, 30(6): 1-6. DOI:10.3969/j.issn.1672-5425.2013.06.001.
[18] ZHOU K, GEORGE S M, MÉTRIS A, et al. Lag phase of Salmonella enterica under osmotic stress conditions[J]. Applied & Environmental Microbiology, 2011, 77(5): 1758-1762. DOI:10.1128/AEM.02629-10.
[19] 许彤, 刘钦松, 张丛丛, 等. 高效JM109感受态细胞制备及转化条件的优化[J]. 湖北农业科学, 2012, 51(21): 4902-4904. DOI:10.3969/j.issn.0439-8114.2012.21.060.
[20] KOCHARUNCHITT C, KING T, GOBIUS K, et al. Global genome response of Escherichia coli O157:H7 Sakai during dynamic changes in growth kinetics induced by an abrupt downshift in water activity[J].PLoS ONE, 2014, 9(3). DOI:10.1371/journal.pone.0090422.
[21] PATTEN C L, KIRCHHOF M G, SCHERTZBERG M R, et al.Microarray analysis of RpoS-mediated gene expression in Escherichia coli K-12[J]. Molecular Genetics & Genomics, 2004, 272(5): 580-591.DOI:10.1007/s00438-004-1089-2.
[22] WEBER H, POLEN T, HEUVELING J, et al. Genome-wide analysis of the general stress response network in Escherichia coli: sigmaS-dependent genes, promoters, and sigma factor selectivity[J]. Journal of Bacteriology, 2005, 187(5): 1591-1603. DOI:10.1128/JB.187.5.1591-1603.2005.
[23] LÉVIMEYRUEIS C, MONTEIL V, SISMEIRO O, et al. Expanding the RpoS/σS-network by RNA sequencing and identification of σS-controlled small RNAs in Salmonella[J]. PLoS ONE, 2014, 9(5):e96918. DOI:10.1371/journal.pone.0096918.
[24] LÉVIMEYRUEIS C, MONTEIL V, SISMEIRO O, et al. Repressor activity of the RpoS/σS-dependent RNA polymerase requires DNA binding[J]. Nucleic Acids Research, 2015, 43(3): 1456-1468.DOI:10.1093/nar/gku1379.
[25] MÉTRIS A, GEORGE S M, ROPERS D. Piecewise linear approximations to model the dynamics of adaptation to osmotic stress by food-borne pathogens[J]. International Journal of Food Microbiology,2016, 240: 63-74. DOI:10.1016/j.ijfoodmicro.2016.06.022.
[26] HAMNER S, MCINNERNEY K, WILLIAMSON K, et al. Bile salts aあect expression of Escherichia coli O157: H7 genes for virulence and iron acquisition, and promote growth under iron limiting conditions[J].PLoS ONE, 2013, 8(9): e74647. DOI:10.1371/journal.pone.0074647.
[27] ZHANG X, ZHU C W, YIN J, et al. RpoS affects gene expression in Salmonella enterica serovar Typhi under early hyperosmotic stress[J].Current Microbiology, 2017, 74(6): 1-5. DOI:10.1007/s00284-017-1243-9.
[28] YANG Y S, KHOO W J, ZHENG Q, et al. Growth temperature alters Salmonella Enteritidi sheat/acid resistance, membrane lipid composition and stress/virulence related gene expression[J].International Journal of Food Microbiology, 2014, 172(1): 102-109.DOI:10.1016/j.ijfoodmicro.2013.12.006.
[29] SPECTOR M P, KENYON W J. Resistance and survival strategies of Salmonella enterica, to environmental stresses[J]. Food Research International, 2012, 45: 455-481. DOI:10.1016/j.foodres.2011.06.056.
[30] BURIN R C K, JR A S, NERO L A. Influence of lactic acid and acetic acid on Salmonella, spp. growth and expression of acid tolerancerelated genes[J]. Food Research International, 2014, 64: 726-732.DOI:10.1016/j.foodres.2014.08.019.
[31] 曾红亮, 曾庆梅, 乔向欣, 等. 肠炎沙门氏菌rpoS基因缺陷株的构建及其生长特性观察[J]. 中国病原生物学杂志, 2013(8): 690-694.DOI:10.13350/j.cjpb.2013.08.001.
