鲜切果蔬又称最少加工果蔬、半加工果蔬、轻度加工果蔬等,它是指以新鲜果蔬为原料,经分级、清洗、整修、去皮、切分、保鲜、包装等一系列处理后,再经过低温运输进入冷柜销售的即食或即用果蔬制品。鲜切果蔬既保持了果蔬原有的新鲜状态,又经过加工使产品清洁卫生,属于净菜范畴。天然、营养、新鲜、方便以及可利用度高(100%可食用),满足了人们追求快节奏,健康等生活方式的需求[1]。目前,在各大超市中,鲜切果蔬制品随处可见,但是,由于其缺少果皮的保护,果肉中的营养成分以及活性成分直接暴露于空气中或是与空气间接接触,会与氧气发生生理生化反应,导致细胞较易氧化、易褐变及易受微生物污染[2]。
据世界卫生组织统计,世界每年食源性病例多达数十亿,其中有220万 人因此而丧生[3]。已将大肠杆菌O157:H7、鼠伤寒沙门菌、单核细胞性李斯特菌及金黄色葡萄球菌列为重要的食源性致病菌[4-5],并且列为中国进出口产品的必检项目。在我国每年发生的细菌性食物中毒占中毒事件的30%~90%,中毒人数占食物中毒总人数的60%~90%[6-7]。调查表明,单核细胞性李斯特菌可以在鲜切哈密瓜、鲜切西瓜、鲜切木瓜[8]、鲜切芹菜[9]以及9 种不同类型的鲜切蔬菜,如莴苣、菠菜、豆苗、芝麻菜、胡萝卜、沙拉等[10]上繁殖。葡萄球菌食源性疾病是金黄色葡萄球菌肠毒素引起食物中毒的最常见的食源性疾病之一。金黄色葡萄球菌同样能够在鲜切火龙果、鲜切芒果、鲜切木瓜上快速繁殖[11]。大肠杆菌O157:H7和沙门菌引起病原微生物食物中毒事件约占微生物引起中毒事件总额的42.9%及34.3%[12]。
目前,常规国标法病原微生物检测手段存在操作步骤繁琐,耗时长等缺点,难以满足市场需求。因此,建立能够对鲜切果蔬中多种病原微生物同时快速检测的技术显得尤为重要,多重聚合酶链式反应(polymerase chain reaction,PCR)技术作为一种能够快速、准确检测多种病原微生物的技术已引起越来越多的重视[13-14]。本实验在建立一共能够同时、快速检测鲜切果蔬中常见病原微生物多重PCR检测方法。对促进鲜切果蔬产业的发展及保障人类生命安全具有重要意义[15]。
1 材料与方法
1.1 材料与试剂
莴苣、黄瓜、木瓜、哈密瓜购自大连市华润万家超市。
鼠伤寒沙门菌(Salmonella typhimurium)(CICC 21484)、金黄色葡萄球菌(Staphylococcus aureus)(CICC 23656)、大肠杆菌O157:H7(Escherichia coli O157:H7)(CICC 10907)、单核细胞性李斯特菌(Listeria monocytogenes)(CICC 21633)、伊氏李斯特菌(L. ivanovii)(CICC 21663)、格氏李斯特菌(L. grayi)(CICC 21670)、斯氏李斯特菌(L. seeligeri)(CICC 21671)、威氏李斯特菌(L. welshimeri)(CICC 21672)、英诺克李斯特菌(L. innocua)(ATCC 10417)、肠侵袭性大肠杆菌(E. coli EIEC)(CICC 10661)、乙型副伤寒沙门菌(S. paratyphi β)(CICC 10437)、肠道沙门菌肠道亚种伤寒血清型(S. enterica subsp. enterica serovar Typhi)(CICC 10871)、奇异变形杆菌(Proteus mirabilis)(CICC 21516)、蜡样芽孢杆菌(Bacillus cereus)(CICC 23832)、大肠埃希氏菌(E. coli)(CICC 10389)、志贺毒性大肠埃希氏菌(E. coli STEC)(CICC 10668)、副溶血性弧菌(V. parahemolyticus)(CICC 21617)、霍乱弧菌(Vibrio cholera)(CICC 23794)、铜绿色假单胞菌(Pseudomonas aeruginosa)(CICC 20236)、空肠弯曲菌(Campylobacter jejuni)(CICC 22936)、福氏志贺菌(Shigella flexneri)(CICC 10865)、宋内志贺菌(S. sonnei)(CICC 21679)、荧光假单胞菌(P. fluorescens)(CICC 20225)、枯草芽孢杆菌(B. subtilis)(CICC 10002) 中国工业微生物菌种保藏管理中心(China Center of Industrial Culture Collection,CICC)。
10×PCR Buffer (Mg2+free)、MgCl2(25 mmol/L)、dNTP Mixture(2.5 mmol/L)、RNase-Free Water、exTaq DNA Polymerase(5 U/μL)、6×DNA loading Buffer、DL1000 DNA Marker 大连宝生物有限公司;琼脂糖西班牙Biowest公司;GelRed染料 上海拜力生物科技有限公司。
1.2 仪器与设备
BagMixer-400W均质器 法国Interscience公司;Scientific Arktik Thermo热循环仪 美国Thermo Fisher Scientific公司;PCR洁净工作台 苏州苏洁净化设备有限公司;D1008E型旋涡混合器 江苏天翎仪器有限公司;BioSpectrum 310凝胶成像系统 美国UVP公司。
1.3 方法
1.3.1 菌悬液制备
将实验室保存的单核细胞性李斯特菌、鼠伤寒沙门菌、金黄色葡萄球菌和大肠杆菌O157:H7分别接种于TSA固体培养基中,36 ℃培养24 h,分别挑取单菌落接种于5 mL TSB养基中,36 ℃静止培养18~24 h。取1 mL菌悬液(约为108CFU/mL)进行DNA提取。
1.3.2 DNA提取
纯培养菌液1 mL于离心管中,12 000 r/min离心2 min,弃上清液,按照细菌基因组DNA提取试剂盒操作方法进行提取。提取的模板储备液通过Thermo酶标仪得到OD260nm、OD280nm以确定提取的DNA浓度及纯度。
1.3.3 引物设计
利用Premier 5.0和Oligo 6.0软件对目的基因进行引物设计以及评价[16],选取出4 对不同片段大小及相近退火温度的特异性引物,经过BLAST分别将引物与目的基因同源性进行比对,确定引物序列[17-18]见表1。
表1 多重PCR引物序列
Table 1 Primer pairs designed for multiplex PCR

1.3.4 多重PCR引物浓度优化
多重PCR总体积为25 μL,其中终浓度分别为1×Ex Taq Buffer、MgCl22 mmol/L、dNTPs Mixture 0.2 mmol/L、exTaq酶1 U/μL,引物浓度按照表2进行四因素四水平正交优化。将鼠伤寒沙门菌、金黄色葡萄球菌、大肠杆菌O157:H7和单核细胞性李斯特菌的DNA等比例混合后,加入4 μL作为模板;每组实验加ddH2O补足至25 μL。反应条件:95 ℃预变性3 min;94 ℃变性30 s,56 ℃复性30 s,72 ℃延伸30 s,40 个循环;72 ℃后延伸10 min。扩增产物检测:经3 mg/100 mL的琼脂糖凝胶电泳后,采用凝胶成像分析系统观察结果。将扩增产物送到宝生物工程有限公司进行测序。
表2 四因素四水平L16(44)正交试验设计
Table 2 Orthogonal array design L16(44)

1.3.5 多重PCR反应液浓度优化[19]
多重PCR总体积为25 μL,其中终浓度分别为1×Ex Taq Buffer;其他反应液成分按照表3进行三因素三水平正交试验优化,影响因素分别为MgCl2(25 mmol/L),优化水平2.0、2.5、3.0 mmol/L;dNTPs Mixture(2.5 mmol/L),优化水平0.15、0.20、0.25 mmol/L;exTaq酶(5 U/μL),优化水平0.5、1.0、1.5 U。
表3 三因素三水平L9(33)正交试验设计
Table 3 Orthogonal array design L9(33)

反应条件:95 ℃预变性3 min;94 ℃变性30 s,56 ℃复性30 s,72 ℃延伸30 s,40 个循环;72 ℃后延伸10 min。扩增产物检测:经质量浓度为3 g/100 mL的琼脂糖凝胶电泳后,采用凝胶成像分析系统观察结果。将扩增产物送宝生物工程(上海)有限公司进行测序。
1.3.6 多重PCR退火温度优化
多重PCR反应总体积为25 μL,包括1×Ex Taq Buffer;MgCl2(25 mmol/L)、dNTPs Mixture (2.5 mmol/L)、exTaq酶(5 U/μL)、引物浓度均为优化后结果,将鼠伤寒沙门菌、金黄色葡萄球菌、大肠杆菌O157:H7和单核细胞性李斯特菌的DNA等比例混合后,加入4 μL作为模板,每组实验加ddH2O补足至25 μL。反应条件:95 ℃预变性3 min;94 ℃变性30 s,退火温度的优化分别为52.0、52.2、52.6、53.2、54.1、55.2、56.6、57.8、58.6、59.4、59.8、60.0 ℃复性30 s,72 ℃延伸30 s,32 个循环;72 ℃后延伸10 min;每组实验加ddH2O 补足至25 μL。扩增产物检测:经质量浓度为3 g/100 mL的琼脂糖凝胶电泳后,采用凝胶成像分析系统观察结果。将扩增产物送到宝生物工程(上海)有限公司进行测序。
1.3.7 多重PCR延伸时间优化
多重PCR总体积为25 μL,包括1×Ex Taq Buffer、MgCl2(25 mmol/L)、dNTPs Mixture(2.5 mmol/L)、exTaq酶(5 U/μL)、引物浓度均为优化后结果,将鼠伤寒沙门菌、金黄色葡萄球菌、大肠杆菌O157:H7和单核细胞性李斯特菌的DNA等比例混合后,加入4 μL作为模板,每组实验加ddH2O 补足至25 μL。反应条件:95 ℃预变性3 min;94 ℃变性30 s,退火温度为优化后结果,复性30 s,72 ℃延伸时间分别为15、30、45、60、75、90 s,32 个循环;72 ℃后延伸10 min;每组实验加ddH2O补足至25 μL。扩增产物检测:经质量浓度为3 g/100 mL的琼脂糖凝胶电泳后,采用凝胶成像分析系统观察结果。将扩增产物送到宝生物工程(上海)有限公司进行测序。
1.3.8 多重PCR引物间特异性验证
为确定所建立多重PCR方法的特异性,按照多重PCR体系与条件随机组合添加模板DNA,每组按照表4进行扩增反应,根据扩增产物分析有无反应内交叉错配现象[20]。
表4 特异性验证实验
Table 4 Experimental design for validation of PCR specificity

注:+.在反应体系中添加相应的模板;-.在反应体系内不含相应的模板。
1.3.9 多重PCR菌间特异性验证
利用优化得到的多重PCR检测方法对非目标菌DNA模板进行扩增。对建立的反应体系进行特异性验证[21]。选择伊氏李斯特菌、格雷李斯特菌、斯氏李斯特菌等20种非目标菌作为阴性对照,并用ddH2O作为空白对照。
1.3.10 多重PCR检出限检测
将菌悬液用0.1%无菌蛋白胨水10 倍梯度稀释至3(lg(CFU/mL)),菌悬液和每个稀释梯度的菌液各取1 mL,用细菌DNA提取试剂盒对目标菌进行提取,以获得DNA模板,按最佳反应体系与反应条件进行实验。同时对目标菌按照涂布法进行菌落计数。
1.3.11 预富集培养
将4 种目标菌菌悬液用0.1%无菌蛋白胨稀释至100、103、106CFU/mL,以TSB为基础的改良的共增菌培养基,共增菌培养基配方如下:胰蛋白胨17 g/L、蛋白胨3 g/L、氯化钠10 g/L、磷酸氢二钠2.5 g/L、葡萄糖2.5 g/L。预富集培养3、6、9 h,菌悬液各取1 mL,用细菌DNA提取试剂盒对目标菌进行提取,以获得DNA模板,按最佳反应体系与反应条件进行实验。
1.3.12 人工接种鲜切果蔬验证
制备4 种致病菌菌悬液,用0.1%无菌蛋白胨水稀释至10 CFU/mL,取1 mL菌悬液分别侵染至清洗后的鲜切莴苣、鲜切黄瓜、鲜切木瓜、鲜切哈密瓜表面,样品质量约10 g,片状样品规格约0.5 cm×0.5 cm,块状样品规格约0.5 cm×0.5 cm×0.5 cm。在安全柜中吹干1 h,将其分别浸泡于90 mL共增菌培养基中培养至可检出的时间、37 ℃[22]。采用已建立的多重PCR技术对其进行验证,以确定该技术在鲜切果蔬中应用的有效性。
2 结果与分析
2.1 多重PCR引物浓度优化结果
各引物对在多重PCR过程中呈相互抑制的关系,适当的调节引物浓度配比可以有效避免目标条带漏检的现象,是影响多重PCR有效扩增目标条带的关键因素[23]。多重PCR引物浓度优化结果如图1所示,不同比例的引物浓度扩增的结果有所不同,引物浓度用量较低时,会出现目标条带未被扩增的现象。而当引物用量过高时,会导致单一目标条带过于明亮且弥散。当4 种目标菌的引物浓度配比不同时,易出现杂带扩增的现象。由图1中泳道1~3所示,均有杂带扩增的现象。图1b中泳道4和泳道8,由于inlA引物浓度含量较低,因此扩增的相应条带较暗。而在图1b中泳道5~7,均可扩增出4 条清晰、明亮且特异性良好的条带。考虑到条带明亮度的均一性及最大限度的提高扩增效率,则选择泳道7中inv引物浓度为0.4 μmol/L,wzy引物浓度为0.3 μmol/L,inlA引物浓度为0.2 μmol/L,nuc引物浓度为0.4 μmol/L时为最终引物浓度配比进行后续实验。
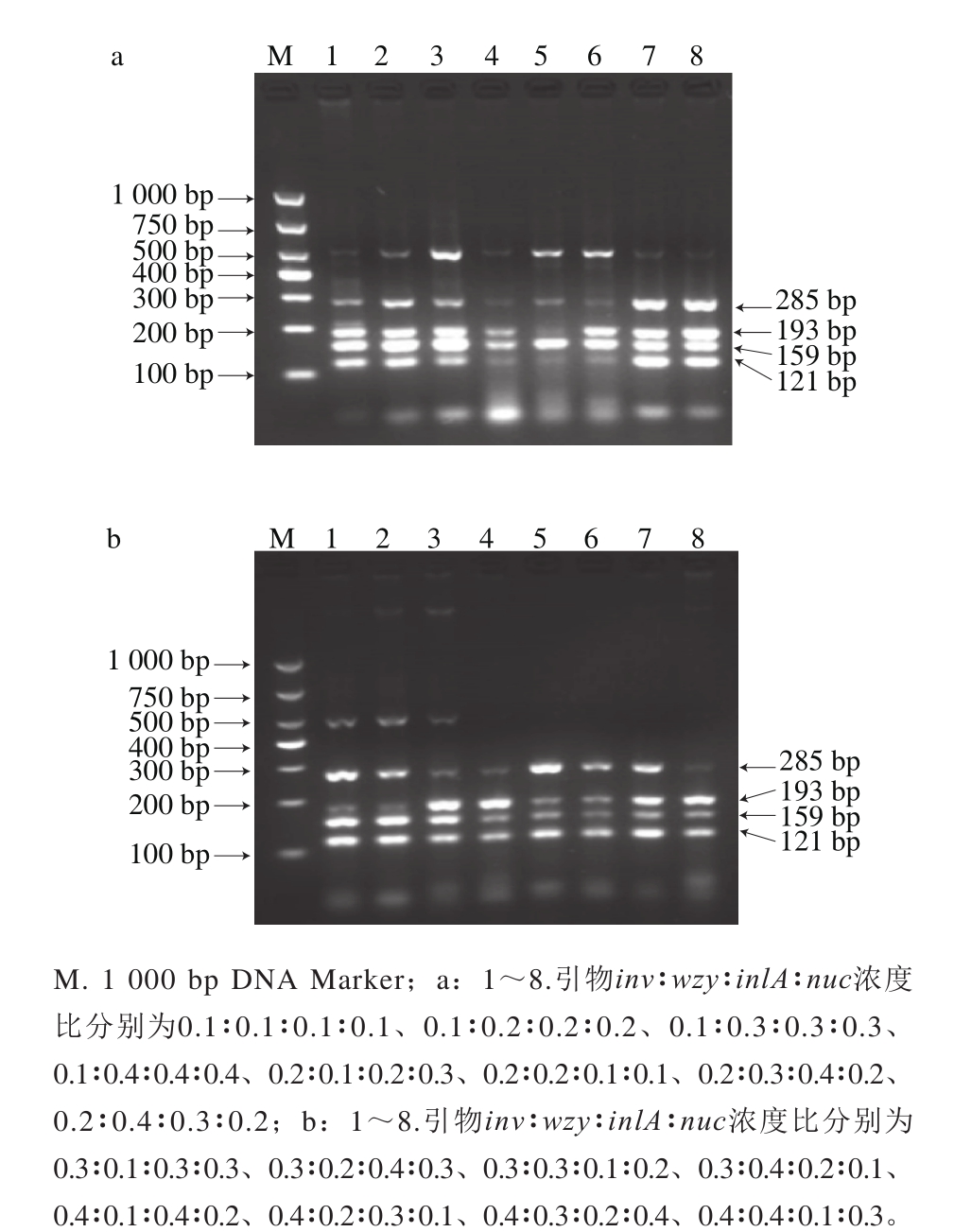
图1 多重PCR引物浓度优化结果
Fig. 1 Optimization of primer concentrations for multiplex PCR
2.2 多重PCR体系优化结果

图2 PCR体系优化结果
Fig. 2 Optimization of reaction system for multiplex PCR
2.3 多重PCR退火温度优化结果
菌nuc引物浓度为0.4 μmol/L时,Mg2+浓度为3.0 mmol/L,dNTP浓度为0.25 mmol/L,exTaq活力为1.0 U时对多重PCR退火温度进行优化,结果如图3所示。
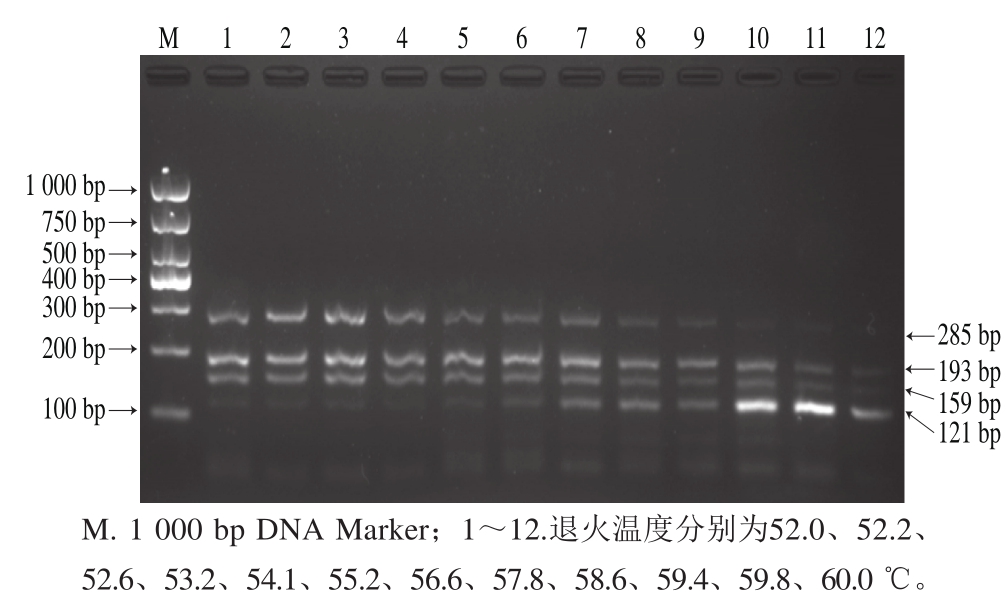
图3 多重PCR退火温度优化结果
Fig. 3 Optimization of annealing temperature for multiplex PCR
在52~60 ℃范围内扩增的目标条带明显不同,当退火温度较低时如泳道1、2,鼠伤寒沙门菌目标条带几乎无法扩增,随着退火温度升高,如图3泳道6~9所示,扩增条带逐渐呈现清晰可见的现象。而当退火温度为58.6、59.4、59.8、60.0 ℃时,单核细胞性李斯特菌inlA基因则呈无法扩增的现象,这是由于退火温度过高导致扩增效率显著降低的结果[25]。综合而言,泳道7中4 条目标条带清晰、明亮,因此选择退火温度为56.6 ℃进行后续实验。
2.4 多重PCR延伸时间优化结果
在退火温度为56.6 ℃条件下,对反应条件中延伸时间优化结果如图4所示,随着延伸时间的延长,扩增效率呈下降趋势。延伸时间在30~60 s时,如图4泳道2~4所示,均可扩增出清晰条带,考虑到缩短扩增时间,则选择泳道2中延伸时间为30 s进行后续扩增。程晓艳等[26]对5 种病原微生物多重PCR延伸时间优化结果同样表明,当延伸时间从1 min增加到2.5 min时多重PCR扩增效率呈现下降趋势。
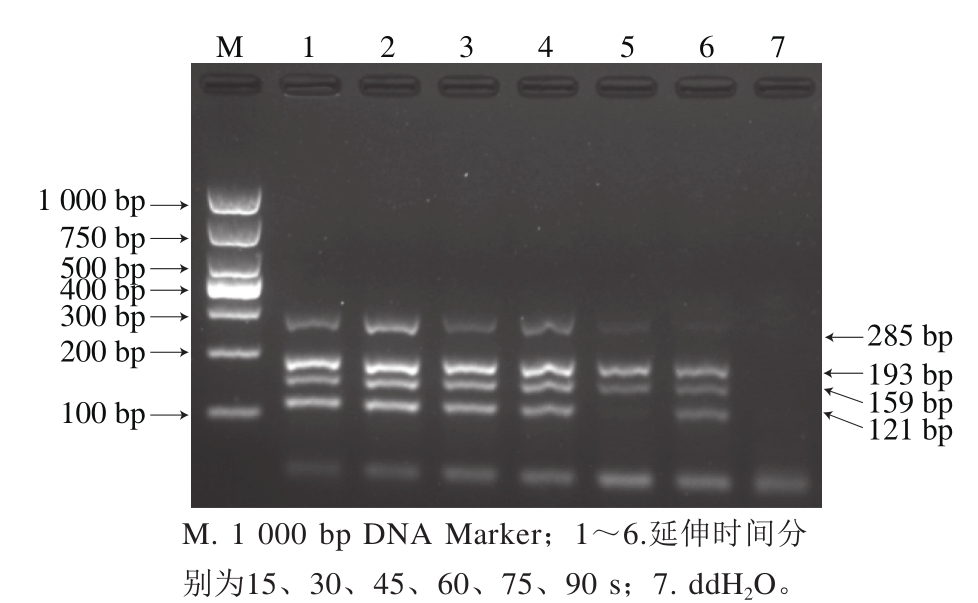
图4 多重PCR延伸时间优化结果
Fig. 4 Optimization of extension time for multiplex PCR
2.5 多重PCR引物间特异性验证
利用已建立的多重PCR体系对目标菌进行引物间特异性的检测,检测结果如图5所示。泳道1~6则可效扩增出两两随机组合的目标病原菌,泳道7~10可分别有效扩增出随机组合的3 种目标病原菌,泳道11中含有4 种目标菌。均无非特异性扩增条带出现。阴性对照无扩增条带。该结果与Germini等[21]建立的大肠杆菌O157:H7、沙门菌属以及单核细胞性李斯特菌多重PCR检测技术引物间特异性验证结果一致。Xu Yigang等[27]同样将弧菌属中的5 种食源性病原微生物随机侵染于海产品中,验证了其建立的多重PCR技术扩增任意目标菌的有效性。

图5 多重PCR引物间特异性验证结果
Fig. 5 specific verification among primers in multiplex PCR
2.6 多重PCR体系中菌间特异性检测
如图6所示,在同一反应管中可同时扩增得4 种目的基因片段,3 g/100 mL的琼脂糖凝胶电泳显示在285、193、159 bp和121 bp处均有条带出现,且没有引物二聚体形成,没有非特异性条带出现。图6泳道2~11为非目标菌扩增结果,与对照组一致无条带扩增,表明其特异性良好。
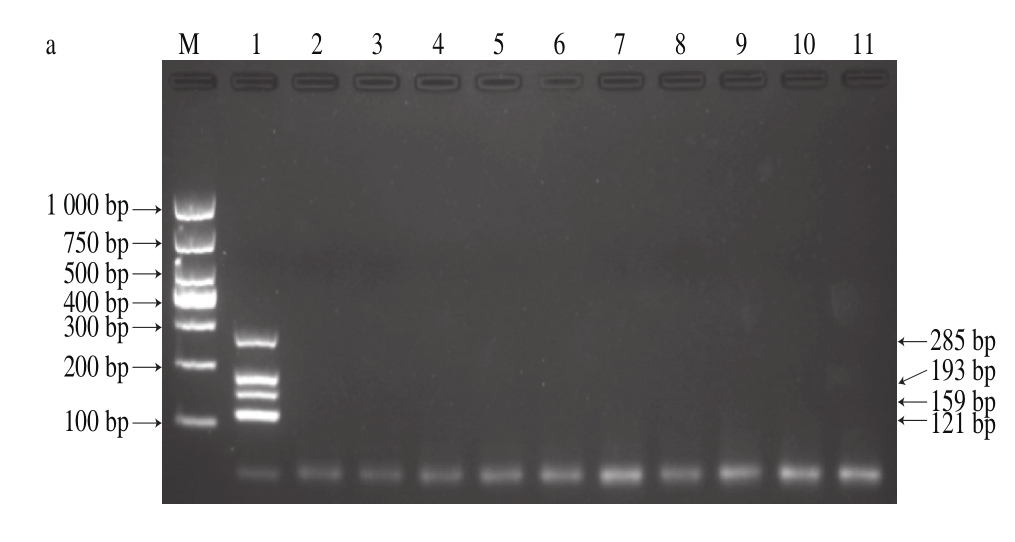
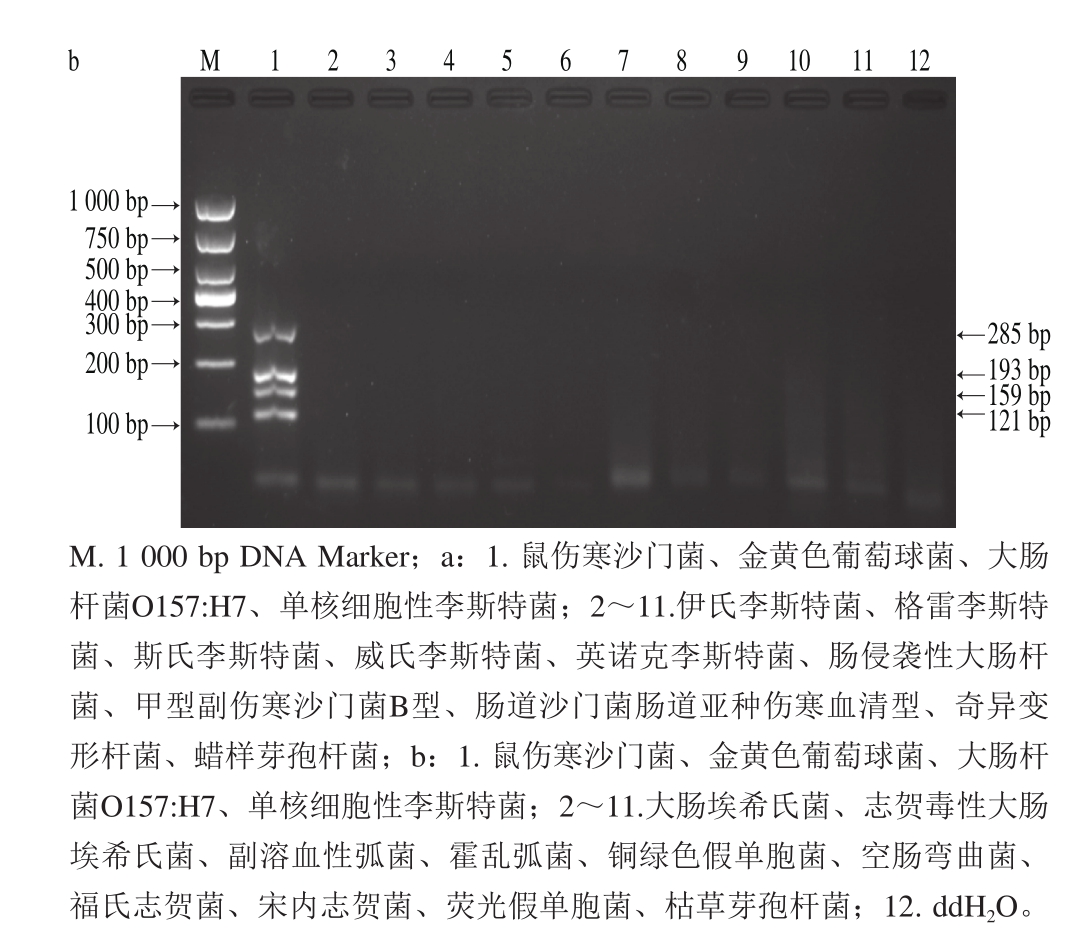
图6 多重PCR菌间特异性验证结果
Fig. 6 Specificity of multiplex PCR
2.7 多重PCR检出限验证结果

图7 多重PCR检出限验证结果
Fig. 7 Detection limits of multiplex PCR
将4 种致病菌的菌悬液进行梯度稀释后,进行菌落数确定,单核细胞性李斯特菌的菌落数为3.5×107CFU/mL,大肠杆菌O157:H7的菌落数为4.8×107CFU/mL,金黄色葡萄球菌的菌落数为2.4×107CFU/mL,鼠伤寒沙门菌的菌落数为1.6×107CFU/mL。分别将稀释后提取的DNA等比例进行混合,用优化好的多重PCR体系进行检测验证,结果如图7所示,4 种目标菌的检测限分别为单核细胞性李斯特菌为3.5×106CFU/mL,大肠杆菌O157:H7为4.8×105CFU/mL,金黄色葡萄球菌为2.4×105CFU/mL,鼠伤寒沙门菌为1.6×105CFU/mL。钱志伟等[29]利用多重PCR技术特异性扩增金黄色葡萄球菌的nuc基因,单核细胞性李斯特菌的prs基因,沙门菌的invA基因,经优化后,其检测水平为3.8、5.1、7.6 pg/μL,该技术可用于牛乳中这3 种菌的检测。陈娟等[22]建立了检测金黄色葡萄球菌、肠炎沙门菌、大肠杆菌O157:H7、单核增生李斯特菌、福氏志贺菌5 种目标菌的多重PCR检测技术,其检出限分别为6.4、32、32、800 pg和160 pg。
2.8 病原微生物预富集多重PCR验证结果
利用已建立的多重PCR方法对不同接种量,单核细胞性李斯特菌(1、3.8×103、3.8×106CFU/mL),大肠杆菌O157∶H7 (1、1.8×103、1.8×106CFU/mL),鼠伤寒沙门菌 (1、5.4×103、5.4×106CFU/mL),金黄色葡萄球菌(1、2.1×103、2.1×106CFU/mL)。在0、3、6、9 h富集后进行验证。如图8所示,泳道1和泳道4所示,1 CFU/mL接种量在0、3 h富集后无条带扩增,泳道10中,在9 h富集后可有效扩增出4 条特异性条带。103CFU/mL接种后,泳道2所示,0 h无扩增条带,富集3 h后,泳道5所示,均可扩增出4 条目标条带,在106CFU/mL接种0 h后,泳道3所示,可扩增出4 条目标条带。综上所述,结果表明,其实接种量低至1 CFU/mL时,富集9 h后均可扩增出4 条目标条带。
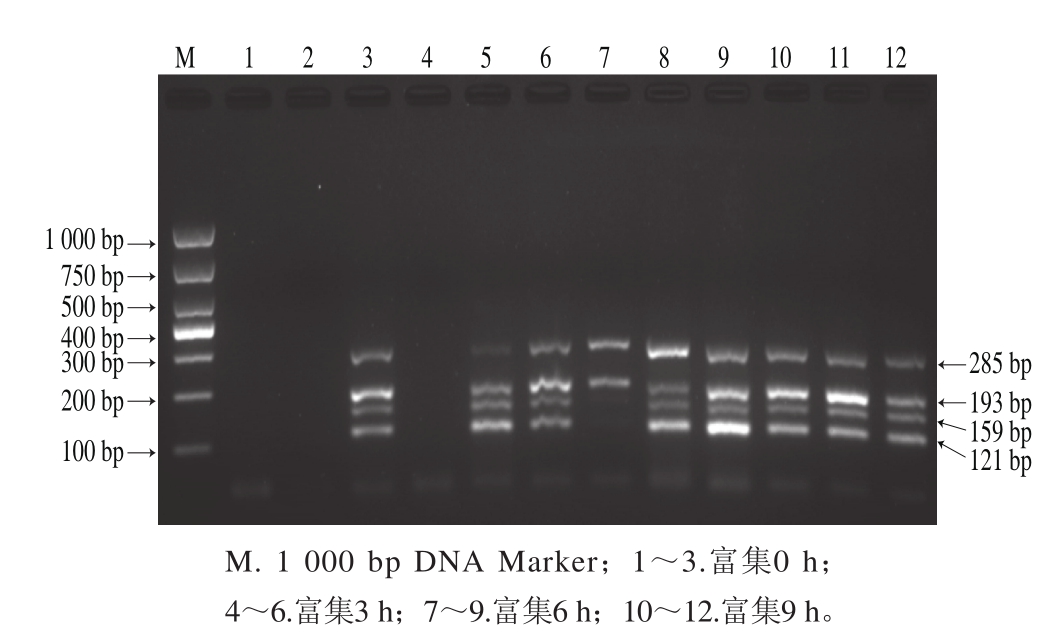
图8 多重PCR预富集检测验证结果
Fig. 8 Detection limits of multiplex PCR with pre-enrichment
2.9 人工污染鲜切果蔬样品验证结果
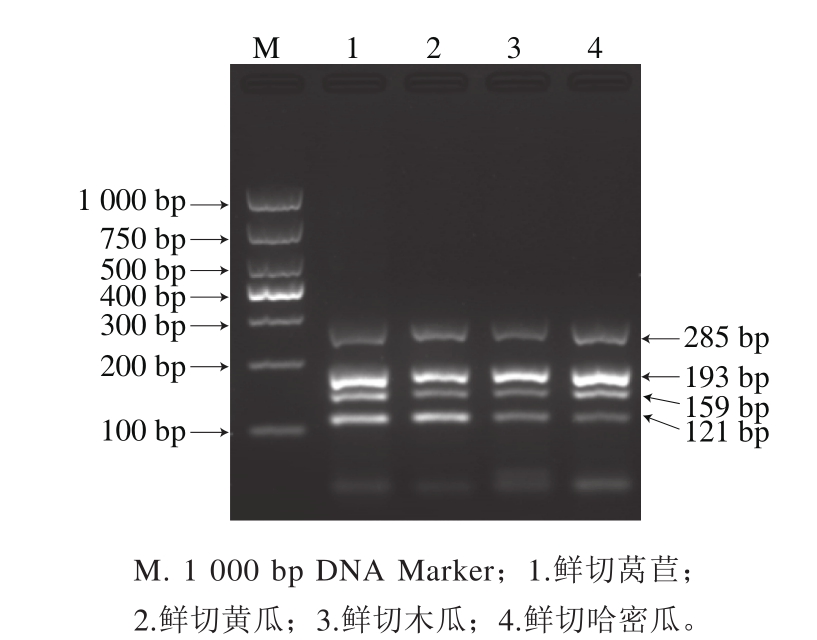
图9 鲜切果蔬样品检测结果
Fig. 9 Multiplex PCR detection of fresh-cut fruit and vegetables
利用已建立的多重PCR方法对染菌量为1 CFU/g的鲜切莴苣、鲜切黄瓜、鲜切木瓜、鲜切哈密瓜,进行富集培养9 h后进行验证。如图9所示,对4 种样品验证均可有效扩增出4 条目标条带,其条带大小与预计大小相符,阴性对照无条带扩增。其中,泳道1~2条带清晰明亮,而泳道3和4中鼠伤寒沙门菌条带较暗,可能是由于鲜切木瓜和鲜切哈密瓜中杂质较多对多重PCR的扩增效率产生了一定的影响。综上所述,鲜切果蔬产品受病原菌侵染后,预富集9 h,利用所建立的多重PCR技术对鲜切果蔬产品的检出限可达到1 CFU/g。
3 结 论
针对单核细胞性李斯特菌inlA基因、鼠伤寒沙门菌invA基因、大肠杆菌O157:H7 wzy基因、金黄色葡萄球菌的nuc基因设计及筛选出4 对引物,引物特异性强,互不干扰。
建立了一种能够同时检测单核细胞性李斯特菌、大肠杆菌O157:H7、鼠伤寒沙门菌、金黄色葡萄球菌4 种致病菌的多重PCR检测方法,其检出限分别为3.5×106、4.8×105、1.6×105、2.4×105CFU/mL。
鲜切莴苣、鲜切黄瓜、鲜切木瓜和鲜切哈密瓜4 种产品受病原菌侵染,预富集9 h后,利用所建立的多重PCR技术对鲜切果蔬产品的检出限可达到1 CFU/g。
方法的建立相较于传统的培养检测方法可节约大量的劳力、试剂、时间等,检测时间由原来的5~7d,缩短至9~11 h,对于企业或分析检验中心大批量样品的监测具有指导意义。
参考文献:
[1] 李超, 冯志宏, 陈会燕, 等. 鲜切果蔬保鲜技术的研究进展[J]. 保鲜与加工, 2010, 10(1): 3-6.
[2] 高雪丽, 高愿军. 鲜切果蔬加工与微生物控制[J]. 食品工程,2006(2): 11-13.
[3] SCALLAN E, HOEKSTRA R M, ANGULO F J, et al. Foodborne illness acquired in the united states major pathogens[J]. Emerging Infectious Diseases, 2011, 17(1): 7-15. DOI:10.3201/eid1701.P11101.
[4] 曾德兴, 黄思思, 陈应坚. 细菌性食物中毒病原菌调查与预防对策分析[J]. 现代诊断与治疗, 2016(8): 1518-1520.
[5] 冯可, 胡文忠, 姜爱丽, 等. 单核细胞增生性李斯特菌分子生物学检测技术的研究进展[J]. 食品工业科技, 2014, 35(4): 392-396.DOI:10.13386/j.issn1002-0306.2014.04.044.
[6] 侯君, 陈玉凤, 栾明春, 等. 2009—2012年大连市食品中食源性致病菌污染状况调查[J]. 医学动物防制, 2015(1): 7-9. DOI:10.7629/yxdwfz201501003.
[7] 崔燕, 梁效成. 2011—2013年甘肃省食源性疾病暴发事件分析[J].疾病预防控制通报, 2015(1): 76-78. DOI:10.13215/j.cnki.jbyfkztb.1408015.
[8] PENTEADO A L, LEITAO M F F. Growth of Listeria monocytogenes in melon, watermelon and papaya pulps[J]. International Journal Food Microbiology, 2004, 92(1): 89-94. DOI:10.1016/j.ijfoodmicro.2003.08.020.
[9] JOSHUA P V, DI L, LINDA J H, et al. Fate of Escherichia coli O157:H7, Listeria monocytogenes, and Salmonella on fresh-cut celery[J]. Food Microbiology, 2013, 34(1): 151-157. DOI:10.1016/j.fm.2012.11.016.
[10] SANT’ANA A S, MATHEUS S B, MARIA T D, et al. Growth potential of Salmonella spp. and Listeria monocytogenes in nine types of ready-to-eat vegetables stored at variable temperature conditions during shelf-life[J]. International Journal Food Microbiology, 2012,157(1): 52-58. DOI:10.1016/j.ijfoodmicro.2012.04.011.
[11] FENG K, HU W Z, JIANG A L, et al. Growth potential of Listeria monocytogenes and Staphylococcus aureus on fresh-cut tropical fruits[J]. Journal of Food Science, 2015, 80(11): M2548-M2554.DOI:10.1111/1750-3841.13089.
[12] NUESCH-INDERBINEN M, STEPHAN R. Fresh fruit and vegetables as vehicles of bacterial foodborne disease: a review and analysis of outbreaks registered by proMED-mail associated with fresh produce[J].Journal of Food Safety and Food Quality-Archiv fur Lebensmittelhygiene,2016, 67(2): 32-39. DOI:10.2376/0003-925X-67-32.
[13] DÍAZ-LÓPEZ A, CANTÚ-RAMÍREZ R C, GARZAGONZÁLEZ E,et al. Prevalence of foodborne pathogens in grilled chicken from street vendors and retail outlets in reynosa, tamaulipas, Mexico[J]. Journal of Food Protection, 2011, 74(8): 1320-1323. DOI:10.4315/0362-028X.JFP-11-014.
[14] 杨柳, 胡文忠, 姜爱丽, 等. 分子生物学方法检测沙门氏菌的研究进展[J]. 食品工业科技, 2016, 37(9): 372-379. DOI:10.13386/j.issn1002-0306.2016.09.065.
[15] CHEN J, TANG J, LIU J, et al. Development and evaluation of a multiplex PCR for simultaneous detection of five foodborne pathogens[J]. Journal of Applied Microbiology, 2012, 112(4): 823-830. DOI:10.1111/j.1365-2672.2012.05240.x.
[16] YAN W. Multiplex PCR primer design for simultaneous detection of multiple pathogens[J]. Methods in Molecular Biology (Clifton, N.J.),2015, 1275: 91-101. DOI:10.1007/978-1-4939-2365-6_6.
[17] 冯可, 胡文忠, 姜爱丽, 等. 多重PCR法检测鲜切哈密瓜中3 种食源性致病菌[J]. 食品科学, 2017, 38(6): 295-302. DOI:10.7506/spkx1002-6630-201706046.
[18] FENG K, HU W Z, JIANG A L, et al. A dual filtration-based multiplex PCR method for simultaneous detection of viable Escherichia coli O157:H7, Listeria monocytogenes and Staphylococcus aureus on fresh-cut cantaloupe[J]. PLoS ONE, 2016, 11(12): e0166874.DOI:10.1371/journal.pone.0166874.
[19] 王如景, 王羽, 李英军, 等. 双正交优化多重PCR检测食源性致病菌的研究[J]. 食品科技, 2012, 37(8): 308-313. DOI:10.13684/j.cnki.spkj.2012.08.071.
[20] KIM J H, RHIM S R, KIM K T, et al. Simultaneous detection of Listeria monocytogenes, Escherichia coli O157:H7, Bacillus cereus,Salmonella spp., and Staphylococcus aureus in low-fatted milk by multiplex PCR[J]. Korean Journal for Food Science of Animal Resources, 2014, 34(5): 717-723. DOI:10.5851/kosfa.2014.34.5.717.
[21] NARI L, KYUNG Y K, SU K O, et al. A multiplex PCR assay for simultaneous detection of Escherichia coli O157:H7, Bacillus cereus,Vibrio parahaemolyticus, Salmonella spp., Listeria monocytogenes,and Staphylococcus aureus in Korean ready-to-eat food[J]. Foodborne Pathogens and Disease, 2014, 11(7): 574-580. DOI:10.1089/fpd.2013.1638.
[22] YU Q, ZHAI L Z, BIE X M, et al. Survey of five food-borne pathogens in commercial cold food dishes and their detection by multiplex PCR[J]. Food Control, 2016, 59: 862-869. DOI:10.1016/j.foodcont.2015.06.027.
[23] 孙鸿燕. 四种食源性致病菌多重PCR检测方法的建立[D]. 长春: 吉林大学, 2011: 37.
[24] 郝玉芹, 孙皆宜, 李艾, 等. 正交优化多重PCR反应体系检测3 种食源性致病菌的研究[J]. 安徽农业科学, 2010, 38(2): 602-605.DOI:10.13989/j.cnki.0517-6611.2010.02.009.
[25] YANG I, KIM Y, BYUN J, et al. Use of multiplex polymerase chain reactions to indicate the accuracy of the annealing temperature of thermal cycling[J]. Analytical Biochemistry, 2005, 338(2): 192-200.DOI:10.1016/j.ab.2004.09.035.
[26] 程晓艳, 刘庆慧, 黄倢. 5 种食源性致病菌多重PCR检测方法的建立[J].渔业科学进展, 2012, 33(2): 97-103.
[27] GERMINI A, MASOLA A, CARNEVALI P, et al. Simultaneous detection of Escherichia coli O175:H7, Salmonella spp. and Listeria monocytogenes by multiplex PCR[J]. Food Control, 2009, 20: 733-738. DOI:10.1016/j.foodcont.2008.09.010.
[28] XU Y G, SUN L M, WANG Y S, et al. Simultaneous detection of Vibrio cholerae, Vibrio alginolyticus, Vibrio parahaemolyticus and Vibrio vulnificus in seafood using dual priming oligonucleotide (DPO)system-based multiplex PCR assay[J]. Food Control, 2017, 71: 64-70.DOI:10.1016/j.foodcont.2016.06.024.
[29] 钱志伟, 孙新城. 食品中3 种致病菌多重PCR检测体系的建立及初步应用[J]. 食品科学, 2011, 32(16): 236-239.
[30] 陈娟, 唐俊妮, 李键, 等. 五种食源性致病菌多重PCR的条件优化和检出限分析[J]. 中国食品卫生杂志, 2014, 26(2): 137-141.DOI:10.13590/j.cjfh.2014.02.008.