基质辅助激光解吸电离质谱成像(matrix-assisted laser desorption mass spectrometry imaging,MALDIMSI)技术是利用MALDI质谱技术,通过质谱仪测定离子的质荷比(m/z)来分析生物分子的标准分子质量,并结合专门的质谱成像软件辅助产生分子图像的新型分析技术。该技术最早于1997年由Caprioli和同事一起提出[1]。同传统的分析技术相比,MALDI-MSI技术无需提取、分离、纯化待测样品,具有检测分子质量范围广、灵敏度高、操作简便、自动化水平高等特点[2]。随着MALDI-MSI技术的发展,其已被广泛应用于生命科学领域,如动植物生理学[3]、病理学[4]、药物研发及疗效监控[5]等。基于MALDI-MSI技术的诸多优点,近几年来其在食品科学领域中也得到了应用[6]。本文主要介绍MALDI-MSI技术样品制备及其在食品分析中的应用,并对其发展方向进行了展望。
1 MALDI-MSI的成像原理
MALDI-MSI技术需要使用冰冻切片机对被研究的组织进行切片处理,获得极薄的组织切片后用基质进行覆盖,然后将干燥结晶后的切片组织置于质谱仪的靶上进行成像分析。该技术的原理是以软电离技术MALDI为基础,将分析物分散在基质分子中并形成晶体。用紫外(ultra-violet,UV)和红外(infrared,IR)的激光束照射晶体时,由于基质分子经辐射所吸收的能量蓄积并迅速产热,基质晶体升华,进而使样品从表面解吸进入气相,基质和分析物发生膨胀,进行气相质子交换反应形成离子[7]。然后利用MALDI系统的质谱成像软件分析样品组织,将质谱仪所获得的样品上每个点的m/z信息转化为照片上的像素点。在每个样品点上,将所有质谱数据经平均化处理获得一幅代表该区域内化合物分布情况的完整质谱图。仪器逐步采集质谱数据,最后得到具有空间信息的整套样品的质谱数据,完成对组织样品的“分子成像”。设定m/z的范围,即可确定该组织区域所含生物分子的种类,选定峰高或者峰面积来代表生物分子的相对丰度[8]。一般通过质谱成像可获取组织切片的二维图谱,在组织发育的过程中,将用连续切片获取的二维图谱组合,可绘出样品组织中靶分子的三维图谱,以分析其空间结构[9]。MALDI-MSI的工作流程如图1所示[10]。
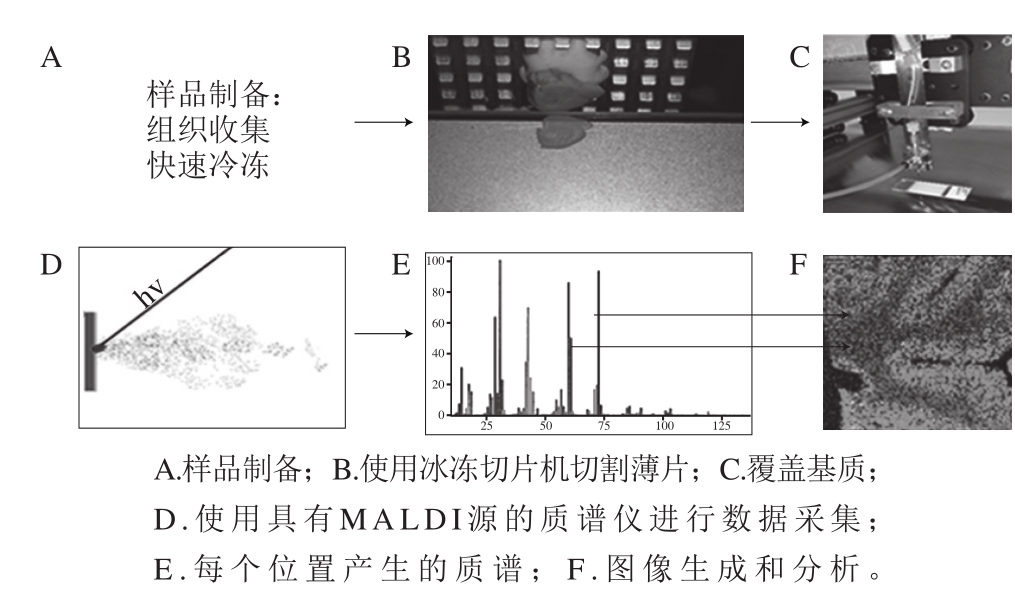
图1 典型MALDI-MSI组织分析的工作流程[10]
Fig. 1 Typical MALDI-MSI workflow for tissue analysis[10]
2 MALDI-MSI样品制备
MALDI-MSI技术的关键在于样品的制备方法和基质的选择。对于植物组织和动物组织,MALDI-MSI技术的样品制备方法已经逐渐成熟,因此,在食品分析中,肉类样品处理方法可参照哺乳动物组织进行处理,蔬菜和水果样品可参照植物组织进行处理。
2.1 样品组织的收集及贮藏
在MALDI-MSI成像中,样品的质量对内部化合物的分布有很大的影响,并且由于样品不能直接用来成像,因此,样品的处理和贮藏是能否获得高质量图像的关键因素。样品的收集和处理的过程一定要快速,因为组织一旦被分离,其内部物质就会发生降解。一般情况下将样品冻藏一段时间,直至分析前取出,样品的贮存时间取决于样品的性质,但是在样品的贮藏期间也要考虑分析物质的降解和迁移[11]。还有研究人员将样品放在液氮或干冰与不同溶剂混合的低温浴中快速冷冻,但不建议直接将样品放入液氮中,以免组织发生断裂或破碎。Schwartz等[12]将新鲜的组织放入到塑料试管中以避免这种情况的发生。也有研究人员建议采用-70 ℃下的乙醇和异丙醇来代替液氮[13]。
2.2 切片
在用冰冻切片机切片时,需要用支持物对样品进行包埋,为切片提供支持。形状较规则的大样品可以用少量的去离子水直接固定在切片机试样夹里,但是较小的样品则需要用支持物进行包埋[14]。常用的支持物有石蜡、冰、质量分数10%明胶和羧甲基纤维素(carboxymethyl cellulose,CMC)溶液等[15]。在食品分析中,切片的厚度取决于样品性质和仪器的限制。一般情况下,组织切片的厚度为10~20 μm[16]。近年来也有研究人员发现,在分析高分子质量的物质(3~21 kDa)时,用2~5 μm厚度的样品切片可以获得高质量的图像[17]。切片之后,通过融裱或双面胶带将样品转移到质谱成像的靶上。
2.3 组织预处理
2.3.1 组织切片的冲洗
通常,在用基质覆盖之前,最好洗涤或漂洗组织切片,除去一些抑制目标分子电离的内源分子和盐,加重信号,以免产生复杂的图谱。也有研究人员认为,组织切片冲洗前须在真空或氮气中脱水,减少湿度对分析的影响。一般情况下使用酒精来清洗切片。对于脂肪含量较高的组织,一般用氯仿或二甲苯等溶液进行清洗。Enthaler等[18]研究表明,组织切片的冲洗会导致切片中蛋白质含量大幅降低。因此,在清洗过程中应避免除去一些成像的目标小分子,以获得高质量的图谱。另一种组织切片的洗涤方法是用溶剂湿润的无纤维纸局部清洗组织切片[19]。这种局部清洗的方法适用于易碎的组织切片,可以用来对比清洗区域和未清洗区域。
2.3.2 切片组织消化
利用MALDI-MSI技术分析蛋白质时,由于该技术具有探测上限,无法探测大分子蛋白质,因此在对大分子蛋白质分析前,需要对其进行组织消化获得小肽段[20]。随后,再通过MALDI-MSI技术用得到的小肽段的空间信息定位母蛋白质。组织消化是利用蛋白酶处理组织切片,产生多个小肽段。由于MALDI-MSI技术可鉴定蛋白酶水解后的小肽段,因此MALDI-MSI技术也适用于鉴定分析未知的蛋白质。
2.4 基质的选择及覆盖方法
2.4.1 基质的选择
在MALDI成像中,基质的类型和覆盖方法是获得高质量可重复的离子图像的关键[21]。由于激光束的不同,MALDI-MSI可分为紫外质谱成像(UV-MALDI-MSI)和红外质谱成像(IR-MALDI-MSI),与UV-MALDI-MSI相比,IR-MALDI-MSI的主要优点是样品中的内源水可以作为基质吸收红外辐射。但是,在分析的过程中,灵敏度可能会因为水分的不均匀分布而发生变化[22]。
在利用UV-MALDI-MSI对食品成分分析时,常用的基质为2,5-二羟基苯甲酸(2,5-dihydroxybenzoic acid,DHB)、芥子酸(sinapinic acid,SA)和α-氰基-4-羟基苯丙烯酸(α-cyano-4-hydroxycinnamic acid,CHCA),其中,SA主要适用于高分子质量的蛋白质,在分子质量约为4 kDa的高分子化合物(如蛋白质和肽)的分析中,SA可以提供更好的信噪比[23]。Cavatorta等[24]利用MALDI-MSI技术分析桃中的变应原时,选择了SA作为基质,获得了高质量的图谱。DHB、9-氨基吖啶(9-aminoacridine,9-AA)和CHCA适用于分析低分子质量的物质。另外,基质混合物的应用可以改善电离效率和信号强度[23]。Lemaire等[11]在MALDI-MSI中,使用CHCA-二甲基苯胺混合液作为基质复合物,研究发现相比于单独使用CHCA,CHCA-二甲基苯胺基质复合物形成了致密且均匀的基质层,其信号强度更高。
2.4.2 基质的覆盖方法
基质对分析物的覆盖方法是能否获得高质量图谱的重要因素,其方法大致可分为湿法和干法。用于湿法覆盖分析物的设备有手动式气喷雾器、压电高频振动基质喷雾器、声控微滴喷射器和化学打印喷射器[25]。其中手动式喷雾器是最早也是最常用于基质喷涂的工具,该方法简单易行,但是人工操作会导致每次喷雾的均匀度不一致,因此,获得均匀的基质涂层很大程度上依赖于操作者的熟练程度[26]。湿法覆盖分析物应该在喷雾和干燥反复循环的条件下操作,每次喷雾要达到刚好润湿切片的效果。根据组织表面性质的不同,循环次数一般为3~10 次。基质干燥涂层法是将基质和样品一同放在真空室里,加热基质,冷却样品,使基质直接升华到冷却后的样品上,大大降低了分析物的离域。然而,对于需要掺入基质晶体中的分析物,其信号强度相对较差[27]。
3 MALDI-MSI在食品成分分析中的应用
食物中含有碳水化合物、脂质和蛋白质等营养成分,有些食物还含有一些内源性毒素。对这些成分进行常规的化学方法分析时操作步骤繁琐,需要提取、分离分析物质,然后采用高效液相色谱法、气相色谱-质谱联用法等进行分析。而MALDI-MSI技术作为一种快速高效的分析技术,可以克服这些缺点,近些年在食品分析中得到了应用。
3.1 对食品中碳水化合物的分析
碳水化合物在生命活动过程中起着重要的作用,是生物体维持生命活动所需能量的主要来源,可分为单糖、二糖、寡糖和多糖。水果和蔬菜中的糖类主要有蔗糖、葡萄糖、果糖等,这3 种成分的含量在不同水果和蔬菜中是不同的。在草莓、香蕉、葡萄、西红柿和茄子的组织切片中呈现出多样化的空间分布[28-29]。
Li Yue等[28]利用MALDI-MSI技术在草莓的表皮组织和种子中发现了丰富的蔗糖(图2)。蔗糖是碳素同化物的主要转移形式,也是植物器官的碳骨架和无法进行光合作用时器官的能量来源。Berisha等[30]利用MALDI-MSI技术探索葡萄的外果皮和种子中糖类的空间分布,发现单糖主要分布在葡萄的外果皮中,而二糖主要分布在中果皮里。目前,质谱成像技术在碳水化合物中的应用主要集中在单糖和二糖等糖类的检测[6],而在一些化学结构复杂的碳水化合物实验中还存在着许多问题。通过MALDI-MSI技术获得的碳水化合物的空间分布信息有助于我们探索水果和蔬菜中碳水化合物的生物合成和代谢途径,控制果蔬在贮藏与加工过程中营养成分的变化,以保持和改善果蔬及其加工制品的感官品质。


图2 草莓表皮与嵌入的种子周围的3 个主要成分的MALDI-MSI图像[28]
Fig. 2 Optical image of a strawberry skin compared to MALDI-MSI images for three major components around embedded seeds[28]
3.2 对食品中脂质的分析
脂质是生物体内一大类微溶于水、溶于有机溶剂的物质,它的种类和品质的好坏直接影响着食品的风味、质地和颜色。尤其是肉制品,在不良的条件下进行贮藏极易发生氧化。脂质氧化会生成低级脂肪酸、醛、酮等物质,它们具有刺鼻的不良气味,影响肉类的风味、质地、颜色和营养。因此控制和减少脂质氧化是肉类食品科学中研究的重点。传统的分析手段可以实现对脂质含量、分布及氧化程度的分析,但是在脂质提取过程中物质的分布信息会有所缺失,无法达到分子水平。而MALDI-MSI技术通过添加基质保护促进了样品的电离。Dyer等[31]首先根据脂质在质谱中的相对丰度,确定出磷脂、甘油三酯和胆固醇为标记物,随后利用MALDI-MSI技术绘制了脂质氧化降解产物的空间分布图,研究了不同的包装条件(高氧、空气和真空)对牛背最长肌在贮藏期间脂质氧化的影响。

图3 牛背最长肌中磷脂酰胆碱(m/z 760.6)和未确定脂质(m/z 428.2)的离子强度的空间异质性[31]
Fig. 3 Observed spatial heterogeneity of the ion intensities of a phosphatidylcholine (m/z 760.6) and an unidentified lipid(m/z 428.2) in bovine M. longissimus dorsi lumborum[31]
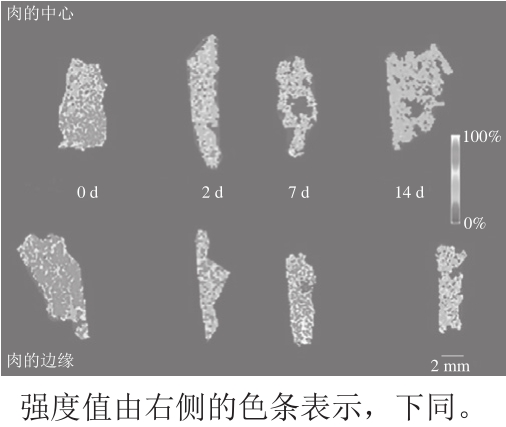
图4 高氧包装牛背最长肌中心和边缘的磷脂酰乙醇胺(m/z 744.4)的离子图像[31]
Fig. 4 Ion images of phosphatidylethanolamine (m/z 744.4) from one bovine M. longissimus dorsi lumborum, sub-sampled, packaged in high-oxygen modified atmosphere from the center and from the edge[31]
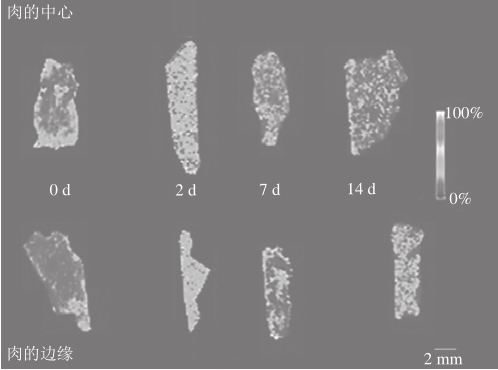
图5 高氧包装牛背最长肌中心和边缘的溶血磷脂酰胆碱(m/z 496.3)的离子图像[31]
Fig. 5 Ion images of a lysophosphatidylcholine (m/z 496.3) from one bovine M. longissimus dorsi lumborum, sub-sampled, packaged in high-oxygen modified atmosphere from the center and from the edge[31]
如图3所示,可明显观察到肌肉的大理石纹。结果表明,肉中的脂质分布具有空间异质性,在高氧的条件下,磷脂酰乙醇胺(图4)和磷脂酰胆碱(图5)的相对丰度快速下降,胆固醇显示出相对较高的氧化稳定性。Zaima等[32]也利用MALDI-MSI技术观察了不同产地牛肉中的脂质空间分布,根据样品中的脂质组成不同,将牛肉样品进行了分组,不同组别中的牛肉脂质含量并没有显著差异。因此,在同位素分析鉴定样品之前,可以利用MALDI-MSI技术对脂质组分含量没有显著差异的牛肉样品进行简单的分类。在允许检测的范围内,研究人员可以利用MALDI-MSI技术快速检测和分析肉类组织切片中的内源性脂质,评估肉类在生产、加工、包装、贮藏和流通中脂质氧化降解程度,以最大限度地提高食品质量与价值。也可将该技术与代谢组学方法结合评估牛肉来源的真实性,以确保消费者食用健康无污染的肉类制品。
3.3 对食品中蛋白质的分析
MALDI-MSI技术也被用来快速定位蛋白质和一些小肽。众所周知,脂质转移蛋白广泛分布于动物、植物和微生物中,是植物生命活动中一类重要的活性蛋白质[33],其中脂质转移蛋白Pru p 3是桃中的主要变应原,能够引起人体的超敏反应,造成组织损伤或生理功能紊乱[34]。Bencivenni等[35]利用MALDI-MSI技术对西红柿的果皮、果肉和种子进行了成像。观察发现,西红柿中的非特异性脂质转移蛋白主要存在于种子区域,果皮和果肉组织中并未观察到,若除去西红柿的种子,可以大大减少西红柿制品中非特异性脂质转移蛋白的含量,这为生产低变应原的农产品提供了技术支持。

图6 桃的MALDI-MSI图像[24]
Fig. 6 MALDI-MSI image of peach[24]
Cavatorta等[24]在桃的MALDI-MSI图像中观察到,如图6所示,脂质转移蛋白(Pru p 3变应原)只存在于桃的外果皮中,这与用免疫组织化学法研究的结果一致[36],人对桃的过敏反应与桃皮中的Pru p 3变应原有关。此外,还有研究人员对大豆子叶进行了成像观察,通过分析蛋白质的空间分布,对不同的样品进行了区分,并深入研究了不同植物的生长遗传差异。Grassl等[37]利用MALDI-MSI技术研究了大豆中的植物蛋白,首先用胰蛋白酶将切片组织消化成多个小肽,结果发现每一种肽都呈现出完全不同的定位分布。这些研究表明MALDI-MSI可以用于研究食品中的蛋白质或肽的分布。
3.4 对食品中其他成分的分析
MALDI-MSI技术也可用来分析和定位食品中的一些生物活性成分。众所周知,黄酮类化合物存在于自然界的植物中,具有良好的抗氧化活性和抗癌活性[38]。Berisha[30]和Yoshimura[39]等分别在葡萄和蓝莓的成像中发现,二者花色苷的分布高度相似,花翠素和矮牵牛苷定位在葡萄的外果皮,花青素、芍药素等其他花色苷分布在葡萄的外果皮和内果皮[37],在蓝莓的外果皮中也同样定位到这些花色苷[39]。通过分析可知,葡萄和蓝莓中花色苷的定位取决于糖苷配基而不是糖基。此外,Zaima等[40]通过MALDI-MSI技术鉴定水稻中的代谢产物,发现γ-谷维素和植酸位于麸皮中,生育酚分布在胚芽中。Tairi等[41]选用了CHCA作为基质,利用MALDI-MSI分别观察了辣椒的胎盘、果皮、种子区的辣椒素,图像显示,与其他部分相比,胎盘区的辣椒素信号强度最强,并且在胎盘表面更丰富。由此可见,通过质谱成像技术获得的空间信息将有助于了解这些生物活性成分相关基因的表达模式,可以更好的筛选代谢物和食源性营养因子,了解其在食品中的生物学意义。此外,MALDI-MSI技术也可用于食品中一些有毒有害物质的检测。马铃薯中含有一种弱碱性的生物碱,致毒成分是茄碱,又称马铃薯毒素,食用过量会引起恶心、呕吐、呼吸衰竭等症状。Cabral等[42]通过MALDI-MSI技术检测到在马铃薯芽中马铃薯毒素的含量最多,周皮的α-卡茄碱和α-茄碱的丰度比髓质区分别高出5 倍和2 倍。
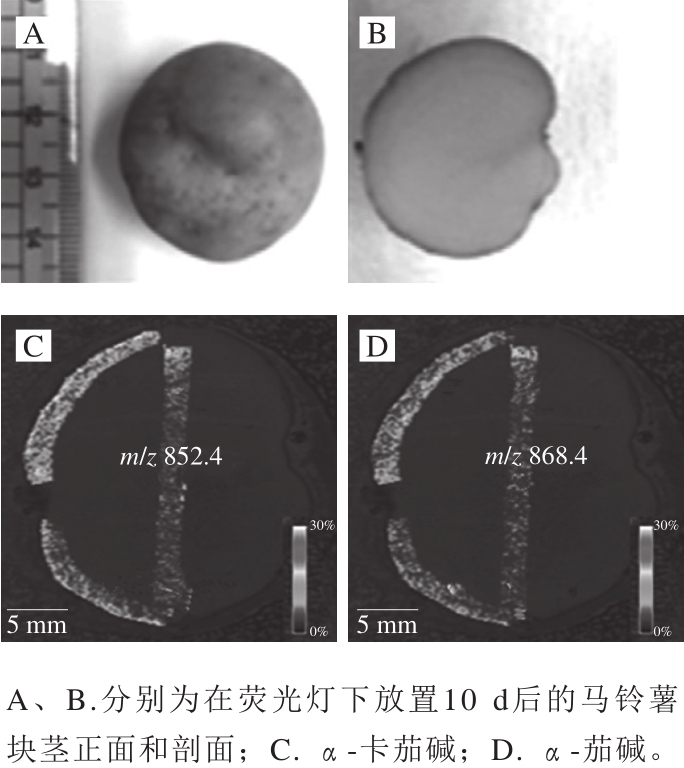
图7 马铃薯块茎中的α-卡茄碱(m/z 852.4)和α-茄碱(m/z868.4)的MALDI-MSI图像[43]
Fig. 7 MALDI-MSI images of α-chaconine (m/z 852.4) and α-solanine (m/z 868.4) from a section of potato tuber[43]
Ha等[43]利用MALDI-MSI技术研究发现,如图7所示,当马铃薯的块茎暴露在光照下时,颜色变绿,生物碱的含量发生变化,与髓部相比,块茎周皮中的生物碱含量显著增加(P<0.05)。
这些应用实例表明,MALDI-MSI技术可以准确的绘制出食品中营养成分和有害物质的空间分布图像,这种可视化分布能够有效的帮助我们分析食品中的营养成分:从而有助于探索果蔬中碳水化合物的生物合成和代谢途径,理清其作为结构物质在果蔬质构方面的作用及其在果蔬贮藏与加工过程中的变化与控制,以保持和改善果蔬及其加工制品的感官品质;有助于快速评估肉类制品在生产、加工、包装、贮藏与流通中脂质氧化降解的程度,以最大限度地提高食品质量和价值;有助于了解食品中某些生物活性成分相关基因的表达模式,更好的筛选代谢物和食源性营养因子;有助于检测和监控食品中一些有毒有害的物质,帮助我们在分子水平上监测食品质量和安全。
4 结 语
MALDI-MSI在食品分析中的应用虽然起步较晚,研究有限,但是凭借其无需标记靶分子、无需提取待测物质、分子质量范围广、具有成像能力等优点在食品分析中取得了一定的进展。但其在具体的实验中还存在着许多问题:一方面,对于组织中蛋白质和多肽鉴定一直是研究质谱成像技术的难点,尤其是未知蛋白质的鉴定,通常利用原位酶切技术可以提高鉴定的成功率,但是仍然会受到目标蛋白分布散乱、丰度较低等因素的干扰,难以得到理想的实验结果;另一方面,在研究食品中某些含量较低的营养物质或有毒有害物质时,对实验的灵敏度或空间分辨率要求较高,因此在实验中,应合理的选择基质的类型和覆盖方法,并对其进行实验优化[7]。
利用MALDI-MSI技术获得的化合物空间分布信息,可以在生产中强化食品营养元素,改善食品加工和包装技术,以及人工定向干预改变代谢产物和筛选食源性营养因子,加强对食品的质量与安全的保障。目前,MALDI-MSI技术在肉品中的应用甚少,未来可以尝试利用该技术研究定位肉品中的风味物质,根据获得的空间分布图分析肉品中风味物质的生成机制,减少风味物质的损失。同时,也可将MALDI-MSI技术与其他技术相结合,如拉曼光谱技术[44]、近红外光谱技术等[45],使MALDI-MSI技术在食品研究中的应用前景更加广阔。
参考文献:
[1] CAPRIOLI R M, FARMER T B, GILE J. Molecular imaging of biological samples: localization of peptides and proteins using MALDI-TOF MS[J]. Analytical Chemistry, 1997, 69(23): 4751-4760.DOI:10.1021/ac970888i.
[2] 何美玉, 王光辉, 熊少祥. 现代生物质谱及其应用[J]. 现代仪器,2001(1): 6-8. DOI:10.3969/j.issn.1672-7916.2001.01.002.
[3] REYZER M L, HSIEH Y, NG K, et al. Direct analysis of drug candidates in tissue by matrix-assisted laser desorption/ionization mass spectrometry[J]. Mass Spectrom, 2003, 38: 1081-1092. DOI:10.1002/jms.525.
[4] ACQUADRO E, CABELLA C, GHIANI S, et al. Matrix-assisted laser desorption ionization imaging mass spectrometry detection of a magnetic resonance imaging contrast agent in mouse liver[J].Analytical Chemistry, 2009, 81(7): 2779-2787. DOI:10.1021/ac900038y.
[5] GENJI T, FUKUZAWA S, TACHIBANA K. Distribution and possible function of the marine alkaloid, norzoanthamine, in the zoanthid Zoanthus sp. using MALDI imaging mass spectrometry[J]. Marine Biotechnology, 2010, 12(1): 81-87. DOI:10.1007/s10126-009-9202-5.
[6] YOSHIMURA Y, GOTO-INOUE N, MORIYAMA T, et al.Significant advancement of mass spectrometry imaging for food chemistry[J]. Food Chemistry, 2016, 210: 200-211. DOI:10.1016/j.foodchem.2016.04.096.
[7] HEEREN R A, SWEEDLER J V. Imaging mass spectrometry imaging[J]. International Journal of Mass Spectrometry, 2007, 260(2):89. DOI:10.1016/j.ijms.2006.11.016.
[8] JUNGMSNN J H, HEEREN R M A. Emerging technologies in mass spectrometry imaging[J]. Journal of Proteomics, 2012, 75: 5077-5092.DOI:10.1016/j.jprot.2012.03.022.
[9] SINHA T K, KHATIB-SHAHIDI S, YANKEELOV T E, et al.Integrating spatially resolved three-dimensional MALDI IMS with in vivo magnetic resonance imaging[J]. Nature Methods, 2008, 5(1): 57-59. DOI:10.1038/NMETH1147.
[10] TRIM P J, SNEL M F. Small molecule MALDI MS imaging: current technologies and future challenges[J]. Methods, 2016, 104: 127-141.DOI:10.1016/j.ymeth.2016.01.011.
[11] LEMAIRE R, WISZTORSKI M, DESMONS A, et al. MALDI-MS direct tissue analysis of proteins: improving signal sensitivity using organic treatments[J]. Analytical Chemistry, 2006, 78(20): 7145-7153.DOI:10.1021/ac060565z.
[12] SCHWARTZ S A, REYZER M L, CAPRIOLI R M. Special feature: perspective direct tissue analysis using matrix-assisted laser desorption/ionization mass spectrometry: practical aspects of sample preparation[J]. Journal of Mass Spectrometry, 2003, 38(7): 699-708.DOI:10.1002/jms.505.
[13] 邹贵勉, 汤冬娥, 睢维国. 基质辅助激光解析质谱成像技术的样品制备及其应用[J]. 国际病理科学与临床杂志, 2010, 30(6): 536-542.DOI:10.3969/j.issn.1673-2588.2010.06.017.
[14] RUBAKHIN S S, ULANOV A, SWEEDLER J V. Mass spectrometry imaging and GC-MS profiling of the mammalian peripheral sensory-motor circuit[J]. Journal of the American Society for Mass Spectrometry, 2015, 26(6): 958-966. DOI:10.1007/s13361-015-1128-8.
[15] BHANDARI D R, SHEN Tian, ROMPP A, et al. Analysis of cyathanetype diterpenoids from Cyathus striatus and Hericium erinaceus by high-resolution MALDI MS imaging[J]. Analytical and Bioanalytical Chemistry, 2014, 406(3): 695-704. DOI:10.1007/s00216-013-7496-7.
[16] GOODWIN R J A, PENNINGTON S R, PITT A R. Protein and peptides in pictures: imaging with MALDI mass spectrometry[J].Proteomics, 2008, 8(18): 3785-3800. DOI:10.1002/pmic.200800320.
[17] PEUKERT M, MATROS A, LATTANZIO G, et al. Spatially resolved analysis of small molecules by matrix-assisted laser desorption/ionization mass spectrometric imaging (MALDI-MSI)[J].New Phytologist, 2012, 193(3): 806-815. DOI:10.1111/j.1469-8137.2011.03970.x.
[18] ENTHALER B, BUSSMANN T, PRUNS J K, et al. Influence of various on-tissue washing procedures on the entire protein quantity and the quality of matrix-assisted laser desorption/ionization spectra[J].Rapid Communications in Mass Spectrometry, 2013, 27(8): 878-884.DOI:10.1002/rcm.6513.
[19] VAN HOVE E R, SMITH D F, FORMAI L, et al. An alternative paper based tissue washing method for mass spectrometry imaging: localized washing and fragile tissue analysis[J]. Journal of the American Society for Mass Spectrometry, 2011, 22(10): 1885-1890. DOI:10.1007/s13361-011-0203-z.
[20] MORITA Y, IKEGAMI K, GOTO-INOUE N, et al. Imaging mass spectrometry of gastric carcinoma in formalin-fixed paraffinembedded tissue microarray[J]. Cancer Science, 2010, 101(1): 267-273. DOI:10.1111/j.1349-7006.2009.01384.x.
[21] COHEN S L, CHAIT B T. Influence of matrix solution conditions on the MALDI-MS analysis of peptides and proteins[J]. Analytical Chemistry, 1996, 68(1): 31-37. DOI:10.1021/ac9507956.
[22] BOKHART M T, MUDDUMAN D C. Infrared matrix-assisted laser desorption electrospray ionization mass spectrometry imaging analysis of biospecimens[J]. Analyst, 2016, 141(18): 5236-5245. DOI:10.1039/c6an01189f.
[23] SHANTA S R, ZHOU Lihua, PARK Y S, et al. Binary matrix for MALDI imaging mass spectrometry of phospholipids in both ion modes[J]. Analytical Chemistry, 2011, 83(4): 1253-1259.DOI:10.1021/ac1029659.
[24] CAVATORTA V, SFORZA S, MASTROBUONI G, et al.Unambiguous characterization and tissue localization of Pru p 3 peach allergen by electrospray mass spectrometry and MALDI imaging[J].Journal of Mass Spectrometry, 2009, 44(6): 891-897. DOI:10.1002/jms.1562.
[25] 杨帆, 原剑, 郑俊杰. MALDI-TOF生物质谱成像技术的进展[J]. 分析仪器, 2010(3): 1-8. DOI:10.3969/j.issn.1001-232X.2010.03.001.
[26] KALETAS B K, VAN DER WIEL I M, STAUBER J, et al. Sample preparation issues for tissue imaging by imaging MS[J]. Proteomics,2009, 9(10): 2622-2633. DOI:10.1002/pmic.200800364.
[27] YANG J Y, PHELAN V V, SIMKOVSKY R, et al. Primer on agar-based microbial imaging mass spectrometry[J]. Journal of Bacteriology, 2012, 194(22): 6023-6028. DOI:10.1128/JB.00823-12.
[28] LI Yue, SHRESTHA B, VERTES A. Atmospheric pressure molecular imaging by infrared MALDI mass spectrometry[J]. Analytical Chemistry, 2006, 79(26): 523-532. DOI:10.1021/ac061577n.
[29] GOTO-INOUE N, SETOU M, ZAIMA N. Visualization of spatial distribution of gamma-aminobutyric acid in eggplant (Solanum melongena) by matrix-assisted laser desorption/ionization imaging mass spectrometry[J]. Analytical Sciences, 2010, 26(7): 821-825.
[30] BERISHA A, DOLD S, GUENTHER S, et al. A comprehensive high-resolution mass spectrometry approach for characterization of metabolites by combination of ambient ionization, chromatography and imaging methods[J]. Rapid Communications in Mass Spectrometry,2014, 28(16): 1779-1791. DOI:10.1002/rcm.6960.
[31] DYER J M, DEB-CHOUDHURY S, CORNELLISON S, et al. Spatial and temporal mass spectrometric profiling and imaging of lipid degradation in bovine M. longissimus dorsi lumborum[J]. Journal of Food Composition and Analysis, 2014, 33(2): 203-209. DOI:10.1016/j.jfca.2013.12.001.
[32] ZAIMA N, GOTO-INOUE N, HAYASAKA T. Authenticity assessment of beef origin by principal component analysis of matrixassisted laser desorption/ionization mass spectrometric data[J].Analytical and Bioanalytical Chemistry, 2011, 400(7): 1865-1871.DOI:10.1007/s00216-011-4818-5.
[33] MARZBAN G, PUEHRINGER H, DEY R, et al. Localization and distribution of the major allergens in apple fruits[J]. Plant Science,2005, 169(2): 387-394.
[34] FEMANDEZ-RIVAS M, GONZALEZ-MANCEBO E, RODRIGUEZPEREZ R, et al. Clinically relevant peach allergy is related to peach lipid transfer protein, Pru p 3, in the Spanish population[J]. Journal of Allergy and Clinical Immunology, 2003, 112(4): 789-795.DOI:10.1016/S0091-6749(03)02016-5.
[35] BENCIVENNI M, FACCINI M, ZECCHI A, et al. Electrospray MS and MALDI imaging show that non-specific lipid-transfer proteins(LTPs) in tomato are present as several isoforms and are concentrated in seeds[J]. Journal of Mass Spectrometry, 2014, 49(12): 1264-1271.DOI:10.1002/jms.3454.
[36] BOTTON A, VEGRO M, DE FRANCESCHI F, et al. Different expression of Pp-LTP1 and accumulation of Pru p 3 in fruits of two Prunus persica L. Batsch genotypes[J]. Plant Science, 2006, 171(1):106-113. DOI:10.1016/j.plantsci.2006.02.016.
[37] GRASSL J, TAYLOR N L, MILLAR A H. Matrix-assisted laser desorption/ionisation mass spectrometry imaging and its development for plant protein imaging[J]. Plant Methods, 2011, 7(1): 21.DOI:10.1186/1746-4811-7-21.
[38] ROSS J A, KASUM C M. Dietary flavonoids: bioavailability,metabolic effects, and safety[J]. Annual Review of Nutrition, 2012,22: 19-34. DOI:10.1146/annurev.nutr.22.111401.144957.
[39] YOSHIMURA Y, ENOMOTO H, MORIYAMA T, et al.Visualization of anthocyanin species in rabbiteye blueberry Vaccinium ashei by matrix-assisted laser desorption/ionization imaging mass spectrometry[J]. Analytical and Bioanalytical Chemistry, 2012, 403(7):1885-1895. DOI:10.1007/s00216-012-5876-z.
[40] ZAIMA N, GOTO-INOUE N, HAYASAKA T, et al. Application of imaging mass spectrometry for the analysis of Oryza sativa rice[J].Rapid Communications in Mass Spectrometry, 2010, 24(18): 2723-2729. DOI:10.1002/rcm.4693.
[41] TAIRI S, UEMATSU K, KANEKO D, et al. Mass spectrometry imaging: applications to food science[J]. Analytical Sciences, 2014,30(2): 197-203. DOI:10.2116/analsci.30.197.
[42] CABRAL E C, MIRABELLI M F, PEREZ C J, et al. Blotting assisted by heating and solvent extraction for DESI-MS imaging[J]. Journal of the American Society for Mass Spectrometry, 2013, 24(6): 956-965.DOI:10.1007/s13361-013-0616-y.
[43] HA M, KWAK J H, KIM Y, et al. Direct analysis for the distribution of toxic glycoalkaloids in potato tuber tissue using matrix-assisted laser desorption/ionization mass spectrometric imaging[J]. Food Chemistry,2012, 133(4): 1155-1162. DOI:10.1016/j.foodchem.2011.11.114.
[44] RADU A I, KUELLMER M, GIESE B. Surface-enhanced raman spectroscopy (SERS) in food analytics: detection of vitamins B2and B12in cereals[J]. Talanta, 2016, 160: 289-297. DOI:10.1016/j.talanta.2016.07.027.
[45] BAKRY R, RAINER M, HUCK C W, et al. Protein profiling for cancer biomarker discovery using matrix-assisted laser desorption/ionization time-of-flight mass spectrometry and infrared imaging: a review[J]. Analytica Chimica Acta, 2011, 690(1): 26-34. DOI:10.1016/j.aca.2011.01.044.