精氨酸酶是具有两个活性中心(金属离子结合位点与底物结合位点)的一类碱性氨基酸水解酶,活性需要金属离子的参与[1-5]。早在20世纪初,人们在哺乳动物的肝脏中首次发现了精氨酸酶[6],该酶具有水解精氨酸的作用。随着研究的深入,在动物、植物和微生物中都发现了精氨酸酶。在脊椎动物中,目前发现存在两种类型的精氨酸酶,精氨酸酶1型称为细胞质或者肝型精氨酸酶,主要存在于动物的肝脏细胞中,精氨酸酶2型为线粒体型或者非肝型精氨酸酶,常见于小肠、肾等肝细胞外的脏器细胞中[7-12]。由于精氨酸酶能够催化精氨酸生成鸟氨酸和尿素[13],而正常细胞对精氨酸缺乏的耐受性要强于肿瘤细胞,因此精氨酸酶能够通过分解细胞中的精氨酸有效地抑制肿瘤细胞的增殖[14-16]。有研究发现,人类1型精氨酸酶的点突变造成了高精氨酸败血症[17-19]。在植物中,精氨酸是很多植物种子N元素的重要组成部分,占总含氮量17%左右,因此在种子萌发和幼苗的生长发育阶段,精氨酸酶活力逐渐升高,分解精氨酸以促进氮素的循环利用。对玉米精氨酸酶的研究发现,提高玉米中精氨酸酶活力能够显著促进植株的生长,提高干物质的积累。在一些微生物中也发现了精氨酸酶,并对其催化特性进行了深入的研究。
目前精氨酸酶的工业应用主要在于酶法生产L-鸟氨酸,L-鸟氨酸是生物体内普遍存在的一种非蛋白氨基酸,参与蛋白质、糖以及脂肪的分解代谢。近年来的医疗研究显示,L-鸟氨酸在保肝护肝、促进病人康复方面疗效显著,同时作为一种功能性氨基酸,L-鸟氨酸能够改善营养,具有保健作用[1-4],目前L-鸟氨酸已被广泛应用于制药、工业、保健和食品生产,其市场前景远大。L-鸟氨酸的化学合成法步骤繁多,产物含有外消旋混合物,分离困难,难以大规模生产。微生物发酵法因微生物代谢途径复杂,突变菌株的筛选困难,易发生突变,并且L-鸟氨酸产量低,发酵液成分复杂,后续分离纯化成本高,而酶水解法因专一性很强受到研究者的关注。
精氨酸酶的挖掘与研究是利用酶法大规模生产L-鸟氨酸的基础。微生物来源的精氨酸酶多见于芽孢杆菌属,其催化精氨酸产生L-鸟氨酸的活性较高。其中Bacillus caldovelox来源的精氨酸酶与L-精氨酸的Km最低,为3.4 mmol/L。其次是芽孢杆菌属B. brevis、B. subtilis KY3281以及B. thuringierisis,它们与L-精氨酸的Km值均较低,这表明它们与底物的亲和力高,非常适用于以L-精氨酸为底物催化生产L-鸟氨酸[4,19-22]。B. caldovelox来源的精氨酸酶相应的重组蛋白发酵生产L-鸟氨酸的产量达112.3 g/L,该酶的最适反应温度较高,达到60 ℃,然而此温度下酶的稳定性受到影响,限制了其工业化应用[4]。最近研究发现芽孢杆菌属B. amyloliquefaciens来源的精氨酸酶在B. subtilis中过量表达,其L-鸟氨酸的产量达到356.9 g/L[13]。由此可见,芽孢杆菌属精氨酸酶的开发潜力巨大。
本研究通过对特基拉芽孢杆菌(B. tequilensis)进行全基因组比对分析,从本实验室1 株B. tequilensis PanD37[23]克隆到一个精氨酸酶基因,并命名为BteqARG,构建原核表达载体后在大肠杆菌(Escherichia coli)BL21(DE3)中高效表达该基因,酶活力分析显示该酶对pH 8.0~10.0的催化环境以及50~60℃高温耐受性好,酶活力高,生产成本低,有重要应用价值。此研究可以为精氨酸酶基因的研究提供参考素材,也为进一步研究和开发新型精氨酸酶提供理论支持。
1 材料与方法
1.1 材料与试剂
产精氨酸酶的菌株来自于本实验保存菌种B. tequilensis PanD37[23];原核表达载体pET28a(+)EMD Biosciences (Novagen)公司;E. coli DH5α、E.coli BL21(DE3) 北京全式金生物有限公司。
Ex Taq DNA聚合酶、限制性内切酶、T4 DNA连接酶、DNA分子质量标准 宝生物工程(大连)有限公司;细菌基因组DNA提取试剂盒、DNA凝胶回收试剂盒、质粒提取试剂盒、改良型Bradford蛋白浓度测定试剂盒 上海生工生物工程股份有限公司;Agarose His标签蛋白纯化试剂盒 北京康为世纪生物科技有限公司;L-精氨酸、L-鸟氨酸、三氯乙酸 北京鼎国生物技术有限公司;PCR引物合成 生工生物工程(上海)股份有限公司;其他试剂均为国产分析纯。
1.2 仪器与设备
核酸电泳仪、蛋白电泳仪 美国伯乐公司;聚合酶链式反应(polymerase chain reaction,PCR)仪德国Eppendorf公司;高效液相色谱(high performance liquid chromatography,HPLC)仪 安捷伦科技(中国)有限公司。
1.3 方法
1.3.1 培养基的配制
LB液体培养基:胰蛋白胨10 g/L,酵母提取物 5 g/L,氯化钠5 g/L,用1 mol/L NaOH溶液调pH值为7.0;LB固体培养基是在LB液体培养基中加入15 g/L的琼脂粉,培养基的灭菌条件皆为:121 ℃,20 min。
1.3.2 BteqARG基因的扩增
根据NCBI公布的B. tequilensis全基因组序列(LGRW01000001)设计引物,在上游加入NcoI酶切位点,引物命名为BteqARG-NcoIF:5’-AACCATGGATAA AACGATTTCGGTTATTGG-3’;在下游加入XhoI酶切位点,引物命名为BteqARG-XhoIR:5’-TTCTCGAGTTACA GCAGCTTCTTCCCTAAC-3’,下划线部分为酶切位点。
在LB液体培养基中接种本实验保存的B. tequilensis,30 ℃、200 r/min培养16 h,离心收集菌体,利用细菌基因组DNA提取试剂盒(B518225)提取其基因组DNA。具体抽提方法详见该试剂盒说明书。
以提取的B. tequilensis基因组DNA为模板,进行BteqARG基因的PCR扩增,反应体系为20 μL,其中基因组DNA 0.5 μL,上下游引物(10 μmol/L)各1 μL,Ex Taq DNA聚合酶 0.2 μL,10×聚合酶缓冲液 2 μL,dNTPs(10 mmol/L) 0.4 μL,不足部分用ddH2O补齐。PCR反应程序:94 ℃预变性3 min;94 ℃变性40 s,56 ℃退火40 s,72 ℃延伸1 min,30个循环;72 ℃延伸10 min;PCR产物使用1.0 g/100 mL琼脂糖凝胶电泳检测,使用DNA凝胶回收试剂盒(B511139)进行回收。
1.3.3 重组原核表达载体的构建
使用NcoI和XhoI对载体pET28a(+)和BteqARG基因琼脂糖凝胶电泳回收的目的片段分别进行双酶切,目的基因片段和载体片段用T4 DNA连接酶于16 ℃连接过夜,连接产物转化E. coli DH5α感受态细胞,并涂布在含50 μg/mL卡那霉素的LB固体培养基上,37 ℃培养12~16 h进行筛选,筛选得到的重组载体进行菌落PCR和质粒双酶切鉴定,阳性克隆进行测序验证(华大基因),将验证正确的质粒载体命名为pET28a-BteqARG。
1.3.4 重组工程菌的构建及重组蛋白的表达纯化
重组表达载体pET28a-BteqARG热激转化至E. coli BL21(DE3)感受态细胞,涂布于含50 μg/mL卡那霉素的LB固体培养基平板进行筛选,37 ℃培养12~16 h后,挑取单菌落进行PCR鉴定,阳性克隆可进一步进行蛋白的诱导和纯化,该重组工程菌菌株被命名为BteqARG-BL21(DE3)。
挑取阳性转化子接种于含有卡那霉素的LB液体培养基,于37 ℃过夜培养获得种子培养液。然后以体积比1∶100的比例将种子培养液接种于含有卡那霉素的LB液体培养基,37 ℃、180 r/min培养至OD600nm达到0.2左右,调节培养温度至20 ℃继续培养至OD600nm达到0.4~0.5时,取出1 mL菌液作对照组,剩下菌液加入终浓度为1.0 mmol/L的异丙基-β-D-硫代吡喃半乳糖苷(isopropyl β-D-1-thiogalactopyranoside,IPTG)进行蛋白诱导表达,诱导时间为12~16 h,诱导完成后,4 ℃离心收集菌体,超声波破碎后离心收集上清液,使用十二烷基硫酸钠-聚丙烯酰胺凝胶电泳(sodium dodecyl sulfate-polyacrylamide gel electrophoresis,SDS-PAGE)分析,收集的上清液可溶性蛋白利用Agarose His标签蛋白纯化试剂盒纯化目的蛋白,获得BteqARG纯化蛋白,利用Bradford蛋白分析试剂盒测定纯化蛋白的浓度,并利用SDS-PAGE进行检测。
1.3.5 BteqARG酶学性质测定
1.3.5.1 BteqARG酶活力测定
以L-精氨酸为底物,根据参考文献[19,24-25]方法在pH 10.0条件下测定BteqARG的酶活力。反应体系为200 μL:5 μL提纯酶液,100 μL Tris-HCl缓冲液(pH 10.0,50 mmol/L)、5 μL底物(L-精氨酸,其终浓度为200 mmol/L),添加终浓度为2 mmol/L Mn2+作为激活剂,反应液45 ℃温育15 min,然后加入等体积10 g/100 mL三氯乙酸终止反应,AccQ·Tag法[17]测定酶活力,使用Waters公司的AccQ·Fluor试剂盒柱前衍生,HPLC检测L-鸟氨酸含量。精氨酸酶活力定义:在pH 10.0、温度45 ℃的条件下,每分钟转化生成产物L-鸟氨酸1 μmol所需的酶量定义为1个酶活力单位,简称为U/mL。
1.3.5.2 温度、pH值和金属离子对BteqARG酶活力的影响
按上述方法,以L-精氨酸为底物,在Tris-HCl缓冲液(pH 10.0、50 mmol/L)中分别测定不同温度(30~60 ℃)条件下的酶活力,以最高酶活力为100%,分析温度对酶活力的影响;分别把提纯的酶液保存在30~60 ℃之间的不同温度条件下4 h,然后依据上述方法测定酶活力,检测酶的温度稳定性,以不进行温度处理直接测定的酶活力为100%。
配制pH值范围为7.0~11.5的Tris-HCl缓冲液,以L-精氨酸为底物,在45 ℃条件下测定酶活力,以最高酶活力为100%,分析pH值对酶活力的影响;利用不同pH值缓冲液稀释酶液,放置4 ℃,4 h后取样测定酶活力,以最高酶活力为100%,分析酶的pH值稳定性。
在上述确定的最适反应条件下,以L-精氨酸为底物,在反应液中分别添加1 mmol/L不同二价金属离子的氯盐(Cu2+、Ca2+、Co2+、Mn2+、Ni2+、Fe2+、Mg2+和Zn2+),以不添加任何金属离子时的酶活力为100%,分析金属离子对酶活力的影响。
1.3.5.3 BteqARG酶米氏常数(Km)和最大反应速率(Vmax)测定
酶的反应在最适温度、最适pH值条件下进行,分别添加不同浓度的L-精氨酸(0、5、10、20、40、80、100 mmol/L)为底物,反应终止后,测定反应生成的鸟氨酸的量,利用米氏方程的双倒数法测定精氨酸对该酶的Km值和最大反应速率Vmax,以已知浓度的鸟氨酸作标准曲线。
2 结果与分析
2.1 BteqARG基因的克隆与重组质粒的构建
利用PCR方法克隆获得B. tequilensis精氨酸酶基因BteqARG(图1A,泳道1,箭头所示),该精氨酸酶基因含有一个891 bp的开放阅读框,编码297 个氨基酸序列。将该基因的PCR片段连接到pET28a载体上,获得重组质粒pET28a-BteqARG。该质粒经NcoI和XhoI双酶切鉴定,可切出预期大小的目的条带(图1B,泳道1)。取该重组质粒送华大基因进行测序,获得的基因序列长度为891 bp,其对应的氨基酸序列与几个文献报道的芽孢杆菌属的精氨酸酶的氨基酸序列进行同源比对(图2),结果显示,BteqARG基因的氨基酸序列与解淀粉芽孢杆菌(B. amyloliquefaciens)精氨酸酶基因的氨基酸序列同源性最高达92%;与热溶芽孢杆菌(B. caldovelox)精氨酸酶的氨基酸序列同源性有70%,与炭疽芽孢杆菌(B. anthracis)来源的精氨酸酶氨基酸同源性为71%。
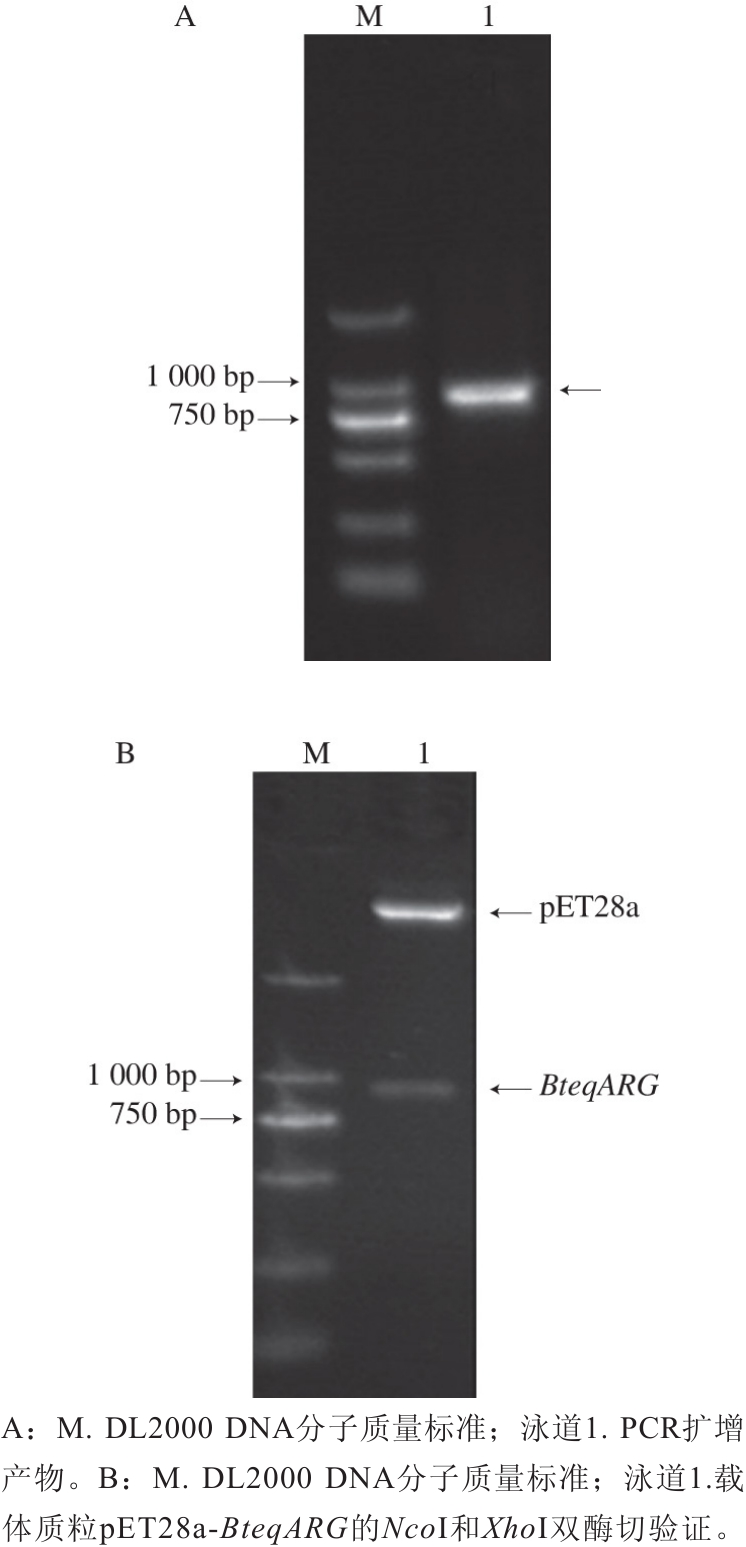
图1 精氨酸酶基因的克隆(A)与重组质粒的酶切验证(B)
Fig. 1 Electrophoretic analysis of PCR products of arginase gene and enzymatic digestion of recombinant plasmid

图2 部分芽孢杆菌属来源的精氨酸酶氨基酸序列同源性比对分析
Fig. 2 Comparative analysis of sequence homology of arginases from some Bacillus species
2.2 重组BteqARG酶的原核表达

图3 重组菌BteqARG-BL21(DE3)表达产物的SDS-PAGE图
Fig. 3 SDS-PAGE analysis of proteins expressed in recombinant BteqARG-BL21(DE3)
利用IPTG诱导重组BteqARG酶的表达,结果如图3所示,目的蛋白即重组BteqARG酶的分子质量在30~40 kDa之间。加入IPTG的终浓度为1 mmol/L,培养温度为37 ℃,重组酶的诱导温度为20 ℃,低温诱导有利于重组蛋白的可溶性表达,避免包涵体的形成。利用Ni-NTA吸附柱,成功纯化出重组BteqARG酶蛋白(图3),箭头所指为目的蛋白条带。
2.3 BteqARG的酶学性质分析
为了确定BteqARG酶活力,测定重组酶的粗酶催化精氨酸生成鸟氨酸的活力,该重组酶具有较高的精氨酸酶的活力,达到了1 109.8 U/mL,具有工业化应用的潜力。
2.3.1 温度、pH值和金属离子对BteqARG酶活性的影响
2.3.1.1 最适反应温度与温度稳定性

图4 温度对BteqARG酶活力及稳定性的影响
Fig. 4 Effect of temperature on the activity and stability of arginase
如图4所示,分析不同温度对BteqARG酶活力的影响,该酶在30~60 ℃均具有一定的酶活力,随着温度的升高,酶活力逐渐增加,45 ℃时达到最高,然后随着温度的升高开始降低。推断该酶的最适反应温度为45 ℃。在不同温度下保存该酶4 h后,在最适反应条件下测定酶活力,结果显示,在30 ℃保温4 h后,酶活力保持不变,继续提高温度,剩余相对酶活力保持稳定,在60 ℃保温4 h后,剩余酶活力仍保持在85%左右。
2.3.1.2 最适反应pH值与pH值稳定性
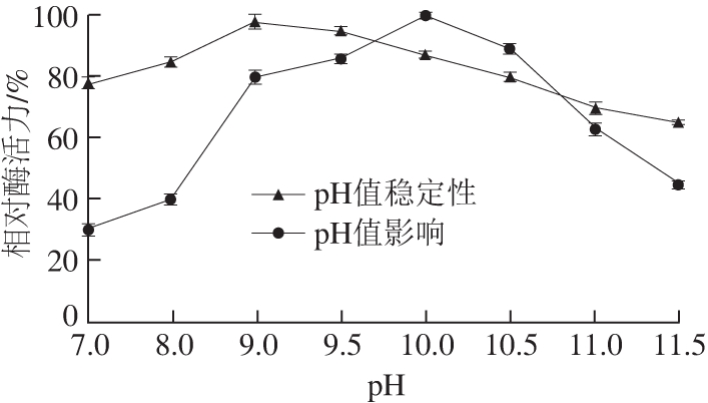
图5 pH值对BteqARG酶活力及稳定性的影响
Fig. 5 Effect of pH value on the activity and stability of arginase
如图5所示,在不同pH值条件下测定BteqARG酶活力,发现在pH值为10.0达到最大。将提纯酶液在不同pH值缓冲液中低温保存24h后测定剩余酶活力。从pH值稳定性曲线可以看出,该酶对pH 8.0~10.0的耐受性较好,在pH 8.0~10.0的缓冲液中4 ℃保存24 h,酶活力都只下降约2.0%~10.0%。在pH 9.0的缓冲液中酶活力基本稳定,仅下降2.0%。
2.3.1.3 不同金属离子对BteqARG酶活力的影响
不同金属离子对该酶的影响不同,其中Mn2+、Ni2+、Co2+对酶活力有明显的激活作用,而其他离子对该酶活力的影响微弱(图6)。以往的研究也表明Mn2+是精氨酸酶的催化因子,能够显著提高酶活力[19]。
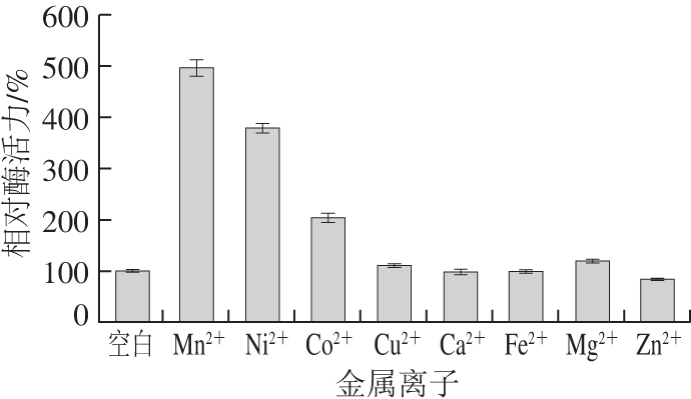
图6 金属离子对BteqARG酶活力的影响
Fig. 6 Effects of metal ions on arginase activity
2.3.2 BteqARG酶的Km和Vmax测定结果
在pH 10.0、45 ℃条件下,以不同浓度的L-精氨酸为BteqARG酶的反应底物,测定其反应速率,利用Lineweaver-Burk作图法求得该酶对精氨酸的Km值为10.72 mmol/L,BteqARG酶的动力学参数Kcat为298.75 s-1,Vmax值为555.6 μmol/(min·mg)(图7),与其他芽孢杆菌来源的精氨酸酶比较接近[4](表1)。上述结果表明BteqARG酶与精氨酸的亲和力较高,酶催化反应速率快,具有催化生产鸟氨酸的潜力。

图7 BteqARG酶的Lineweaver-Burk图
Fig. 7 Lineweaver-Burk plot of arginase
表1 不同来源精氨酸酶动力学参数的比较
Table 1 Enzymatic characteristics of arginases derived from different Bacillus species

注:—.无相关数据。
3 结 论
作为生物体内氮素循环的关键酶,精氨酸酶是一类二价阳离子依赖的水解酶,能够水解精氨酸形成鸟氨酸和尿素。精氨酸酶一般存在于产生尿素的动物体内,随着研究的深入,目前在一些不生成尿素的鱼类、植物和微生物中也发现了该酶。精氨酸酶因其催化特性在生物技术领域发挥着重要作用。作为半必需氨基酸,肿瘤细胞对精氨酸的耐受力低于正常细胞,因此精氨酸酶能够有效地抑制肿瘤的生长[26-28];在黄酒酵母的改造选育过程中,通过降低精氨酸酶的表达量能够降低发酵液中尿素的含量[29]。玉米精氨酸酶的深入研究也发现,精氨酸酶的过量表达能够协调作物的氮素循环,提高作物产量[30]。而工业上L-鸟氨酸的生产也开始从起始的工业合成和发酵法向精氨酸酶法转移。目前已有许多文献报道了不同来源的精氨酸酶的分离和性质研究,因为酶学性质的限制,能够应用于工业用途的精氨酸酶相对较少。本研究室从前期分离到的1 株枯草芽孢杆菌的近缘菌株B. tequilensis PanD37菌株中克隆到一个精氨酸酶基因,该基因全长891 bp,编码297 个氨基酸。酶活力分析显示,该酶的最适催化温度为45 ℃,最适pH值为10.0,而且该酶的稳定性较好,大肠杆菌中表达量高,通过诱导温度的调整能够使重组酶可溶性表达,保持了酶活力,具有一定的应用价值。通过反应条件的优化可以进一步提高产酶及催化能力,适合用于L-鸟氨酸的工业化生产。
参考文献:
[1] LEE H, YOON S, JANG H, et al. Effects of mixing on fedbatch fermentation of L-ornithine[J]. Journal of Bioscience and Bioengineering, 2000, 89(6): 539-544. DOI:10.1016/S1389-1723(00)80053-5.
[2] 万红贵, 陆彬, 蔡恒, 等. L-鸟氨酸的制备及应用[J]. 生物加工过程,2008, 6(1): 12-16. DOI:10.3969/j.issn.1672-3678.2008.01.003.
[3] ABID S, JAFRI W, MUMTAZ K, et al. Efficacy of L-ornithine-L-aspartate as an adjuvant therapy in cirrhotic patients with hepatic encephalopathy[J]. Journal of the College of Physicians and Surgeons Pakistan, 2011, 21(11): 666-671. DOI:11.2011/JCPSP.666671.
[4] SONG W, NIU P, CHEN X, et al. Enzymatic production of L-ornithine from L-arginine with recombinant thermophilic arginase[J]. Journal of Molecular Catalysis B Enzymatic, 2014, 110: 1-7. DOI:10.1016/j.molc atb.2014.09.005.
[5] NESMEYANOV A N, FREIDLINA R K, PETROVA R G. Preparation of DL-proline and DL-ornithine from 1,1,1,5-tetrachloropentane[J].Russian Chemical Bulletin, 1957, 6(4): 459-466. DOI:10.1007/BF01171966.
[6] STROJNY R A, WHITE H C. Preparation of ornithine from methyl 2,5-diazidovalerate[J]. The Journal of Organic Chemistry, 1963, 28(7):1942-1943. DOI:10.1021/jo01042a534.
[7] 万红贵, 叶慧, 陆彬. L-鸟氨酸发酵培养基的中心复合法优化[J].食品与发酵工业, 2006, 32(1): 37-40. DOI:10.3321/j.issn:0253-990X.2006.01.009.
[8] 苏国成, 黄爱清, 蔡慧农. DPS数据处理系统在L-鸟氨酸发酵培养基优化中的应用[J]. 食品科学, 2006, 27(10): 402-406. DOI:10.3321/j.issn:1002-6630.2006.10.098.
[9] MAKRYALEAS K, DRAUZ K. Method for the preparation of salt of L-ornithine: US 5405761[P]. 1995-11-04[2017-03-01]. http://www.google.com/patents/US5405761.
[10] 焦庆才, 李加友, 曹瑜. 固定化细胞酶法制备L-鸟氨酸的方法: CN 166102[P]. 2005-08-31[2017-03-01]. http://www.google.com/patents/CN1661026.
[11] KINOSHITA S, NAKAYAMA K, UDUKA S. The fermentatve production of L-ornithine (preliminary report)[J]. Journal of General and Applied Microbiology, 1957, 3(4): 276-277. DOI:10.2323/jgam.3.276.
[12] 李欢, 周磊, 陈悦, 等. L-鸟氨酸的混合菌种发酵研究[J]. 工业微生物, 2006, 36(4): 37-42. DOI:10.3969/j.issn.1001-6678.2006.04.008.
[13] MORRIS S M. Regulation of enzymes of the urea cycle and arginine metabolism[J]. Annual Review of Nutrition, 2002, 22(1): 87-105.DOI:10.1146/annurev.nutr.22.110801.140547.
[14] MORRIS S M. Recent advances in arginine metabolism: roles and regulation of the arginases[J]. British Journal of Pharmacology, 2009,157(6): 922-930. DOI:10.1111/j.1476-5381.2009.00278.x.
[15] MUMENTHALER S M, YU H, TZE S, et al. Expression of arginase II in prostate cancer[J]. International Journal of Oncology, 2008, 32(2):357-365. DOI:10.3892/ijo.32.2.357.
[16] 杨洪强, 高华君. 植物精氨酸及其代谢产物的生理功能[J]. 植物生理与分子生物学学报, 2007, 33(1): 1-8. DOI:10.3321/j.issn:1671-3877.2007.01.001.
[17] TODD C D, COOKE J E, MULLEN R T, et al. Regulation of loblolly pine (Pinus taeda L.) arginase in developing seedling tissue during germi nation and post- germinative growth[J]. Plant Molecular Biology, 2001, 45(5): 555-565. DOI:10.1023/A:1010645616920.
[18] TODD C D, GIFFORD D J. The role of the megagametophyte in maintaining loblolly pine (Pinus taeda L.) seedling arginase gene expression in vitro[J]. Planta, 2002, 215(1): 110-118. DOI:10.1007/s00425-001-0720-2.
[19] WANG M, XU M, RAO Z, et al. Construction of a highly efficient Bacillus subtilis 168 whole-cell biocatalyst and its application in the production of L-ornithine[J]. Journal of Industrial Microbiology &Biotechnology, 2015, 42(11): 1427-1437. DOI:10.1007/s10295-015-1672-z.
[20] KANDA M, OHGISHI K, HANAWA T, et al. Arginase of Bacillus brevis Nagano: purification, properties, and implication in gramicidin S biosynthesis[J]. Archives of Biochemistry and Biophysics, 1997,344(1): 37-42. DOI:10.1006/abbi.1997.0174.
[21] ZHANG T, GUO Y, ZHANG H, et al. Arginase from Bacillus thuringiersis SK 20.001: purification, characteristics, and implication for L-ornithine biosynthesis[J]. Process Biochemistry, 2013, 48(4):663-668. DOI:10.1016/j.procbio.2013.02.023.
[22] GARCÍA D, URIBE E, SALGADO M, et al. Mutagenic and kinetic support for an allosteric site in arginase from the extreme thermophile Bacillus caldovelox, which allows activation by arginine[J]. Biochimie, 2015, 108: 8-12. DOI:10.1016/j.biochi.2014.10.017.
[23] 冯志彬, 张娟, 陈国忠, 等. 产L-天冬氨酸α-脱羧酶细菌的分离、鉴定及发酵条件优化[J]. 微生物学报, 2016, 56(1): 44-55.DOI:10.13343/j.cnki.wsxb.20150169.
[24] CHINARD F P. Photometric estimation of proline and ornithine[J].Journal of Biological Chemistry, 1952, 199(1): 91-95.
[25] SCHNEIDER J, NIERMANN K, WENDISCH V F. Production of the amino acids L-glutamate, L-lysine, L-ornithine and L-arginine from arabinose by recombinant Corynebacterium glutamicum[J].Journal of Biotechnology, 2011, 154(2/3): 191-198. DOI:10.1016/j.jbiotec.2010.07.009.
[26] ENSOR C M, HOLTSBERG F W, BOMALASKI J S, et al. Pegylated arginine deiminase (ADI-SS PEG20,000 mw) inhibits human melanomas and hepatocellular carcinomas in vitro and in vivo[J].Cancer Research, 2002, 62(19): 5443-5450.
[27] FEUN L, SAVARAJ N. Pegylated arginine deiminase: a novel antica ncer enzyme agent[J]. Expert Opinion on Investigational Drugs, 2006,15(7): 815-822. DOI:10.1517/13543784.15.7.815.
[28] 孙冰, 赵浩如, 杜广增. 精氨酸及其相关药物的抗肿瘤作用研究近况[J]. 药学进展, 2007, 31(1): 21-25. DOI:10.3969/j.issn.1001-5094.2007.01.004.
[29] 赵然然, 陆健, 谢广发. 基于PCR方法敲除黄酒酵母精氨酸酶基因的工程菌构建[J]. 食品工业科技, 2012, 33(17): 159-162.
[30] 张佩, 杨小艳, 邸宏, 等. 玉米精氨酸酶基因过表达载体的构建与遗传转化分析[J]. 玉米科学, 2012, 20(6): 42-48. DOI:10.3969/j.issn.1005-0906.2012.06.009.