
图1 不同链长脂肪酸存在情况下及未加脂肪酸酵母菌株的基因转录情况分析
Fig. 1 Transcriptional analysis of S. cerevisiae in the presence and absence of fatty acids with different chain lengths
a.样品间相关性分析热图;b.样品间维恩图;c.样品间层次聚类分析图。对照表示未加脂肪酸的酿酒酵母菌株。
酿酒酵母(Sacchromyces cerevisiae)是一种重要的食品安全级微生物[1-2],目前通过酿酒酵母可以生物合成多种脂肪酸及脂肪酸类衍生物如脂肪酸乙酯[3-4]、脂肪醇[5-6]及油脂等[7-10]。脂肪酸乙酯及脂肪醇可以作为香料物质添加到食品当中[11],如己酸乙酯是浓香型白酒香气的重要成分之一,而己醇被研究证明是桑葚香气成分的组成之一[12-14]。通过酿酒酵母合成脂肪酸类衍生物如己酸乙酯等首先需要酿酒酵母合成脂肪酸,再进一步通过不同的酶催化合成不同的化合物,而研究表明酿酒酵母体内游离的脂肪酸主要包括C18及C16的饱和及不饱和脂肪酸[15-16],短链及中长链脂肪酸的含量相对较少[17-18],因此限制其下游短链及中长链脂肪酸酯及脂肪醇的产量。如何控制酿酒酵母体内游离脂肪酸的链长,促进短链及中长链脂肪酸的合成,是提高酿酒酵母生物合成短链及中长链脂肪酸酯类及脂肪醇类等化合物首先需要解决的问题。
有研究提出在大肠杆菌中建立动态感应调控系统的概念[19-20],其原理主要基于脂肪酸进入细胞内会转化为脂酰基辅酶A,脂酰基辅酶A与转录因子FadR结合,从而开启FadR抑制的启动子启动其下游基因的转录。通过将这种受调控的启动子用于大肠杆菌脂肪酸合成及由脂肪酸起始的脂肪酸乙酯合成途径中,胞内脂肪酸处于一种动态平衡态,细胞在感应脂肪酸的同时受其调控,其结果可增加产物脂肪酸乙酯的同时避免脂肪酸过度积累造成的一系列问题,同时减少副产物的积累。Dahl等[19]进一步通过转录组测序等在大肠杆菌中筛选到可响应法呢基焦磷酸胁迫的启动子作为动态感应调控元件,提高了目标产物青蒿酸的积累并减少中间产物的量。Torella等[21]也在大肠杆菌中发现了通过类似的调控策略动态调控脂肪酸链长从而达到截短脂肪酸链长的目的。Xu Peng等[22]在大肠杆菌中设计并获得响应丙二酰单酰基辅酶A调控元件,建立了在胞内响应丙二酰单酰基辅酶A的动态感应调控系统,脂肪酸的产量提高2.1 倍,达到3.8 g/L。
目前,酿酒酵母中源于脂肪酸的动态感应调控系统方面的报道较少[23]。这主要是因为酿酒酵母中脂肪酸的合成及链长延伸机制还未被明确阐述清楚[24-25],酿酒酵母中响应脂肪酸的转录因子及转运蛋白等还未被研究清楚[26],因此,本实验将代表3 种不同链长的脂肪酸己酸(C6)、十二烷酸(C12)及十六烷酸(C16)分别添加到对数期的酿酒酵母培养液中,通过转录组测序分析其对酿酒酵母基因转录水平的影响,对不同链长脂肪酸存在下酿酒酵母差异基因的表达进行分析,并对可能参与脂肪酸合成的转录因子及转运蛋白进行预测,最后进一步分析3 种不同链长脂肪酸存在下,酵母脂肪酸合成途径中关键酶基因的表达情况,为进一步阐明酿酒酵母响应不同链长脂肪酸的动态感应调控元件及其作用机制提供支持。
实验中用到的酿酒酵母为BY4741(实验室保存)。
Total RNA Extractor(Trizol) 生工生物工程(上海)股份有限公司;Qubit2.0 RNA检测试剂盒、Qubit2.0 DNA检测试剂盒 美国Thermo Fisher公司。
摇床、超净台、台式高速冷冻离心机、聚合酶链式反应(polymerase chain reaction,PCR)仪 美国Bio-Rad公司;NanoDrop 2000C超微量分光光度计、电泳仪北京市六一仪器厂。
1.3.1 培养基配制
YPD培养基:葡萄糖10 g/L,酵母粉10 g/L,蛋白胨20 g/L,pH值自然。
发酵培养基:胰蛋白胨10 g/L,酵母粉5 g/L,葡萄糖5 g/L,氯化钠10 g/L。
1.3.2 不同链长脂肪酸加入酵母细胞中的浓度及诱导时间确定
根据文献报道并结合本实验的要求[19,27-28],将不同浓度的脂肪酸加入到酵母细胞中,通过OD600 nm测定酵母细胞的生长情况,选取对酵母细胞生长影响较为一致的脂肪酸浓度作为最终的浓度加入酵母处于对数生长期的酵母培养基中,诱导培养6 h。其中C6终浓度为1 mmol/L,C12浓度为1 mmol/L,C16由于其存在一定溶解性的问题,最终确定浓度为1 mmol/L。在实验过程选取30 mL培养基中加入600 μL混合的异丙醇-曲拉通X-100(3∶2)作为脂肪酸在酵母培养基中的助溶剂,其中对照组酵母中加入相同比例及体积的助溶剂以消除转录组测序过程中助溶剂产生的影响。
1.3.3 总RNA的提取
总RNA提取方法按照试剂盒说明书进行。通过Trizol提取试剂盒进行RNA提取,利用Nanodrop检测RNA浓度及琼脂糖凝胶检测RNA完整性以及基因组污染情况。检测合格的RNA用于后续测序。
1.3.4 转录组测序数据处理及分析
将提取后的RNA送入华大基因进行转录组测序分析,同时实验设置2 个重复。使用R软件里的cor函数进行相关性分析,使用R软件里的hclust函数进行层次聚类分析,使用维恩图展示样品间基因表达分布。转录因子及转运蛋白等的预测分析由R软件中的phyper函数进行富集分析,此外,不同样本间的基因表达差异均已RNA测序后的试剂读数比值取log2的值来展示。

图1 不同链长脂肪酸存在情况下及未加脂肪酸酵母菌株的基因转录情况分析
Fig. 1 Transcriptional analysis of S. cerevisiae in the presence and absence of fatty acids with different chain lengths
a.样品间相关性分析热图;b.样品间维恩图;c.样品间层次聚类分析图。对照表示未加脂肪酸的酿酒酵母菌株。
在确保所提样品RNA的质量及通过Nanodrop测定RNA的纯度之后,对加入不同脂肪酸的酵母RNA样本进行转录组测序,将所得数据结果对3 种不同链长脂肪酸存在时基因的转录情况进行归类比较分析后发现,未加任何脂肪酸的酵母与C12脂肪酸存在时相关性最高,C16脂肪酸存在时次之,C6脂肪酸存在时其与对照菌株、C12及C16脂肪酸存在时相关性最低。进一步根据基因转录信息对各样品进行聚类分析,发现这4 个样品的聚类关系依次为C6、C16、C12及对照(图1a、c)。进一步对C6、C12及C16存在时酵母的转录情况进行分析后发现,加入脂肪酸C6、C12及C16的酵母均有5 553 个转录本被检测到(包括预测的新转录本),C6特异性的转录本包括132 个,C12特异性的转录本90 个,C16特异性转录本63 个,C12与C16共同转录本145 个,C6与C12共同转录本67 个,C6与C16共同转录本95 个(图1b)。
在本实验中,选取C6、C12以及C16脂肪酸分别代表不同链长的脂肪酸,通过转录组测序发现C6及C12相比差异最为不同,其中上调基因为466 个而下调基因为1 087 个,而与长链的脂肪酸如C16相比则上调307 个,下调733 个(图2)。与对照未加脂肪酸菌株BY4741相比,加入脂肪酸之后,其中C6对酿酒酵母影响最大(上调基因591 个,下调基因317 个),此外,从数据分析来看,不同链长的脂肪酸加入酵母细胞之后,酵母胞内的基因表达差异更为明显,这提示酵母细胞对不同链长的脂肪酸表现出不同的反应机制。
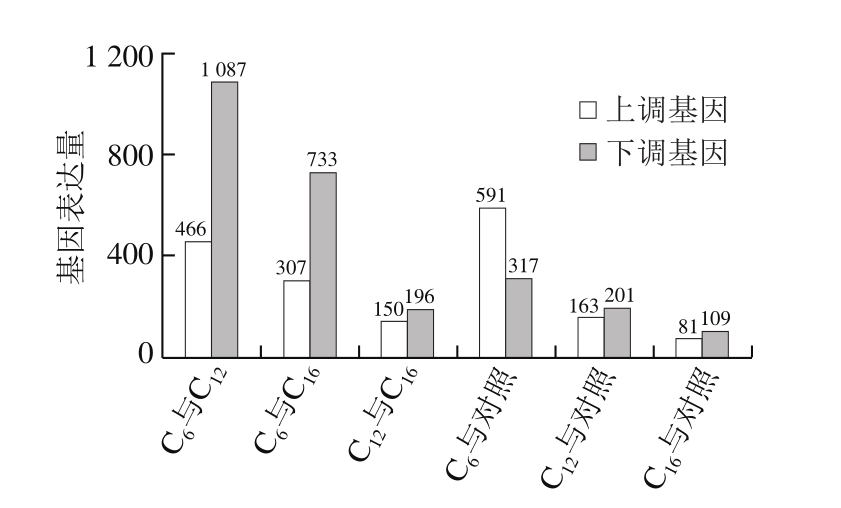
图2 不同脂肪酸存在条件下的酿酒酵母基因表达差异情况分析
Fig. 2 Differential expression of genes in S. cerevisiae in the presence and absence of fatty acids with different chain lengths
目前,已有报道包括酿酒酵母对多不饱和脂肪酸的响应[23]以及酿酒酵母对抗辛酸(C8)及癸酸(C10)的反应机制[24-25]等。本研究将结合已有的资料进一步分析其对不同链长脂肪酸的响应情况等。分别与对照未加入任何脂肪酸的菌株相比,3 种不同链长的脂肪酸共使得21 个基因上调及18 个基因下调(选取log2比值不小于1的进行统计)。C6、C12及C16脂肪酸均显著促进UTP15、URA7、FDR4、PET117等基因的上调,在3 种不同链长的脂肪酸存在条件下,其与对照菌株相比,上调的倍数较为一致,这体现出3 种不同链长脂肪酸共同促进基因上调的趋势较为一致。而对于3 种链长脂肪酸均使得转录水平下调的基因来说,C6、C12及C16均显著抑制基因TOD6、TRM2、FEX1、STH1、RAD7、HEK2、POB3、NUP53、FMP49等的转录,而C6明显抑制PHO5、LCP5及POL31基因的转录,C12则明显抑制CRP1基因的转录(表1)。同时,实验还对不同脂肪酸对酿酒酵母基因转录的影响进行了比较,C6与C12均会使得RCK2、YBR056W、YBR287W、PEX5、POX1、SAM50、PUS2、GPB2基因转录水平上调,SMM1、ETS1-1、ETS1-2、YLR256W-A、YIL156W-B、snR31、NME1、CEX1、MSG5、GFD1基因转录水平下调。C6与C16对酿酒酵母转录影响一致的基因包括37 个,其中RDH54、MRP10、ATG34、NST1、PHM8、MRPL32、SLU7、UBP10、SPC72、SNA2、RGD2基因上调,IMD4、ADR1、RPA190、CHS2、FAS1等26 个基因下调。C12与C16对酿酒酵母转录影响一致的基因包括50 个,其中16 个上调,34 个下调。在此基础上,剔除掉上述各个脂肪酸影响一致的基因之后,分别对C6、C12及C16脂肪酸分别对酵母基因转录水平的影响进行分析(除去转录组测序时转录条数小于10,Read counts小于10的基因),发现C6特异性影响酿酒酵母的基因数为165 个,C12为40 个,C16为5 个。
表1 C6、C12及C16对酿酒酵母转录水平影响一致的基因
Table 1 Transcription levels of genes inS. cerevisiae consistently affected by C6, C12 and C16
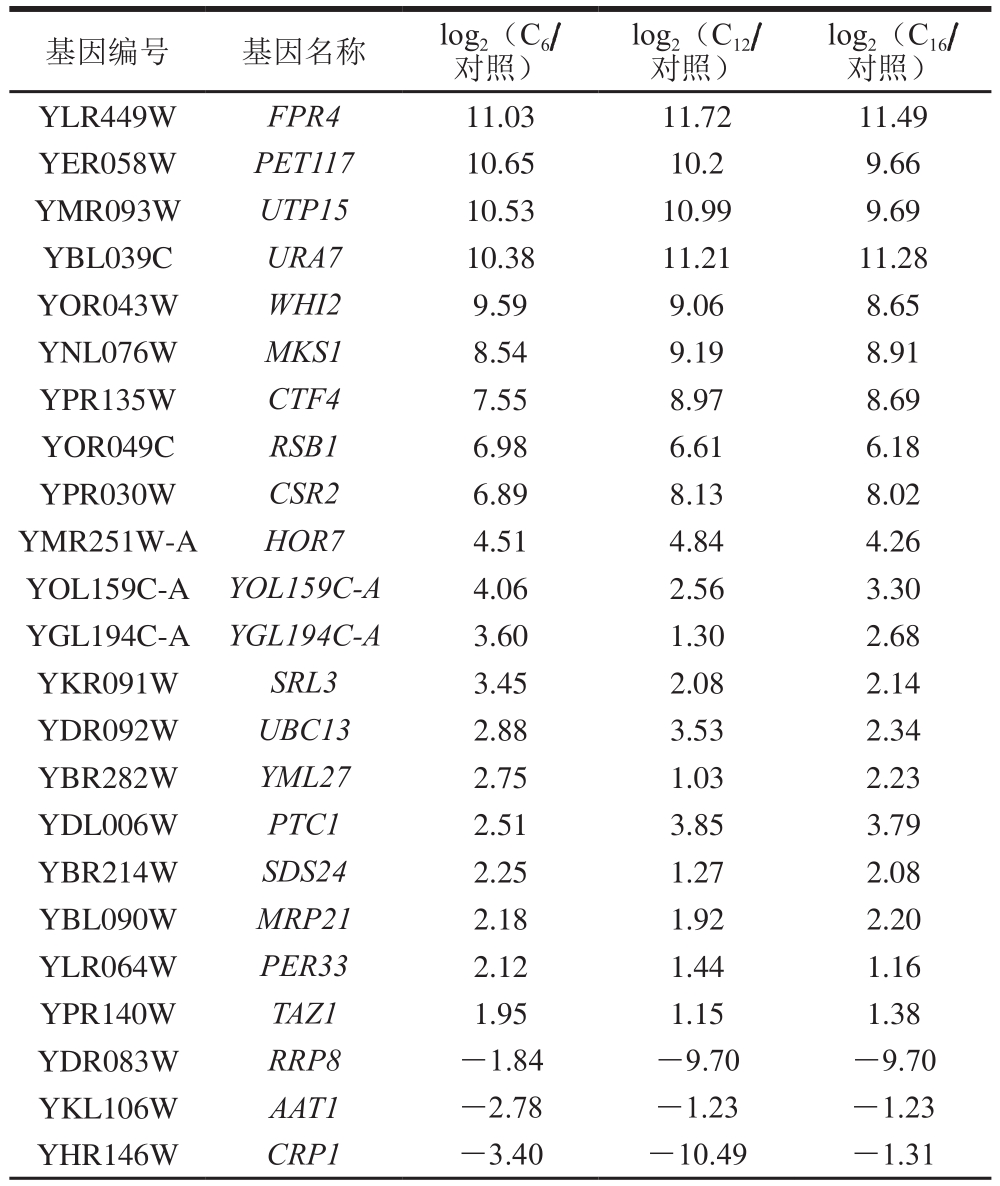
log2(C12/对照)基因编号 基因名称 log2(C6/对照)log2(C16/对照)YLR449WFPR4 11.03 11.72 11.49 YER058WPET117 10.65 10.2 9.66 YMR093WUTP15 10.53 10.99 9.69 YBL039CURA7 10.38 11.21 11.28 YOR043WWHI2 9.59 9.06 8.65 YNL076WMKS1 8.54 9.19 8.91 YPR135WCTF4 7.55 8.97 8.69 YOR049CRSB1 6.98 6.61 6.18 YPR030WCSR2 6.89 8.13 8.02 YMR251W-AHOR7 4.51 4.84 4.26 YOL159C-AYOL159C-A 4.06 2.56 3.30 YGL194C-AYGL194C-A 3.60 1.30 2.68 YKR091WSRL3 3.45 2.08 2.14 YDR092WUBC13 2.88 3.53 2.34 YBR282WYML27 2.75 1.03 2.23 YDL006WPTC1 2.51 3.85 3.79 YBR214WSDS24 2.25 1.27 2.08 YBL090WMRP21 2.18 1.92 2.20 YLR064WPER33 2.12 1.44 1.16 YPR140WTAZ1 1.95 1.15 1.38 YDR083WRRP8 -1.84 -9.70 -9.70 YKL106WAAT1 -2.78 -1.23 -1.23 YHR146WCRP1 -3.40 -10.49 -1.31
续表1
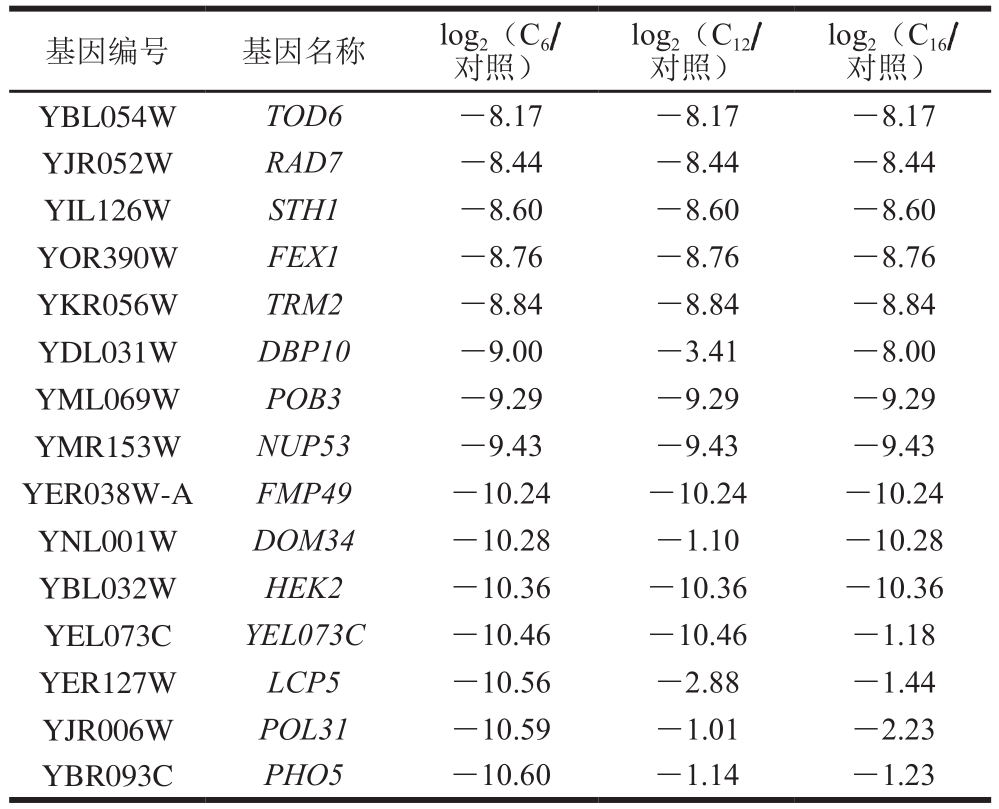
log2(C12/对照)基因编号 基因名称 log2(C6/对照)log2(C16/对照)YBL054WTOD6 -8.17 -8.17 -8.17 YJR052WRAD7 -8.44 -8.44 -8.44 YIL126WSTH1 -8.60 -8.60 -8.60 YOR390WFEX1 -8.76 -8.76 -8.76 YKR056WTRM2 -8.84 -8.84 -8.84 YDL031WDBP10 -9.00 -3.41 -8.00 YML069WPOB3 -9.29 -9.29 -9.29 YMR153WNUP53 -9.43 -9.43 -9.43 YER038W-AFMP49 -10.24 -10.24 -10.24 YNL001WDOM34 -10.28 -1.10 -10.28 YBL032WHEK2 -10.36 -10.36 -10.36 YEL073CYEL073C -10.46 -10.46 -1.18 YER127WLCP5 -10.56 -2.88 -1.44 YJR006WPOL31 -10.59 -1.01 -2.23 YBR093CPHO5 -10.60 -1.14 -1.23
在生物体内,转录因子的表达量相对较少,通过转录组测序并进一步分析转录数据,发现3 种不同链长的脂肪酸存在条件下对于酿酒酵母体内的转录因子的转录水平是有影响的,共有17 条转录因子的转录受到了影响(表2),其中HMRA1受到3 种链长脂肪酸的影响后转录明显上调。脂肪酸C6及C16促进GSM1、SWI4、TEC1、RSC3转录因子的转录,短链脂肪酸C6与长链脂肪酸C16相比明显促进上述基因的转录,中长链脂肪酸C12则抑制以上转录因子的转录;而同样C6及C16抑制CUP2、DUR1,2的转录,C12则相反使得其上调。此外,C6特异性促进CIN5、YAP7、VPS72、HMLALPHA2、MATALPHA2、CAD1转录因子的转录(C12及C16抑制)而抑制ARG80的转录。3 种脂肪酸共同使得MCM1转录水平下调。
表2 3 种不同链长脂肪酸对酿酒酵母转录因子转录水平的影响
Table 2 Inf l uence of fatty acids with different lengths on transcript factors inS. cerevisiae
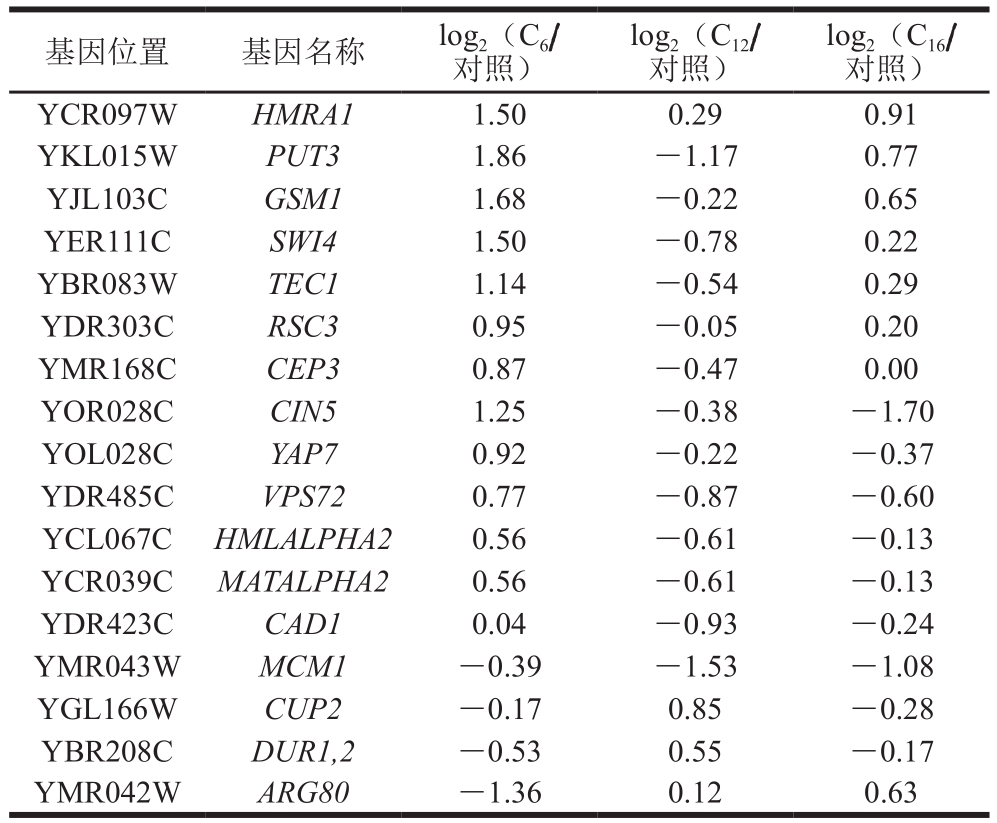
log2(C16/对照)YCR097WHMRA1 1.50 0.29 0.91 YKL015WPUT3 1.86 -1.17 0.77 YJL103CGSM1 1.68 -0.22 0.65 YER111CSWI4 1.50 -0.78 0.22 YBR083WTEC1 1.14 -0.54 0.29 YDR303CRSC3 0.95 -0.05 0.20 YMR168CCEP3 0.87 -0.47 0.00 YOR028CCIN5 1.25 -0.38 -1.70 YOL028CYAP7 0.92 -0.22 -0.37 YDR485CVPS72 0.77 -0.87 -0.60 YCL067CHMLALPHA2 0.56 -0.61 -0.13 YCR039CMATALPHA2 0.56 -0.61 -0.13 YDR423CCAD1 0.04 -0.93 -0.24 YMR043WMCM1 -0.39 -1.53 -1.08 YGL166WCUP2 -0.17 0.85 -0.28 YBR208CDUR1,2 -0.53 0.55 -0.17 YMR042WARG80 -1.36 0.12 0.63基因位置 基因名称 log2(C6/对照)log2(C12/对照)
进一步对3 种链长脂肪酸存在条件下,对转运蛋白的基因转录情况进行分析后发现,共得到受3 种脂肪酸影响的转运蛋白有61 个,进一步对其中40 个转运蛋白进行分析(表3),其中11 个基因均受C6、C12及C16的影响,ALR1、RIP1、MRH1、QCR9、ATR7及QCR7分别被上调而KAP120、DNF2、THI7、FMP49及MMP1被下调;C6及C12均会导致APL1、TRK1、QCR8及QCR6基因转录水平的上调而C12及C16对转运蛋白影响相一致的基因包括12 个,其中3 个上调,9 个下调;此外,C6特异性影响8 个转运蛋白的转录,C12特异性影响3 个而C16则特异性影响2 个。
表3 3 种不同链长脂肪酸对酿酒酵母中转运蛋白转录水平的影响
Table 3 Inf l uence of fatty acids with different chain lengths on transporter proteins in S. cerevisiae
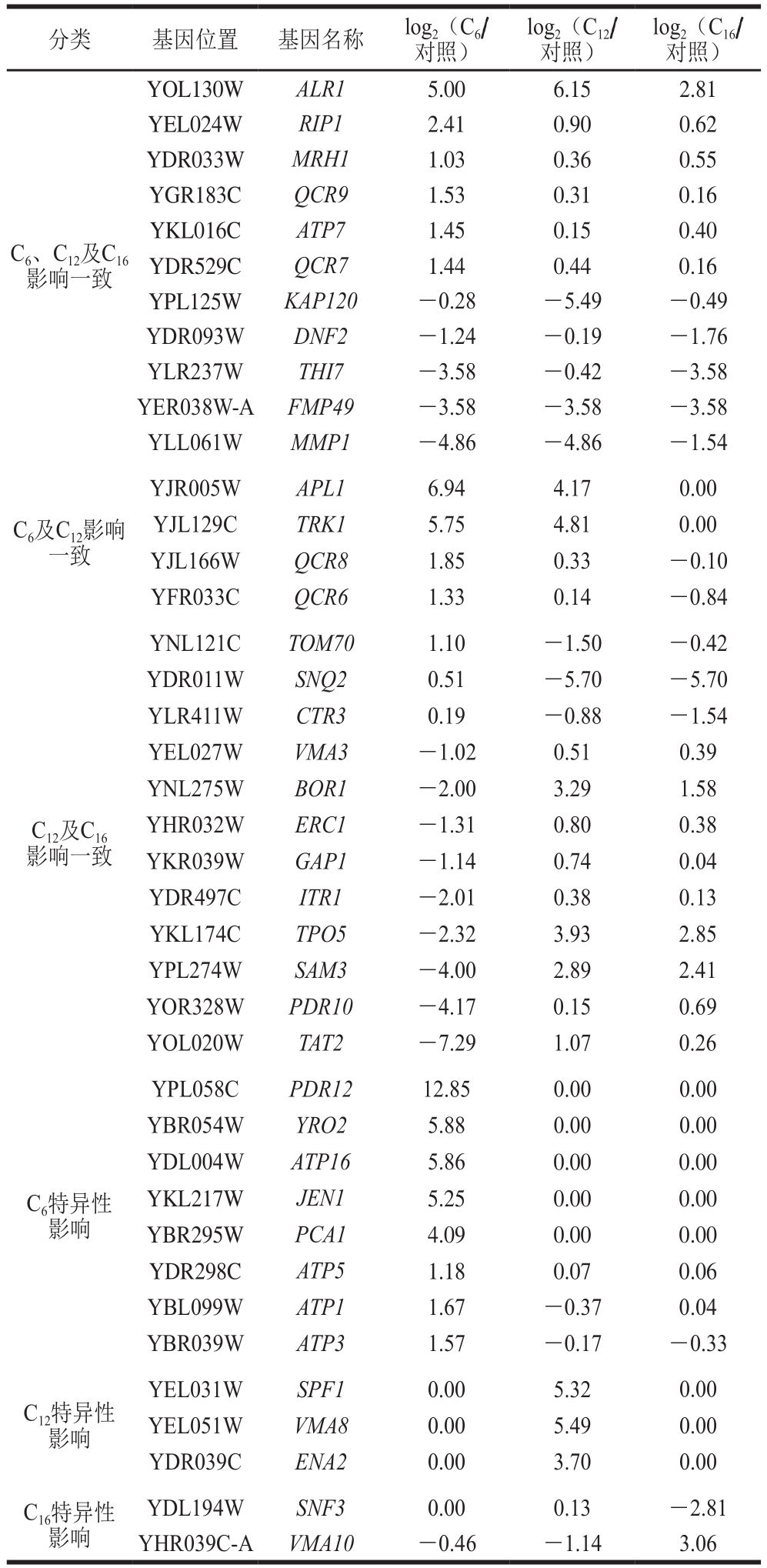
分类 基因位置 基因名称 log2(C6/对照)log2(C12/对照)log2(C16/对照)C6、C12及C16影响一致YOL130WALR1 5.00 6.15 2.81 YEL024WRIP1 2.41 0.90 0.62 YDR033WMRH1 1.03 0.36 0.55 YGR183CQCR9 1.53 0.31 0.16 YKL016CATP7 1.45 0.15 0.40 YDR529CQCR7 1.44 0.44 0.16 YPL125WKAP120 -0.28 -5.49 -0.49 YDR093WDNF2 -1.24 -0.19 -1.76 YLR237WTHI7 -3.58 -0.42 -3.58 YER038W-AFMP49 -3.58 -3.58 -3.58 YLL061WMMP1 -4.86 -4.86 -1.54 C6及C12影响一致YJR005WAPL1 6.94 4.17 0.00 YJL129CTRK1 5.75 4.81 0.00 YJL166WQCR8 1.85 0.33 -0.10 YFR033CQCR6 1.33 0.14 -0.84 C12及C16影响一致YNL121CTOM70 1.10 -1.50 -0.42 YDR011WSNQ2 0.51 -5.70 -5.70 YLR411WCTR3 0.19 -0.88 -1.54 YEL027WVMA3 -1.02 0.51 0.39 YNL275WBOR1 -2.00 3.29 1.58 YHR032WERC1 -1.31 0.80 0.38 YKR039WGAP1 -1.14 0.74 0.04 YDR497CITR1 -2.01 0.38 0.13 YKL174CTPO5 -2.32 3.93 2.85 YPL274WSAM3 -4.00 2.89 2.41 YOR328WPDR10 -4.17 0.15 0.69 YOL020WTAT2 -7.29 1.07 0.26 C6特异性影响YPL058CPDR12 12.85 0.00 0.00 YBR054WYRO2 5.88 0.00 0.00 YDL004WATP16 5.86 0.00 0.00 YKL217WJEN1 5.25 0.00 0.00 YBR295WPCA1 4.09 0.00 0.00 YDR298CATP5 1.18 0.07 0.06 YBL099WATP1 1.67 -0.37 0.04 YBR039WATP3 1.57 -0.17 -0.33 C12特异性影响YEL031WSPF1 0.00 5.32 0.00 YEL051WVMA8 0.00 5.49 0.00 YDR039CENA2 0.00 3.70 0.00 C16特异性影响YDL194WSNF3 0.00 0.13 -2.81 YHR039C-AVMA10 -0.46 -1.14 3.06
酿酒酵母脂肪酸合成途径包括前期脂肪酸合成到16 个碳脂肪酸之后进入长链脂肪酸的合成,而脂肪酸机体需要时可以通过β氧化途径进一步降解为乙酰辅酶A(图3),通过转录组测序,进一步比较分析不同链长脂肪酸存在条件下,酿酒酵母脂肪酸的合成及降解途径受到的影响,发现与对照未加任何脂肪酸酵母菌株相比,3 种不同链长的脂肪酸存在条件下,8 个基因的转录水平受到了影响,其中除2 个不饱和脂肪酸降解所需的异构酶ECI1及ECI2外,其他6 个基因在图3中标注了其作用位点。从图3可发现,以C12为代表的中长链脂肪酸在整个酿酒酵母的脂肪酸合成及降解过程中均起到促进基因转录的作用,如ACC1、FAS1、FAS2、ELO1、FAA3及POX1;而短链脂肪酸C6则抑制脂肪酸的合成过程中ACC1、FAS1及FAS2的转录及长链脂肪酸的合成相关基因Elo1,C6却促进脂肪酸降解途径β氧化途径关键酶基因POX1的转录水平上调;C16与C6作用相似,但是其与C6脂肪酸不同的是,C16促进长链脂肪酸的合成,抑制了脂肪酸到乙酰辅酶A的降解过程。
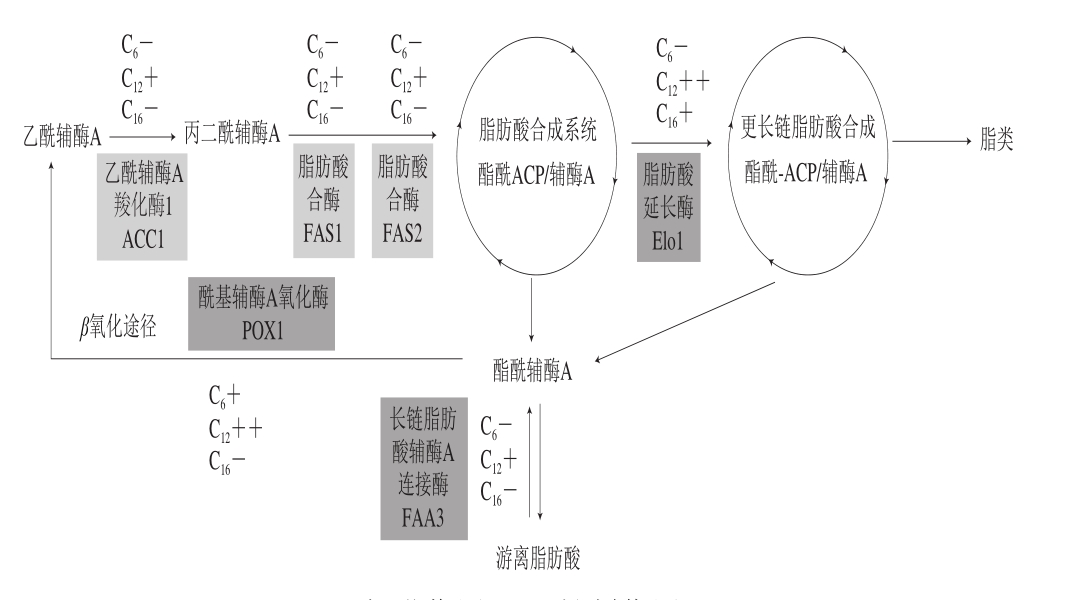
图3 3 种不同链长脂肪酸对酿酒酵母脂肪酸合成及降解途径的影响
Fig. 3 Effects of fatty acids with different chain lengths on fatty acid biosynthesis and degradation pathways in S. cerevisiae
+.促进作用;-.抑制作用。
本研究通过转录组测序分析了不同链长脂肪酸对于酿酒酵母基因转录水平的影响,结果发现3 种脂肪酸中短链脂肪酸如C6对酵母基因的转录水平影响最大,相比于C12及C16脂肪酸,C6对酵母细胞的形成过程、细胞膜的组成以及细胞的生长及凋亡等影响最为明显,如MIC27、RGI1、PIC2、INH1、ATP1、YPR010C-A、ATP3、EXO70、MGM1、IRR1、YKR078W、MCR1、SST2、CLB4、GRX1、STE2、PEP12、GIN4、COX12、TOM70、ATP25、RNY1、CMK2、AIM24、GDI1、ISM1、COA6基因转录水平明显上调,而ALG3、TMA19、EMC3、SUR7、MCH5、RPS27B、HUT1、ERC1、ARG80、HEM1、PPX1、MDE1、NAP1、ARN2、RCL1、RPS21A、SMX3、EFM4、DUT1、PDR10、AIM46等基因则下调,C6脂肪酸存在条件下,细胞受到胁迫后的应激基因如FUS2、HSP82、HSP78、HSP104均明显上调;此外,C6脂肪酸存在时一些基因在基因信息的传递过程如结合、折叠、分选及降解过程、转录过程及翻译调控过程等受到影响,这些基因包括RIE1、TPH3、TFA1、MSC1、HRR25、NMD2、ZTA1、COX6、DOG2、BAR1、DOA4、SKO1、MSH5、APL2、TMC1、MSS1、HFD1、RPL36B、YER152C、RPL23B、ADO1、RPA12、ITR1、OPI3等,此类基因中明显受C12影响的基因则包括RPN8、HDA2、MKT1、YKR070W等,相比之下,这类基因中受C16脂肪酸影响的较少;3 种脂肪酸存在条件下除脂肪酸代谢途径受到影响,糖的代谢途径会受到一定影响,如C6导致SOL4、MDH1基因明显上调,而C12导致GSC2、INO1、PRM7基因明显下调;同时,C6影响氨基酸代谢途径中TDA10、MDE1基因上调,而C12明显使得TYR1基因上调;值得关注的是,C16特异性影响基因HAM1明显上调,HAM1为三磷酸肌苷焦磷酸酶,为细胞中能量储存过程中的酶[27-30]。
通过转录组测序,对一些可能的转录因子及转运蛋白进行分析及预测,在生物体内,转录因子的表达量并不多,在后续的实验中,将进一步研究不同脂肪酸存在下,分析差异表达转录因子的功能,同时将结合不同脂肪酸的表达差异基因进一步寻找不同的转录因子[27,30]。而对于转运蛋白的差异,后续的实验需要进一步对其验证,转运蛋白对于酿酒酵母生物合成脂肪酸途径具有重要意义。
最后本研究分析不同脂肪酸存在下,酿酒酵母脂肪酸合成途径受到的影响,研究发现短链及中长链脂肪酸可一定程度上促进β氧化途径的进行,中长链脂肪酸促进整个脂肪酸合成及延伸途径的进行,而长链脂肪酸促进长链脂肪酸的延伸过程,对于此现象的机理还需要进一步研究。
本研究通过高通量测序分析不同链长脂肪酸存在条件下,酿酒酵母体内基因转录的差异,为后续研究酿酒酵母脂肪酸合成途径的动态感应调控元件提供了丰富的数据资源。
[1] ABBOTT D A, ZELLE R M, PRONK J T, et al. Metabolic engineering of Saccharomyces cerevisiae for production of carboxylic acids:current status and challenges[J]. FEMS Yeast Research, 2009, 9: 1123-1136. DOI:10.1111/j.1567-1364.2009.00537.x.
[2] LIAN J, MISHRA S, ZHAO H. Recent advances in metabolic engineering of Saccharomyces cerevisiae: new tools and their applications[J]. Metabolic Engineering, 2018, 50: 85-108.DOI:10.1016/j.ymben.2018.04.011.
[3] ZHOU Y J, BUIJS N A, ZHU Z, et al. Production of fatty acid-derived oleochemicals and biofuels by synthetic yeast cell factories[J]. Nature Communications, 2016, 7: 11709. DOI:10.1038/ncomms11709.
[4] GUO W, SHENG J, ZHAO H, et al. Metabolic engineering of Saccharomyces cerevisiae to produce 1-hexadecanol from xylose[J].Microbial Cell Factories, 2016, 15: 24. DOI:10.1186/s12934-016-0423-9.
[5] JONG B W, SHI S, VALLE-RODRíGUEZ J O, et al. Metabolic pathway engineering for fatty acid ethyl ester production in Saccharomyces cerevisiae using stable chromosomal integration[J].Journal of Industrial Microbiology & Biotechnology, 2015, 42: 477-486. DOI:10.1007/s10295-014-1540-2.
[6] CHEN Y, LI F, GUO J, et al. Enhanced ethyl caproate production of Chinese liquor yeast by overexpressing EHT1 with deleted FAA1[J].Journal of Industrial Microbiology & Biotechnology, 2014, 41: 563-572. DOI:10.1007/s10295-013-1390-3.
[7] LEBER C, POLSON B, FERNANDEZ-MOYA R, et al.Overproduction and secretion of free fatty acids through disrupted neutral lipid recycle in Saccharomyces cerevisiae[J]. Metabolic Engineering, 2015, 28: 54-62. DOI:10.1016/j.ymben.2014.11.006.
[8] GAJEWSKI J, BUELENS F, SERDJUKOW S. Engineering fatty acid synthases for directed polyketide production[J]. Nature Chemical Biology, 2017, 13: 363-365. DOI:10.1038/nchembio.2314.
[9] LEBER C, DA SILVA N A. Engineering of Saccharomyces cerevisiae for the synthesis of short chain fatty acids[J]. Biotechnology and Bioengineering, 2014, 111: 347-358. DOI:10.1002/bit.26288.
[10] ZHU Z, ZHOU Y J, KANG M K, et al. Enabling the synthesis of medium chain alkanes and 1-alkenes in yeast[J]. Metabolic Engineering, 2017, 44: 81-88. DOI:10.1016/j.ymben.2017.09.007.
[11] BRAULT G, SHARECK F, HURTUBISE Y, et al. Short-chain fl avor ester synthesis in organic media by an E. coli whole-cell biocatalyst expressing a newly characterized heterologous lipase[J]. PLoS ONE,2014, 9(3): e91872. DOI:10.1371/journal.pone.0091872.
[12] SU L, HONG R, GUO X, et al. Short-chain aliphatic ester synthesis using Thermobifida fusca cutinase[J]. Food chemistry, 2016, 206: 131-136. DOI:10.1016/j.foodchem.2016.03.051.
[13] 于怀龙, 马永昆, 张荣, 等. 不同品种桑椹香气成分的主成分分析[J]. 食品工业科技, 2016, 37(10): 62-66. DOI:10.13386/j.issn1002-0306.2016.10.003.
[14] 李秋棉, 罗均, 李雪萍, 等. 果实香气物质的合成与代谢研究进展[J].广东农业科学, 2012, 39(19): 104-107. DOI:10.16768/j.issn.1004-874x.2012.19.061.
[15] LIAN J, ZHAO H. Recent advances in biosynthesis of fatty acids derived products in Saccharomyces cerevisiae via enhanced supply of precursor metabolites[J]. Journal of Industrial Microbiology &Biotechnology, 2015, 42: 437-451. DOI:10.1007/s10295-014-1518-0.
[16] TEHLIVETS O, SCHEURINGER K, KOHLWEIN S D. Fatty acid synthesis and elongation in yeast[J]. Biochimica et Biophysica Acta,2007, 1771: 255-270. DOI:10.1016/j.bbalip.2006.07.004.
[17] SARRIA S, KRUYER N S, PERALTA-YAHYA P. Microbial synthesis of medium-chain chemicals from renewables[J]. Nature Biotechnology,2017, 35: 1158-1166. DOI:10.1038/nbt.4022.
[18] GAJEWSKI J, PAVLOVIC R, FISCHER M, et al. Engineering fungal de novo fatty acid synthesis for short chain fatty acid production[J].Nature Communications, 2017, 8: 14650. DOI:10.1038/ncomms14650.
[19] DAHL R H, ZHANG F, ALONSO-GUTIERREZ J, et al. Engineering dynamic pathway regulation using stress-response promoters[J].Nature Biotechnology, 2013, 31: 1039-1046. DOI:10.1038/nbt.2689.
[20] ZHANG F, CAROTHERS J M, KEASLING J D. Design of a dynamic sensor-regulator system for production of chemicals and fuels derived from fatty acids[J]. Nature Biotechnology, 2012, 30: 354-359.DOI:10.1038/nbt.2149.
[21] TORELLA J P, FORD T J, KIM S N, et al. Tailored fatty acid synthesis via dynamic control of fatty acid elongation[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013,110: 11290-11295. DOI:10.1073/pnas.1307129110.
[22] XU P, LI L, ZHANG F, et al. Improving fatty acids production by engineering dynamic pathway regulation and metabolic control[J].Proceedings of the National Academy of Sciences of the United States of America, 2014, 111: 11299-112304. DOI:10.1073/pnas.1406401111.
[23] TEIXEIRA P G, FERREIRA R, ZHOU Y J, et al. Dynamic regulation of fatty acid pools for improved production of fatty alcohols in Saccharomyces cerevisiae[J]. Microbial Cell Factories, 2017, 16: 45.DOI:10.1186/s12934-017-0663-3.
[24] LOMAKIN I B, XIONG Y, STEITZ T A. The crystal structure of yeast fatty acid synthase, a cellular machine with eight active sites working together[J]. Cell, 2007, 129: 319-332.
[25] DENIC V, WEISSMAN J S. A molecular caliper mechanism for determining very long-chain fatty acid length[J]. Cell, 2007, 130: 663-677. DOI:10.1016/j.cell.2007.03.013.
[26] SIBIRNY A A. Yeast peroxisomes: structure, functions and biotechnological opportunities[J]. FEMS Yeast Research, 2016, 16(4).DOI:10.1093/femsyr/fow038.
[27] LEGRAS J L, ERNY C, LE JEUNE C, et al. Activation of two different resistance mechanisms in Saccharomyces cerevisiae upon exposure to octanoic and decanoic acids[J]. Applied and Environmental Microbiology, 2010, 76: 7526-7535. DOI:10.1128/AEM.01280-10.
[28] BORRULL A, LOPEZ-MARTINEZ G, POBLET M, et al. New insights into the toxicity mechanism of octanoic and decanoic acids on Saccharomyces cerevisiae[J]. Yeast, 2015, 32: 451-460.
[29] ANDRISIC L, COLLINSON E J, TEHLIVETS O, et al.Transcriptional and antioxidative responses to endogenous polyunsaturated fatty acid accumulation in yeast[J]. Molecular and Cellular Biochemistry, 2015, 399: 27-37. DOI:10.1007/s11010-014-2229-6.
[30] JANSURIYAKUL S, SOMBOON P, RODBOON N, et al. The zinc cluster transcriptional regulator Asg1 transcriptionally coordinates oleate utilization and lipid accumulation in Saccharomyces cerevisiae[J]. Applied Microbiology and Biotechnology, 2016, 100:4549-4560.
Transcriptomic Sequence Analysis of the Effect of Three Fatty Acids with Different Chain Length on Gene Transcription in Saccharomyces cerevisiae
HAN Li, LI Lei, YANG Hourong, et al. Transcriptomic sequence analysis of the effect of three fatty acids with different chain length on gene transcription in Saccharomyces cerevisiae[J]. Food Science, 2019, 40(6): 106-112. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-20180316-221. http://www.spkx.net.cn