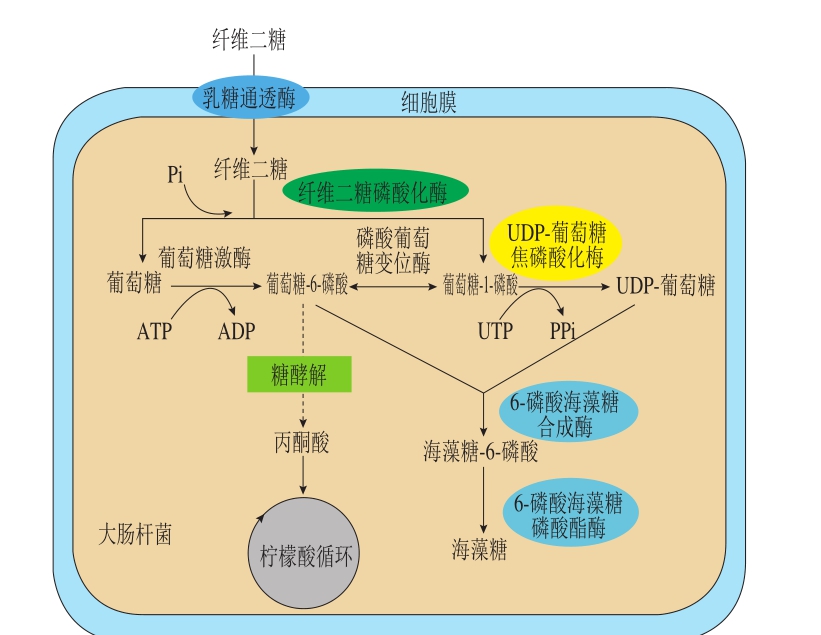
图1 重组大肠杆菌纤维二糖合成海藻糖的途径
Fig. 1 Biosynthesis pathway of trehalose from cellobiose in recombinantE. coli
木质纤维原料是地球上最丰富、最廉价的可再生资源,是制备生物燃料和生物基化学品的首选原料[1-3]。由于其结构顽固性,需要对其进行预处理和酶水解才能得到微生物可以利用的可发酵性糖[4-5]。其中,纤维二糖是纤维素酶水解纤维素时的主要产物。由于绝大多数微生物不能直接利用纤维二糖,需要外源添加β-葡萄糖苷酶进一步水解纤维二糖为葡萄糖,这进一步增加了酶制剂成本。此外,纤维二糖是纤维素酶的非竞争性抑制剂,强烈抑制纤维素酶的水解作用,极大地影响了同步糖化发酵效率[6-8]。因此,近年来,研究人员致力于开发能利用纤维二糖的微生物生产各种高附加值化学品。例如,Adachi等[9]通过将β-葡萄糖苷酶展示在谷氨酸棒状杆菌细胞表面,使得谷氨酸棒杆菌具备纤维二糖水解能力,实现了纤维二糖至L-赖氨酸的发酵生产。Muñoz-Gutiérrez等[10]使用属于V型分泌系统的自动展示系统AIDA-I,在产乙醇的大肠杆菌菌株MS04的外膜上展示来自嗜热放线菌的β-葡萄糖苷酶,以纤维二糖为底物,乙醇产率达到了理论最大值的81%。Soma等[11]通过将异丙醇合成途径导入到大肠杆菌,并在大肠杆菌外膜上展示β-葡萄糖苷酶,证实了利用大肠杆菌发酵纤维二糖生产异丙醇的可行性。
海藻糖是由两个葡萄糖分子以α,α-1,1-糖苷键结合的非还原性双糖,广泛存在于细菌、真菌和动植物体内。海藻糖不仅可以作为生物体的结构成分和能量物质,而且在氧化或干燥等极端条件下对生物体和生物大分子具有非特异性保护作用。海藻糖在食品、化妆品、农业和制药工业中有着广泛的应用前景[12-13]。例如,海藻糖可用于疫苗的稳定和器官的保存[14-15]。Martinetti等[16]建立了一种使用海藻糖冷冻保存纯的外周血干细胞的方法,与正常冷冻保存手段相比,该方法提高了解冻后的细胞存活率。Vílchez等[17]筛选了一系列微生物作为耐干燥保护剂以保护植物免受干旱的影响,研究发现这些微生物产生的海藻糖是保护植物免受干旱的重要物质。随着市场对海藻糖的需求不断增大,研究人员也在不断开发合成海藻糖的新方法。生物体内研究最多的海藻糖合成途径主要有三条:一是OtsAB途径,该途径以葡萄糖为底物,通过6-磷酸海藻糖合成酶和6-磷酸海藻糖磷酸酯酶催化形成海藻糖[18-19];二是TreYZ途径,该途径以麦芽糊精为底物,通过麦芽寡糖基海藻糖合成酶和麦芽寡糖基海藻糖水解酶催化形成海藻糖[20];三是TreS途径,该途径以麦芽糖为底物,通过海藻糖合酶催化形成海藻糖[21]。
假单胞菌是一种条件致病菌,其可以利用多种不同的策略抵抗干旱等恶劣环境,其中的一种策略就是在细胞内合成海藻糖[22-23]。Zheng Zhaojuan等[21]克隆了施氏假单胞菌(Pseudomonas stutzeri)A1501来源的treS基因,并在大肠杆菌(Escherichia coli)中重组表达,全细胞催化实验表明该重组菌株具有高效转化麦芽糖为海藻糖的能力。基因组分析表明,P. stutzeri A1501基因组上还具备海藻糖合成的OtsAB途径和TreYZ途径。本研究克隆了来自P. stutzeri A1501的otsAB基因,外源导入E. coli BL21(DE3)构建合成海藻糖的OtsAB途径。并通过过表达来自大肠杆菌的尿苷二磷酸(uridine diphosphate,UDP)-葡萄糖焦磷酸化酶基因galU来强化合成海藻糖的OtsAB途径。在此基础上,引入来自1 株天然纤维素分解细菌Saccharophagus degradans的纤维二糖磷酸化酶基因cepA,使大肠杆菌具备纤维二糖代谢能力。重组大肠杆菌以纤维二糖为底物合成海藻糖的代谢途径如图1所示。最后,本研究探讨了该重组大肠杆菌以纤维二糖为底物全细胞催化合成海藻糖的可行性,为纤维二糖来源精细化学品的生产提供了新的思路。

图1 重组大肠杆菌纤维二糖合成海藻糖的途径
Fig. 1 Biosynthesis pathway of trehalose from cellobiose in recombinantE. coli
1.1.1 菌株及质粒
表1 本研究使用的菌株和质粒
Table 1 Strains and plasmids used in this study
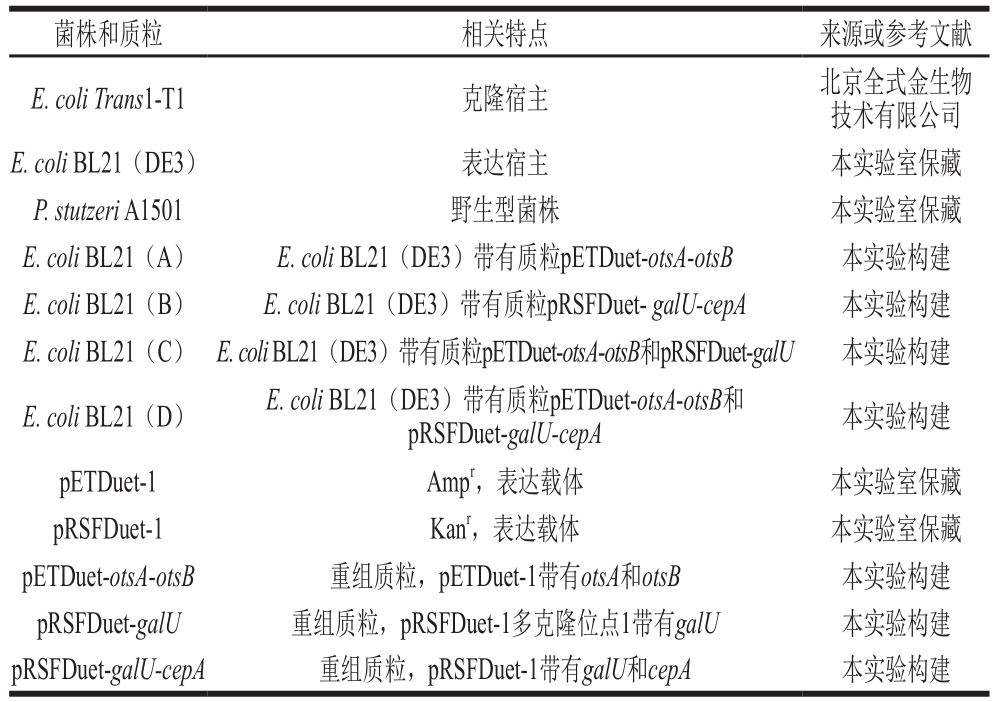
菌株和质粒 相关特点 来源或参考文献E. coli Trans1-T1 克隆宿主 北京全式金生物技术有限公司E. coli BL21(DE3) 表达宿主 本实验室保藏P. stutzeri A1501 野生型菌株 本实验室保藏E. coli BL21(A)E. coli BL21(DE3)带有质粒pETDuet-otsA-otsB 本实验构建E. coli BL21(B)E. coli BL21(DE3)带有质粒pRSFDuet- galU-cepA 本实验构建E. coli BL21(C)E. coli BL21(DE3)带有质粒pETDuet-otsA-otsB和pRSFDuet-galU 本实验构建E. coli BL21(D)E. coli BL21(DE3)带有质粒pETDuet-otsA-otsB和pRSFDuet-galU-cepA 本实验构建pETDuet-1 Ampr,表达载体 本实验室保藏pRSFDuet-1 Kanr,表达载体 本实验室保藏pETDuet-otsA-otsB 重组质粒,pETDuet-1带有otsA和otsB 本实验构建pRSFDuet-galU 重组质粒,pRSFDuet-1多克隆位点1带有galU 本实验构建pRSFDuet-galU-cepA 重组质粒,pRSFDuet-1带有galU和cepA 本实验构建
本研究所用菌株和质粒见表1,聚合酶链式反应(polymerase chain reaction,PCR)扩增所用引物序列见表2。其中纤维二糖磷酸化酶基因cepA(GenBank NC_007912,范围:1710240~1712675)按照大肠杆菌密码子偏好性优化后,由上海捷瑞生物工程有限公司合成。
表2 本研究使用的PCR扩增引物
Table 2 Sequences of PCR primers used in this study

注:加粗的字体为限制性内切酶酶切位点。
引物 碱基方向(5′-3′)otsA-F GGATCCGATGAGCCGTTTAGTAGTGGT(BamHI)otsA-R AAGCTTTCAGTCCGGCTCGACGAGTT(HindIII)otsB-F GGGCCTTCCCATATGATGAATGATCAAAACGACCAA(NdeI)otsB-R TTATCCCTCGAGCTAGTCGGGTTTTATTCTGG(XhoI)galU-F AAACTTGGATCCCATGGCTGCCATTAATACGAA(BamHI)galU-R TTGGCCCAAGCTTTTACTTCTTAATGCCCATCT(HindIII)cepA-F GTTCCAGATCTCATGAAATTTGGCCATTTTGATG(BglII)cepA-R ATTAACTCGAGTTAGCCCAGGGTCACTTCAACAT(XhoI)
1.1.2 酶与试剂
Pfu DNA聚合酶、限制性内切酶、T4 DNA连接酶TaKaRa(大连)公司;酵母提取物、胰蛋白胨 德国Sigma公司;异丙基-β-D-硫代半乳糖苷(isopropyl-β-D-thiogalactopyranoside,IPTG)、氨苄青霉素钠、硫酸卡那霉素、井冈霉素 生工生物工程(上海)股份有限公司;基因组提取试剂盒 北京全式金生物技术有限公司;质粒小量快速提取试剂盒和琼脂糖凝胶DNA回收试剂盒 上海捷瑞生物工程公司;其余试剂均为国产分析纯;引物由上海捷瑞生物工程公司合成。
MasterCycler Gradient 5331梯度PCR仪、5415R高速冷冻离心机 德国Eppebdorf公司;DYY-8B核酸电泳仪、GIS system凝胶成像系统 上海天能科技有限公司;752S型紫外-可见分光光度计 上海棱光技术有限公司;ICS·3000型离子色谱仪 美国Dionex公司;MSC1.2无菌操作台 美国Thermo公司。
1.3.1 分子克隆
DNA酶切、连接、感受态细胞制备和转化、基因组提取等基本基因操作技术参照文献[24]及供应商提供的操作手册进行。
质粒pETDuet-otsA-otsB构建:参照GenBank数据库中已公布的P. stutzeri A1501的otsA基因序列(GenBank NC_009434,范围:3495671~3497086)和otsB基因序列(GenBank NC_009434,范围:3497103~3497879),使用引物otsA-F、otsA-R扩增otsA基因插入质粒pETDuet-1的BamHI、HindIII位点,获得重组质粒pETDuet-otsA。然后再使用引物otsB-F、otsB-R扩增otsB基因插入质粒pETDuet-otsA的NdeI、XhoI位点,获得重组质粒pETDuetotsA-otsB,测序验证。
质粒pRSFDuet-galU-cepA的构建:参照GenBank数据库中已公布的E. coli BL21的galU基因序列(GenBank NC_012892,范围:1279243~1280151),使用引物galU-F、galU-R扩增galU基因插入质粒pRSFDuet-1的BamHI、HindIII位点,获得重组质粒pRSFDuet-galU。然后参照GenBank数据库中已公布的S. degradans 2-40的cepA基因序列(GenBank NC_007912,范围:1710240~1712675),对cepA基因序列进行密码子优化,送去上海捷瑞生物工程有限公司合成。使用引物cepA-F、cepA-R扩增合成的cepA基因插入质粒pRSFDuetgalU的BglII、XhoI位点,获得重组质粒pRSFDuet-galU-cepA,测序验证。
1.3.2 培养基与培养条件
LB培养基:胰蛋白胨10.0 g/L,酵母粉5.0 g/L,NaCl 10.0 g/L,pH 7.0。
磷酸盐缓冲液(phosphate buffer saline,PBS):Na2HPO4·12 H2O 19.100 8 g/L,KH2PO4 1.815 6 g/L。
大肠杆菌培养时根据需要加入终质量浓度为100 mg/L的氨苄青霉素钠或40 mg/L的硫酸卡那霉素。
1.3.3 重组大肠杆菌全细胞催化合成海藻糖
1.3.3.1 菌种活化
将保存于-80 ℃的菌种接入装有5 mL LB的摇管中,接种量1%,并根据需要加入终质量浓度100 mg/L氨苄青霉素钠,40 mg/L硫酸卡那霉素,放置于37 ℃、200 r/min条件下过夜培养。
1.3.3.2 扩培和诱导
将活化好的菌种转入装有1/5装液量的500 mL的三角摇瓶中,接种量1%,并根据需要加入终质量浓度100 mg/L氨苄青霉素钠、40 mg/L硫酸卡那霉素,放置于37 ℃、200 r/min条件下培养,OD600 nm达到0.6~0.8时加入终浓度1 mmol/L的IPTG,转移到30 ℃、200 r/min条件下继续培养8 h。8 000 r/min离心收集诱导好的菌体,用PBS洗涤菌体2 遍,重悬于PBS中备用。
1.3.3.3 全细胞催化
全细胞催化在150 mL的锥形瓶中进行,装有10 mL催化反应混合液,混合液中的菌体OD600 nm为20,葡萄糖或纤维二糖质量浓度为20 g/L,并根据需要加入终浓度0.05 mmol/L井冈霉素。反应在30 ℃、200 r/min的条件下进行,定时取样测定底物和产物浓度。
1.3.4 指标检测
菌体密度测定采用比浊法。将培养液用水稀释适当的倍数,使OD600 nm值在0.6~1.0之间,进行浊度测定。测定条件:采用紫外-可见分光光度计,波长600 nm。
葡萄糖、纤维二糖、海藻糖质量浓度测用采用离子色谱法[25]。取1 mL反应混合液,室温下12 000 r/min离心2 min,取上清液,用孔径为0.22 μm的无菌滤膜过滤。分析条件:采用离子色谱仪,分析柱为CarboPacTM10(250 mm×2 mm);检测方式:四电位脉冲安培检测。以超纯水、200 mmol/L NaOH(A)和500 mmol/L NaAc(B)为淋洗液进行洗脱。0~7.00 min,采用75%超纯水和25% A相混合液淋洗,7.01~10 min变为100% A相,然后A相线性减少,B相线性增加,至15.00 min时,变为90% A相和10% B相混合液;15.01~30 min恢复为75%超纯水和25% A相冲洗系统。进样量为10 μL,流速为0.3 mL/min,柱温为30 ℃。与标准曲线对照计算各物质的含量。数据测定后取平均值,本研究中误差线代表3 个平行实验的标准偏差。
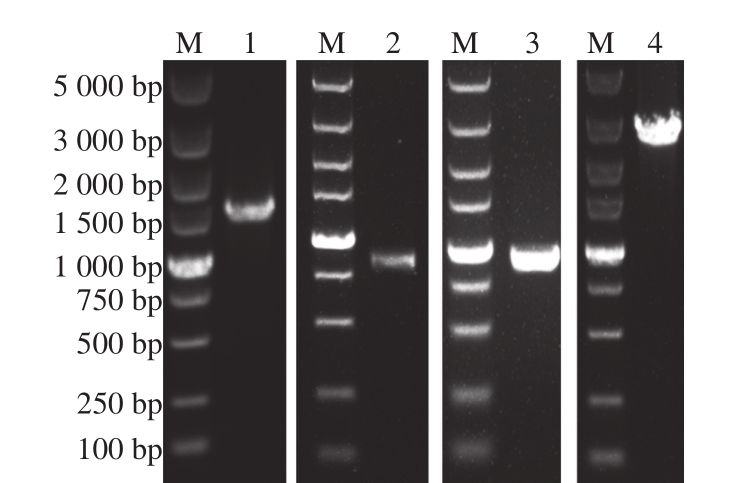
图2otsA、otsB、galU和cepA基因PCR扩增产物电泳图
Fig. 2 Electrophoretograms of PCR-amplifiedotsA,otsB,galU andcepA
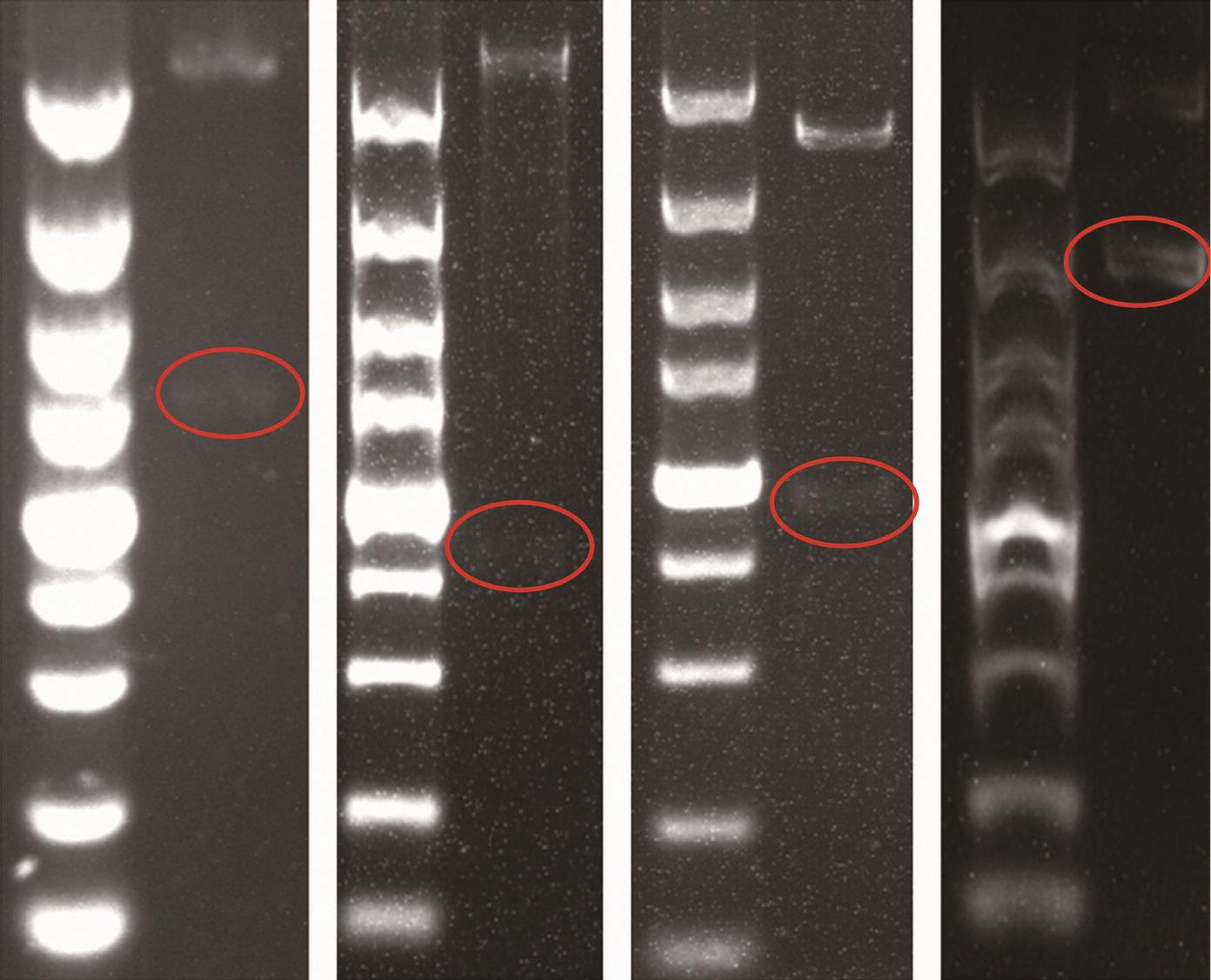
图3 重组表达质粒pETDuet-otsA-otsB、 pRSFDuet-galU和pRSFDuet-galU-cepA双酶切验证
Fig. 3 Double restriction enzyme digestion analysis of recombinant plasmid pETDuet-otsA-otsB, pRSFDuet-galU and pRSFDuet-galU-cepA
M. Marker DL5000;泳道1.自pETDuet-otsA-otsB双酶切otsA;泳道2.自pETDuet-otsA-otsB双酶切otsB;泳道3.自pRSFDuetgalU双酶切galU;泳道4.自pRSFDuet-galU-cepA双酶切cepA。
提取P. stutzeri A1501的基因组,以其为模板分别PCR扩增得到otsA和otsB基因,按照1.3.1节所述实验方法将其连接至pETDuet-1表达载体,获得重组表达质粒pETDuetotsA-otsB并送至安徽通用生物有限公司测序。提取E. coli BL21的基因组,以其为模板PCR扩增得到galU基因,按照1.3.1节所述实验方法将其连接至pRSFDuet-1表达载体,获得重组表达质粒pRSFDuet-galU并送至安徽通用生物有限公司测序。cepA基因由上海捷瑞生物工程有限公司按照大肠杆菌密码子优化后序列合成,之后以其合成基因为模板进行PCR扩增,按照1.3.1节所述实验方法将其连接至pRSFDuet-galU,获得重组表达质粒pRSFDuetgalU-cepA并送至安徽通用生物有限公司测序。otsA、otsB、galU和cepA基因的PCR扩增如图2所示。琼脂糖凝胶电泳和测序结果均表明,otsA基因长度为1 416 bp,otsB基因长度为777 bp,galU基因长度为909 bp,cepA基因长度为2 436 bp,其中otsA、otsB和galU基因序列与NCBI公布序列大小相符,序列一致性为100%。cepA基因与密码子优化后的序列完全一致。重组表达质粒pETDuet-otsA-otsB、pRSFDuet-galU和pRSFDuet-galU-cepA双酶切验证如图3所示,otsA、otsB、galU和cepA基因均已成功连接。
虽然有报道表明大肠杆菌具备海藻糖合成的OtsAB途径[26],但可能由于该途径太弱,无法检测到海藻糖产量。因此本研究克隆了来自P. stutzeri A1501的otsAB基因,在E. coli BL21(DE3)内重构了一条合成海藻糖的途径。OtsAB途径合成海藻糖的前体物质是葡萄糖-6-磷酸和UDP-葡萄糖(图1),由于菌体细胞自身合成的UDP-葡萄糖在细胞内积累水平较低,本实验通过过表达大肠杆菌内参与UDP-葡萄糖合成的关键酶UDP-葡萄糖焦磷酸化酶来提高重组菌株细胞内的UDP-葡萄糖水平。以葡萄糖为底物全细胞催化合成海藻糖,比较出发菌株E. coli BL21(DE3)和重组菌株E. coli BL21(A),E. coli BL21(C)利用葡萄糖合成海藻糖的能力,结果如图4所示。
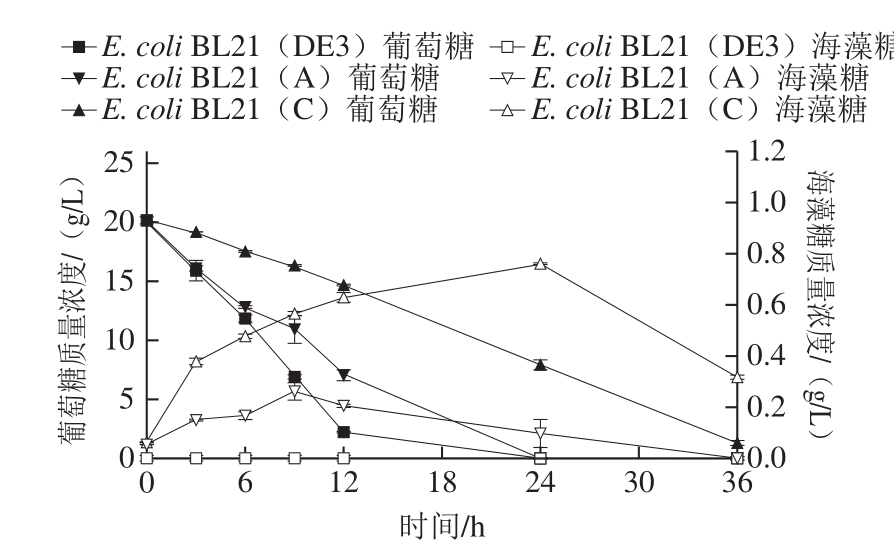
图4 对照菌与重组菌全细胞催化合成海藻糖的过程分析
Fig. 4 Time course of trehalose synthesis catalyzed by whole control and recombinant cells
从图4可以看出,出发菌株E. coli BL21(DE3)不能将葡萄糖转化为海藻糖。重组菌株E. coli BL21(A)可以将葡萄糖转化为海藻糖,海藻糖产量在8 h达到最高值0.25 g/L左右,这说明外源otsA/otsB基因可在大肠杆菌中成功表达并发挥功能。与E. coli BL21(A)相比,重组菌株E. coli BL21(C)转化葡萄糖为海藻糖的能力大幅提高,海藻糖的产量在24 h达到0.75 g/L左右。过表达来自大肠杆菌的UDP-葡萄糖焦磷酸化酶后,虽然葡萄糖利用速率减慢,但是增加了海藻糖合成前体UDP-葡萄糖的含量,海藻糖的产量提高了3 倍左右。
大肠杆菌细胞内含有降解海藻糖的酶,即海藻糖酶,可以分解海藻糖为两分子葡萄糖[27],从图4可以看出,海藻糖产量在达到一定程度后开始下降,这也进一步验证了海藻糖酶的存在。据报道[28],井冈霉素可以降低海藻糖酶的活性,抑制海藻糖在菌体内的降解。为进一步提高海藻糖的产量,在添加井冈霉素的条件下,以葡萄糖为底物全细胞催化合成海藻糖,比较重组菌株E. coli BL21(A),E. coli BL21(C)合成海藻糖的能力,结果如图5所示。
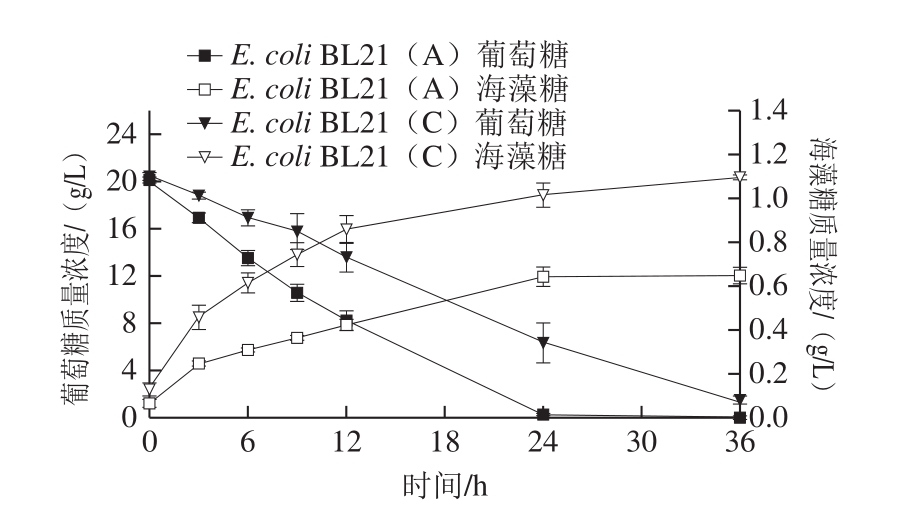
图5 井冈霉素对重组菌全细胞催化合成海藻糖的影响
Fig. 5 Effect of validamycin A on the synthesis of trehalose
由图5可以看出,随着底物葡萄糖的不断消耗,海藻糖也在不断积累,E. coli BL21(A)转化葡萄糖生成海藻糖的产量在36 h达到0.68 g/L左右,E. coli BL21(C)转化葡萄糖生成海藻糖的产量在36 h达到1.1 g/L左右。同时,由图4和图5对比可以看出,加入井冈霉素后,海藻糖的产量得到明显提高,并且维持在一定的水平不再下降。
据Sekar等[29]报道,S. degradans 2-40是一株天然纤维素分解细菌,可依靠cepA基因将纤维二糖磷酸解为葡萄糖和葡萄糖-1-磷酸。因此,在上述实验的基础上,在重组菌株E. coli BL21(C)胞内进一步过表达cepA基因,以期实现纤维二糖至海藻糖的合成通路。在添加井冈霉素的条件下,以纤维二糖为底物全细胞催化合成海藻糖,比较重组菌株E. coli BL21(C)和重组菌株E. coli BL21(D)利用纤维二糖合成海藻糖的能力,结果如图6所示。

图6 以纤维二糖为底物合成海藻糖的过程曲线
Fig. 6 Time course of trehalose synthesis from cellobiose
由图6可以看出,重组菌株E. coli BL21(C)不能将纤维二糖转化为海藻糖,重组菌株E. coli BL21(D)可以将纤维二糖转化为海藻糖,海藻糖的产量在48 h达到最高值1.3 g/L左右,这是以纤维二糖为底物合成海藻糖的首次报道。但是,由于大肠杆菌糖酵解途径的存在,使得大量葡萄糖-6-磷酸进入糖酵解途径被消耗,这导致海藻糖合成的前体物质大为减少(图1),因此下一步应该采取合适的代谢工程策略,在不影响菌株存活的前提下,增加纤维二糖至海藻糖的代谢流量。
目前,海藻糖的生产方法主要是酶合成法和微生物发酵法。依赖于麦芽寡糖基海藻糖合成酶和麦芽寡糖基海藻糖水解酶的酶法合成途径是最早建立的规模化海藻糖生产方法,但该方法生产工艺复杂、生产成本高,无法满足市场对海藻糖的大量需求[30]。微生物发酵法主要是以筛选的野生型菌株、过表达6-磷酸海藻糖合成酶和6-磷酸海藻糖磷酸酯酶或者过表达海藻糖合酶的基因工程菌株作为生物催化剂,以生长体系或者全细胞催化体系实现海藻糖的合成[19]。其中海藻糖合酶的优点是只需要一步反应即可以实现麦芽糖至海藻糖的转化,缺点是海藻糖合酶具有一定的麦芽糖水解活性,该反应常伴有副产物葡萄糖的产生[21]。依赖于6-磷酸海藻糖合成酶和6-磷酸海藻糖磷酸酯酶的OtsAB途径在自然界分布最为广泛,是研究最多的一种胞内合成海藻糖的方式。微生物利用OtsAB途径合成海藻糖的底物为葡萄糖,近年来,研究者开发了多种不同的底物通过OtsAB途径实现了海藻糖的合成。例如,Li He等[28]通过在E. coli MG1655过表达来自E. coli DH5α的OtsAB途径,以甘油为底物实现了海藻糖的生产。Habe等[31]通过利用筛选的Burkholderia stabilis LA20W为出发菌株,以乙酰丙酸为底物合成了海藻糖。Kar等[32]利用专性嗜盐菌Actinopolyspora halophile MTCC 263为出发菌株,以酸乳清为底物合成了海藻糖。本研究构建了以纤维二糖为底物合成海藻糖的途径,这为以木质纤维原料为底物合成海藻糖提供了研究基础。但是,和其他利用OtsAB途径合成海藻糖的菌株一样,海藻糖的产量不高,后续工作中需要结合代谢工程手段敲除支路途径以提高海藻糖的产量。
本研究克隆了来自P. stutzeri A1501的otsA/otsB基因,成功构建用于海藻糖生产的重组大肠杆菌。通过增加海藻糖合成前体物质的含量和抑制海藻糖的降解,进一步提高海藻糖的产量。UDP-葡萄糖是合成海藻糖的前体物质之一,它是提高海藻糖产量的限制因素之一,本实验通过提高胞内UDP-葡萄糖水平,使海藻糖的产量提高了3 倍。同时,菌体内还有降解海藻糖的酶系,通过添加一定浓度的井冈霉素抑制海藻糖酶的活性,进一步使提高海藻糖产量。最后,为实现菌株对纤维二糖的利用,在前期改造的菌株中进一步过表达了纤维二糖磷酸化酶基因,使重组大肠杆菌具有利用纤维二糖产海藻糖的能力,最高产量为1.3 g/L。
[1] ISIKGOR F H, BECER C R. Lignocellulosic biomass: a sustainable platform for the production of bio-based chemicals and polymers[J].Polymer Chemistry, 2015, 6(25): 4497-4559. DOI:10.1039/C5PY00263J.
[2] KAWAGUCHI H, HASUNUMA T, OGINO C, et al. Bioprocessing of bio-based chemicals produced from lignocellulosic feedstocks[J].Current Opinion in Biotechnology, 2016, 42: 30-39. DOI:10.1016/j.copbio.2016.02.031.
[3] WHEELDON I, CHRISTOPHER P, BLANCH H. Integration of heterogeneous and biochemical catalysis for production of fuels and chemicals from biomass[J]. Current Opinion in Biotechnology, 2017,45: 127-135. DOI:10.1016/j.copbio.2017.02.019.
[4] ALVIRA P, TOMÁS-PEJÓ E, BALLESTEROS M J, et al. Pretreatment technologies for an efficient bioethanol production process based on enzymatic hydrolysis: a review[J]. Bioresource Technology, 2010,101(13): 4851-4861. DOI:10.1016/j.biortech.2009.11.093.
[5] KUMAR P, BARRETT D M, DELWICHE M J, et al. Methods for pretreatment of lignocellulosic biomass for efficient hydrolysis and biofuel production[J]. Industrial & Engineering Chemistry Research,2009, 48(8): 3713-3729. DOI:10.1021/ie801542g.
[6] BEZERRA R M F, DIAS A A. Enzymatic kinetic of cellulose hydrolysis[J]. Applied Biochemistry and Biotechnology, 2005, 126(1):49-59. DOI:10.1007/s12010-005-0005-5.
[7] GRUNO M, VÄLJAMÄE P, PETTERSSON G, et al. Inhibition of the Trichoderma reesei cellulases by cellobiose is strongly dependent on the nature of the substrate[J]. Biotechnology and Bioengineering,2004, 86(5): 503-511. DOI:10.1002/bit.10838.
[8] ZHAO Y, WU B, YAN B, et al. Mechanism of cellobiose inhibition in cellulose hydrolysis by cellobiohydrolase[J]. Science in China Series C:Life Sciences, 2004, 47(1): 18-24. DOI:10.1360/02yc0163.
[9] ADACHI N, TAKAHASHI C, ONO-MUROTA N, et al. Direct L-lysine production from cellobiose by Corynebacterium glutamicum displaying β-glucosidase on its cell surface[J]. Applied Microbiology and Biotechnology, 2013, 97(16): 7165-7172. DOI:10.1007/s00253-013-5009-4.
[10] MUÑOZ-GUTIÉRREZ I, OROPEZA R, GOSSET G, et al. Cell surface display of a β-glucosidase employing the type V secretion system on ethanologenic Escherichia coli for the fermentation of cellobiose to ethanol[J]. Journal of Industrial Microbiology &Biotechnology, 2012, 39(8): 1141-1152. DOI:10.1007/s10295-012-1122-0.
[11] SOMA Y, INOKUMA K, TANAKA T, et al. Direct isopropanol production from cellobiose by engineered Escherichia coli using a synthetic pathway and a cell surface display system[J]. Journal of Bioscience and Bioengineering, 2012, 114(1): 80-85. DOI:10.1016/j.jbiosc.2012.02.019.
[12] OHTAKE S, WANG Y J. Trehalose: current use and future applications[J]. Journal of Pharmaceutical Sciences, 2011, 100(6):2020-2053. DOI:10.1002/jps.22458.
[13] SCHIRALDI C, DI LERNIA I, DE ROSA M. Trehalose production:exploiting novel approaches[J]. Trends in Biotechnology, 2002,20(10): 420-425. DOI:10.1016/S0167-7799(02)02041-3.
[14] KIM Y C, QUAN F S, COMPANS R W, et al. Formulation and coating of microneedles with inactivated inf l uenza virus to improve vaccine stability and immunogenicity[J]. Journal of Controlled Release, 2010,142(2): 187-195. DOI:10.1016/j.jconrel.2009.10.013.
[15] PATIST A, ZOERB H. Preservation mechanisms of trehalose in food and biosystems[J]. Colloids and Surfaces B: Biointerfaces, 2005,40(2): 107-113. DOI:10.1016/j.colsurfb.2004.05.003.
[16] MARTINETTI D, COLAROSSI C, BUCCHERI S, et al. Effect of trehalose on cryopreservation of pure peripheral blood stem cells[J].Biomedical Reports, 2017, 6(3): 314-318. DOI:10.3892/br.2017.859.
[17] VÍLCHEZ J I, GARCÍA-FONTANA C, ROMÁN-NARANJO D, et al. Plant drought tolerance enhancement by trehalose production of desiccation-tolerant microorganisms[J]. Frontiers in Microbiology,2016, 7: 1577. DOI:10.3389/fmicb.2016.01577.
[18] PADILLA L, KRÄMER R, STEPHANOPOULOS G, et al.Overproduction of trehalose: heterologous expression of Escherichia coli trehalose-6-phosphate synthase and trehalose-6-phosphate phosphatase in Corynebacterium glutamicum[J]. Applied and Environmental Microbiology, 2004, 70(1): 370-376. DOI:10.1128/AEM.70.1.370-376.2004.
[19] WU Y, SUN X, LIN Y, et al. Establishing a synergetic carbon utilization mechanism for non-catabolic use of glucose in microbial synthesis of trehalose[J]. Metabolic Engineering, 2017, 39: 1-8.DOI:10.1016/j.ymben.2016.11.001.
[20] CARPINELLI J, KRÄMER R, AGOSIN E. Metabolic engineering of Corynebacterium glutamicum for trehalose overproduction: role of the TreYZ trehalose biosynthetic pathway[J]. Applied and Environmental Microbiology, 2006, 72(3): 1949-1955. DOI:10.1128/AEM.72.3.1949-1955.2006.
[21] ZHENG Z J, XU Y, SUN Y, et al. Biocatalytic production of trehalose from maltose by using whole cells of permeabilized recombinant Escherichia coli[J]. PLoS ONE, 2015, 10(10): e0140477.DOI:10.1371/journal.pone.0140477.
[22] FREEMAN B C, CHEN C, BEATTIE G A. Identification of the trehalose biosynthetic loci of Pseudomonas syringae and their contribution to fitness in the phyllosphere[J]. Environmental Microbiology, 2010, 12(6): 1486-1497. DOI:10.1111/j.1462-2920.2010.02171.x.
[23] DJONOVIĆ S, URBACH J M, DRENKARD E, et al. Trehalose biosynthesis promotes Pseudomonas aeruginosa pathogenicity in plants[J]. PLoS Pathogens, 2013, 9(3): e1003217. DOI:10.1371/journal.ppat.1003217.
[24] SAMBROOK J, RUSSELL D W, RUSSELL D, et al. Molecular cloning: a laboratory manual[M]. UK: Coldspring-Harbour Laboratory Press, 2001.
[25] 徐颖. 海藻糖合酶基因的克隆表达及海藻糖的制备[D]. 南京: 南京林业大学, 2015.
[26] STROM A R, KAASEN I. Trehalose metabolism in Escherichia coli:stress protection and stress regulation of gene expression[J]. Molecular Microbiology, 1993, 8(2): 205-210. DOI:10.1111/j.1365-2958.1993.tb01564.x.
[27] GIBSON R P, GLOSTER T M, ROBERTS S, et al. Molecular basis for trehalase inhibition revealed by the structure of trehalase in complex with potent inhibitors[J]. Angewandte Chemie International Edition,2007, 46(22): 4115-4119. DOI:10.1002/anie.200604825.
[28] LI H, SU H, KIM S B, et al. Enhanced production of trehalose in Escherichia coli by homologous expression of otsBA in the presence of the trehalase inhibitor, validamycin A, at high osmolarity[J]. Journal of Bioscience and Bioengineering, 2012, 113(2): 224-232. DOI:10.1016/j.jbiosc.2011.09.018.
[29] SEKAR R, SHIN H D, CHEN R. Engineering Escherichia coli cells for cellobiose assimilation through a phosphorolytic mechanism[J].Applied and Environmental Microbiology, 2012, 78(5): 1611-1614.DOI:10.1128/AEM.06693-11.
[30] 吴秀丽, 岳明, 丁宏标. 海藻糖合酶的研究进展[J]. 微生物学通报,2009, 36(7): 1067-1072. DOI:10.13344/j.microbiol.china.2009.07.009.
[31] HABE H, SATO S, MORITA T, et al. Bacterial production of shortchain organic acids and trehalose from levulinic acid: a potential cellulose-derived building block as a feedstock for microbial production[J]. Bioresource Technology, 2015, 177: 381-386.DOI:10.1016/j.biortech.2014.11.048.
[32] KAR J R, HALLSWORTH J E, SINGHAL R S. Fermentative production of glycine betaine and trehalose from acid whey using Actinopolyspora halophila (MTCC 263)[J]. Environmental Technology &Innovation, 2015, 3: 68-76. DOI:10.1016/j.eti.2015.02.001.
Biosynthesis of Trehalose from Cellobiose by Recombinant Escherichia coli
LI Xin, ZHENG Zhaojuan, YUE Taiwen, et al. Biosynthesis of trehalose from cellobiose by recombinant Escherichia coli[J]. Food Science, 2019, 40(6): 180-186. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-20180411-132.http://www.spkx.net.cn