海参鉴别技术研究进展
胡冉冉1,2,邢冉冉1,张九凯1,葛毅强2,3,陈 颖1,*
(1.中国检验检疫科学研究院,北京 100123;2.中国农业大学食品科学与营养工程学院,北京 100083;3.中国农村技术开发中心,北京 100045)
摘 要:海参营养价值高且味道鲜美,深受消费者的喜爱。目前市场上海参种类繁多,不同品种间的海参品质和价格存在差异。受经济利益驱动,高值海参产品近年来在贸易过程中存在标签错误、掺杂掺假等现象,极大地损害了消费者的权益。本文基于海参的掺假现状,综述了海参鉴别技术的研究进展,主要包括形态学鉴定、理化技术和分子生物学鉴别技术,并对海参的鉴别技术研究进行了展望。
关键词:海参;鉴别技术;形态学鉴定;理化技术;分子生物学技术
海参是一种高蛋白、低脂肪的名贵海产品,其营养价值高且味道鲜美,富含蛋白质、氨基酸、必需脂肪酸以及微量元素等50多种营养成分,同时含有多糖、皂苷、胶原蛋白等重要的生物活性物质[1],在抗癌[2]、抗肿瘤[3]、免疫调节[4]及抗凝血[5]等方面可发挥显著功效。目前全球已知的海参共有1 500多种,但具有较高经济和食用价值的海参约有60 种。全球捕捞的海参中有90%左右供给亚洲地区以满足其市场需求,中国是海参的最大消费市场[6]。在贸易中海参多以干海参、水发海参和海参胶囊、海参口服液、海参罐头等海参制品的形式流通,鲜活产品和冻品仅占小部分[7]。经过干制、切片、制粉等处理过的海参及制品使得消费者、经销商和监管部门难以从形态上鉴别其种类和品质。加之市场上海参种类繁多,产地不同、营养成分及其含量各异等因素导致其价格相差可高达10 倍。一些不规范厂家为了谋取高额经济利益,在海参贸易中掺假造假时有发生,如以人造海参冒充真海参,通过贴假标签用低值海参冒充高值海参干货、产地造假冒充地理标志产品等[8]。《中华人民共和国产品质量法》第五十条规定产品不得掺杂、掺假,不得以假充真、以次充好,不得以不合格产品冒充合格产品。GB 7718—2011《食品安全国家标准 预包装食品标签通则》以及GB 13432—2013《食品安全国家标准 预包装特殊膳食用食品标签》也都规定了食品标签的真实性,不允许利用食品标签名称混淆食品的真实属性欺骗消费者。鉴于此,上述海参贸易中存在的经济利益驱动的食品掺假行为(economically motivated adulteration,EMA)不仅直接损害消费者的经济利益,而且侵犯了地理标志产品生产商的权益,干扰了海参市场秩序。因此加强海参物种和产地的鉴别对打击海参掺假造假行为十分必要。
随着生物技术的不断发展,目前国内外学者已经利用形态学、光谱和质谱以及分子生物学等技术从海参形态、所含的化学物质、核酸差异等方面鉴别海参。本文对近年来常用的海参鉴别方法进行综述,介绍各方法的特点和应用情况,以期为进一步建立海参鉴别技术提供科学依据。
1 食用海参的分类及分布
目前世界海参渔业中已知的60 种食用海参分属于海参纲的3 个目、4 个科、14 个属(表1)[9]。这些食用海参主要分布于70多个国家的部分极地地区、温带和整个热带海域地区[10]。其中,热带地区海参资源丰富、种类多、数量大,约占世界海参总产量的86%,多分布在太平洋和印度洋的热带区;温带海域海参资源呈单种性,集中分布在太平洋东西两岸,主要以刺参科为主。在我国北方的温寒带海域仅有一种仿刺参可供食用,南方海域产有约20 种食用海参[11]。根据种类、外观、颜色、体壁厚度、营养价值及市场趋势、需求,一般将商品海参分为低值、中值或高值3 个等级。高值海参有仿刺参、黑乳海参、黄乳海参、糙海参等,中值海参有乌皱辐肛参、棘辐肛参、梅花参等,而玉足海参、绿刺参、花刺参等商业价值较低。
表1 食用海参的生物学分类
Table 1 Biological classification of edible sea cucumber

目科属主要的种辐肛参属(Actinopyga) 白底辐肛参(Actinopyga mauritiana)乌皱辐肛参(Actinopyga miliaris)等白尼参属(Bohadschia) 蛇目白尼参(Bohadschia argus)图纹白尼参(Bohadschia marmorata)等海参科(Holothuriidae) 黑海参(Holothuria atra)黄乳海参(Holothuria fuscogliva)玉足海参(Holothuria leucospilota)墨西哥海参(Holothuria mexicana)糙海参(Holothuria scabra)等皮氏海参属(Pearsonothuria) 格皮氏海参(Pearsonothuria gareffei)海参属(Holothuria)楯手目(Aspidochirotida) 仿刺参属(Apostichopus)刺参科(Stichopodidae)绿刺参(Stichopus chloronotus)花刺参(Stichopus variegatus)糙刺参(Stichopus horrens)等刺参属(Stichopus) 仿刺参(Apostichopus japonicus)具疣拟刺参(Apostichopus parvimensis)梅花参属(Thelenata) 梅花参(Thelenata ananas)等暗刺参属(Isostichopus) 四边刺参(Isostichopus badionotus)巨刺参(Isostichopus fuscus)拟刺参属(Parastichopus) 加州拟刺参(Parastichopus californicus)类刺参属(Astichopus) 毛刺参(Astichopus multifidus)澳洲刺参属(Australostichopus) 褐斑刺参(Australostichopus mollis)瓜参属(Cucumaria) 大西洋瓜参(Cucumaria frondosa)圆瓜参属(Athyonidium) 智利瓜参(Athyonidium chilensis)芋参目(Molpadiida) 尻参科(Caudinidae) 海地瓜属(Acaudina) 海地瓜(Acaudina molpadioides)指手目(Dactylochirotida)瓜参科(Cucumariidae)
2 海参鉴别技术
2.1 形态学鉴定
形态学鉴定主要是通过显微镜观察海参骨片形状、数量的差异实现对海参物种的分类和鉴定。骨片是位于海参真皮表层的内骨骼。随着海参幼体的成长,海参骨片形态和数量发生变化,直到海参成体后其骨片种类和形态非常稳定。廖玉麟[12]详细描述了我国134 种海参骨片的形态学特征,为后续我国海参骨片的形态学鉴定奠定了基础。一项对我国西沙群岛6 种热带海参背脊中部和腹部骨片超显微结构的研究表明:同一属的海参骨片种类有一定共性,而不同属的海参骨片种类有所差异,根据骨片的细微结构可在种水平上鉴别这8 种海参[13]。在此研究中,使用放大倍数更高的扫描电子显微镜观察,还在6 种海参中发现了新的海参骨片,丰富了海参骨片的形态学资料。Kamarudin等[14]利用扫描电子显微镜观察海参的胃肠道和呼吸树的骨片形状,根据呼吸树骨片中是否观察到Y形棒状骨片这一特征可区分马来西亚的两种海参Stichopus horrens和Holothuria leucospilota。随后其又通过腹侧角质层的骨片形状对Holothuria scabra、S.horrens和Stichopus ocellatus 3 种海参进行了有效地区分[15]。但此研究在S. horrens和S. ocellatus中没有观察到之前报道过的骨片形状,可能是由于这两种海参受到环境的影响,形态发生了变化,导致骨片形状随之改变,这也说明海参生长环境对骨片形态学鉴定结果的准确性有一定的影响。文菁等[16]通过对新鲜仿刺参、市售干仿刺参和冷冻仿刺参产品的背脊部骨片观察,发现鲜仿刺参和干仿刺参的骨片形态和结构基本相似,而与冷冻仿刺参的骨片存在明显差异,初步判定冷冻仿刺参为假冒产品,将形态学鉴定结果结合DNA测序的鉴别结果,最终确定此冷冻仿刺参为假冒仿刺参。
上述研究均表明利用形态学鉴定方法可以实现对完整个体较为准确的鉴定。但当海参个体不完整或经加工处理后用于鉴别的形态特征消失,形态学鉴定方法就受到了限制[17]。此外,海参的形态学鉴定方法仅限于有丰富鉴别经验的专业人员,因此应用形态学方法鉴定海参难以普及,不易标准化。
2.2 理化鉴定方法
随着现代仪器分析技术的发展,理化鉴定方法操作越来越简单,逐渐应用到了水产品的真伪鉴别中。目前以光谱、色谱和质谱为主结合化学计量学法的理化鉴定技术已经应用于海参的种类鉴定和产地溯源中。特别是在鉴别不同地理区域分布的物种时,理化鉴定技术被认为是行之有效的方法。
2.2.1 光谱技术
2.2.1.1 红外光谱
红外光谱是根据红外光谱图中振动峰的位置、数目、形状和强度,结合化学计量学方法建立数学模型,可以实现对样品的定性和定量分析的技术。其中,中红外(mid-infrared,MIR)区和近红外(near infrared,NIR)区是红外活性振动和旋转吸收最强的区域,常应用于食品化合物的检测[18]。NIR信号主要由复杂的倍频峰和短波组合构成,而MIR信号来源于简单的分子振动能级跃迁,因此MIR比NIR呈现出更清晰的光谱峰值,具有更强的鉴别能力。但NIR相比MIR仪器成本较低,受空气湿度的影响小,且方便携带,更适合现场分析。
在1 700~600 cm-1的MIR区,Wu Zhongchen等[19]采用漫反射中红外傅里叶变换光谱技术(diffuse reflectance infrared Fourier transform spectroscopy,DRIFTS)对来自荣成、威海、大连和烟台4 个不同地理区域的96 个干刺参样品进行鉴别,结合主成分分析法(principal component analysis,PCA)建立的二维散点图以及簇类独立软模式识别(soft independent modeling of class analogy,SIMCA),成功区分出了4 个不同产地分布的干刺参。随后,陶琳等[20]对同样来自荣成、威海、大连和烟台4 个产地的96 个干刺参样品进行NIR光谱特征分析,主成分聚类建立的聚类三维图显示4 个产地的海参分别聚在4 个相对独立的区域,由此可成功区分4 个产地的海参。上述研究表明MIR和NIR均可为打击产地造假行为提供有效的技术手段。此外,NIR还应用于海参化学物质的定量分析中,在海参品质的快速检测方面发挥作用。邹小波等[21]利用NIR对大连、福建、连云港和山东4 个产地的43 个海参进行产地鉴别时,建立的三维PCA图显示4 个产地的海参样品存在交叉,无法准确区分4 个产地的海参,进一步采用最小二乘支持向量机(least squares support vector machine,LSSVM)模型识别对4 个产地的海参校正集识别率达到100%,预测集识别率为97.67%。此研究基于NIR联合区间向后区间偏最小二乘法(backwards interval partial least squares,Bi PLS)挑选特征波段建立海参NIR与胶原蛋白含量的偏最小二乘法(partial least squares,PLS)预测模型,还可实现对不同产地海参中胶原蛋白的定量检测。
尽管利用红外光谱鉴别时需要大量测量值已知的样品建立数据分析模型,但红外光谱技术不需要繁琐的样品制备,可对完整的食品样本进行无损检测,是一种快速、简单、绿色的鉴定方法,在海参产地溯源中具有较好的应用前景。
2.2.1.2 核磁共振
核磁共振(nuclear magnetic resonance,NMR)根据原子核跃迁产生的共振图谱特征达到对样品鉴定的目的。常用的原子核有1H、13C、17O、31P和23Na等。其中,氢核磁共振(1H-NMR)只有1 个中子,具有很强的磁矩,因食品中各组分都含有氢元素,使得其在食品检测中应用广泛[22]。目前,NMR技术在海参鉴别中的研究较少,主要集中在利用图谱信息解析海参多糖结构。海参体壁的多糖主要分为海参硫酸软骨素(sea cucumber chondroitin sulfate,SC-CHS)和海参岩藻聚糖硫酸酯(sea cucumber fucoidan,SC-FUC)两大类,不同种类的海参其多糖结构有一定的差异。陈士国等[23]从阿拉斯加刺参、日本刺参、墨西哥刺参、八刺参、北极海参、冰岛刺参、明秃参、海地瓜8 种海参中的粗多糖中分离纯化到SC-CHS,利用高温1H-NMR技术比较了不同海参的SC-CHS结构,结果显示8 种海参的SC-CHS异头氢信号差异明显,表明通过SC-CHS的图谱差异可鉴别不同种类的海参。与此研究类似,Wu Nian等[24]利用傅里叶变换红外光谱分析仪结合高温1H-NMR技术对挪威海岸的Holothuria vagabunda、印度太平洋的Holothuria graeffei、西印度洋的Stichopus tremulu和大西洋的Isostichopus badionotu 4 种海参的粗多糖中分离纯化的SC-FUC结构进行了鉴定,根据SC-FUC异头氢信号差异初步建立了不同海域的海参指纹图谱,对于海参的产地是否与SC-FUC的结构有一定关系还需要扩大不同海域的海参样本数量进一步研究。
2.2.2 高效液相色谱
高效液相色谱(h i g h p e r f o r m a n c e l i q u i d chromatography,HPLC)技术以液体作为流动相,靠分离组分的分子与流动相争夺吸附表面活性中心的能力差别而实现对混合物的快速分离,检测过程中无需衍生化,被广泛用于鉴别蛋白质、氨基酸、碳水化合物、酚类化合物等物质[25]。
皂苷是HPLC在海参真实属性鉴定中的主要检测对象。海参皂苷的组成和含量受分类和生长环境的影响。通过确定皂苷寡糖链中单糖的结构、种类、比例,可对海参进行分类鉴定[26]。有研究采用HPLC技术采集了8 种市售海参的总皂苷特征图谱,发现不同海参的皂苷组成和含量存在明显差异。其中,海参科含有的皂苷种类较少,同一属的海参皂苷组成有交叉;刺参科含有的皂苷组成复杂并有其独特性,根据总皂苷色谱峰数目和保留时间的差异可有效区分这8 种海参[27]。在此研究基础上,Yang Jie等[28]将HPLC和电喷雾电离质谱法结合,建立了一种分离海参三萜皂苷的新型方法。根据三萜皂苷的含量和特殊结构可区分不同种类的海参,并将此方法成功应用于8 种市售海参的物种鉴定。虽然HPLC在海参种类鉴定方面发挥了很大的作用,但采用HPLC技术对皂苷结构进行分析的方法在海参产地鉴别方面却未取得理想的效果。于林芳等[29]对皂苷作为指标鉴别海参产地的可行性进行了研究,采用HPLC建立了10 批不同产地的仿刺参的皂苷指纹图谱,结果发现虽然个别产地的海参皂苷含量相对比值有所差异,但这种个体差异是正常的现象,不能据此鉴别出不同产地的仿刺参,对于HPLC技术利用皂苷鉴别不同产地的仿刺参还需要扩大样本数量进一步研究。
利用HPLC技术对海参样品的种属特异性化学物质进行检测,可实现鲜海参、初级加工的干海参物种的鉴定。此方法具有较高的准确性、精密度和可重复性,但不适用于深加工海参产品的鉴别。这主要是因为海参加工制品中含有的其他配料不利于特定化学组分的分离纯化和结构鉴定,而且化学物质结构和组成在加工过程中可能会受到高温、酸、碱等处理而发生变化,影响鉴定结果的准确性。
2.2.3 质谱技术
2.2.3.1 稳定同位素比率质谱
稳定同位素比率质谱(isotope ratio mass spectrometry,IRMS)的工作原理是将样品转化为气体,在离子源和电磁作用下离子得到分离,将离子束强度转化为同位素丰度进而得到同位素比值(如13C/12C、2H/1H、15N/14N等)。IRMS具有操作简单、准确性和灵敏度高等优点,目前已经初步应用于海参的产地溯源中。但此方法的鉴定结果容易受到实验外界环境条件的影响,导致鉴定结果不稳定[30]。王志锐[31]采用IRMS技术对不同采样点的仿刺参肠道、体壁、疣足的δ13C和δ15N指纹特征进行了研究,根据海参肠道的δ13C和δ15N值的分布特征差异可鉴别出渤海湾和黄海湾的大部分海参,不同海域下的少数海参样品的δ13C和δ15N值分布区域有重叠;同时,同一海域不同采样点的海参也可根据海参肠道、体壁的δ13C和δ15N值的特异分布特征区分开。此研究还提出通过海参疣足δ15N值的差异区分海参的生长年限进而区分野生海参和养殖海参具有可行性。针对该研究δ13C和δ15N值分布区域重叠的海参样品,需借助其他的方法进一步鉴别。最近,Zhang Xufeng等[32]也进行了类似的研究,利用IRMS技术分析中国北部海域7 个采样点133 个仿刺参样品的δ13C和δ15N值,将数据进行PCA分析和判别分析(discriminant analysis,DA),结果得到不同地理区域分布的仿刺参体壁δ13C和δ15N值,发现除了烟台担子岛和大连长海岛的δ13C和δ15N同位素组成有部分重叠,其他5 个采样点的仿刺参δ13C和δ15N值都存在差异,但是大连瓦房店和皮口的海参同位素差异不明显,可能是因为大连瓦房店和皮口地理位置较近或两地的海参食物来源相近。因此仅利用IRMS技术对海参的δ13C和δ15N值的检测不能完全实现对不同产地海参的鉴别。
2.2.3.2 电感耦合等离子质谱
电感耦合等离子质谱(inductively coupled plasma mass spectrometry,ICP-MS)技术是一种可以同时对多个无机微量元素进行检测的技术,具有高灵敏度、超痕量检测限以及快速分析等优点[33]。水产品体内的微量元素组成及含量会受其地理环境因素的影响,因此通过检测海参体内的微量元素差异可以鉴别不同产地的海参。Liu Xiaofang等[34]利用ICP-MS技术对我国渤海、黄海和东海3 个海域的39 个刺参体内Al、Fe、Mn、Zn、Cu、V、Cr、Co、As、Ni、Se、Mo、Hg、Pb、Cd共15 种元素进行检测,结合PCA、聚类分析(cluster analysis,CA)以及线性判别分析(linear discriminant analysis,LDA)3 种统计学方法,结果显示3 种分析方法对刺参产地的识别交叉率为100%,大幅降低了误判率,证实ICP-MS技术可应用于中国三大海域刺参的产地溯源。
此外,在利用ICP-MS技术应注意尽管海参体内的微量元素受地域因素的影响常会有很大差异,但这种差异并不稳定,受海参食物来源改变的影响,一些污染元素如Pb、Zn等,其含量在地域之间的差异也可能会随着时间的推移而发生转变。ICP-MS技术的另一个缺点是样品的前处理与分析费时费力,且价格昂贵,尤其在分析挥发性元素时,对样品处理的要求极高。
2.2.3.3 表面解吸常压化学电离质谱
近几年新兴的表面解吸常压化学电离质谱(desorption atmospheric pressure chemical ionizationmass spectrometry,DAPCI-MS)技术无需经过样品预处理,在常温、常压、无解吸溶剂的作用下,即可通过对样品表面痕量物质的解吸电离从而获取样品的指纹图谱。此方法作为一种样品无损检测方法,在食品真伪鉴别中具有广阔的应用前景[35]。目前,DAPCI-MS技术已经初步应用于海参的产地溯源。Wu Zhongchen等[36]通过DAPCI-MS方法获得我国威海、大连、烟台产的162 个刺参的质谱图,发现m/z集中在50~800区域质谱具有显著强度的信号。结合PCA和SIMCA模型对指纹图谱进行分析,发现根据海参指纹图谱间的差异可成功区分3 个不同产地的刺参。此研究实验数据还表明,虽然海参样品表面粗糙度在一定程度上影响质谱信号水平,但这并不会影响干海参产地鉴别的准确性,证明DAPCI-MS有望成为一种方便、实时、快速的产地鉴别分析技术。
2.2.3.4 气相色谱-质谱联用
气相色谱-质谱联用(gas chromatography-mass spectrometer,GC-MS)技术充分发挥了气相色谱高效分离和质谱定性能力强的特点,使鉴定结果更加准确。脂肪酸是GC-MS在海参鉴别中的主要研究对象,Zhang Xufeng等[32]利用GC-MS技术测定我国北部海域不同采样点的仿刺参的脂肪酸成分含量,发现不同产地的仿刺参样品中脂肪酸含量均存在显著差异;莱州的仿刺参单不饱和脂肪酸和饱和脂肪酸含量高,但多不饱和脂肪酸含量低;而产于獐子岛的仿刺参样品饱和脂肪酸含量最低,多不饱和脂肪酸含量高。通过脂肪酸成分含量差异结合PCA分析可以有效鉴定不同地理区域分布的仿刺参。在GC-MS基础上,Liu Yu等[37]联合使用GC-MS和IRMS建立了一种新型的特定化合物同位素分析法。将气相色谱分离出的不同产地刺参的脂肪酸注入稳定的同位素比值质谱仪,研究脂肪酸的稳定碳同位素组成,并结合多元统计分析和判别分析方法进行数据处理,结果表明除了长海岛和獐子岛,其他不同产地和不同季节的刺参根据脂肪酸的稳定碳同位素显著差异可有效区分开。此方法联合使用气相色谱和同位素质谱结合统计学分析,避免了混合化合物中其他组分的影响,可为海参的产地溯源和鉴定提供理论依据,同时也可为其他水产品的稳定同位素比值和脂肪酸成分的分析提供参考。这一研究也提示在食品真伪鉴别中,多种检测方法联合使用可以打破单种方法的局限性。
2.3 分子生物学鉴定
以DNA为基础的分子生物学鉴定是近十年来发展最快的真伪鉴别技术。DNA稳定性强,不受生物体生长发育阶段、生理状态等因素影响,具有特异性好、灵敏度高、准确性强等优点。而且DNA分子耐热性强,即使在深加工产品中,部分DNA分子发生降解断裂,仍能提取出小片段的DNA。因此,基于DNA的分子生物学方法在食品鉴别中应用越来越广泛。目前应用于海参鉴定的分子生物学技术主要包括聚合酶链式反应(polymerase chain reaction,PCR)技术、分子指纹图谱技术、DNA条形码技术等。
2.3.1 PCR技术
以DNA为基础的PCR技术是食品鉴别常用的方法之一。最早发展起来的PCR技术是普通PCR,一个体系中只有一对特异性引物和单一模板。Wen Jing等[38]根据11 种海参设计出11 个种特异性PCR反向引物,结合通用正向引物16 Sar序列进行PCR扩增,得到269~406 bp的种特异性16S rRNA片段。此方法成功应用于市售海参样品鉴定中,并检测出2 种市售冷冻海参以次充好。随后,2014年Wen Jing等[39]利用上述技术又设计了10 个种特异性PCR反向引物,成功鉴定出另外10 种海参物种。此方法操作简单、特异性强,可用于单一成分的市售干海参鉴别中,但由于扩增目的片段长度差异较小,无法区分混合加工制品中的海参物种。在普通PCR的基础上发展起来的多重PCR为混合海参物种的鉴定提供了新的工具。它是在一个PCR反应中加入多对特异性引物,同时扩增出多个具有明显长度差异的目的片段的PCR技术。整个多重PCR过程中最关键的步骤就是引物的设计和反应体系、反应条件的优化(如引物浓度、退火温度等),这些关键点给多重PCR技术的实验设计带来了困难。Zuo Tao等[40]基于692 bp的线粒体细胞色素c氧化酶亚基I(cytochrome oxidase subunit I,COI)基因片段,针对仿刺参、北大西洋海参、梅花参和红海参分别设计了4 对特异性引物,得到不同长度的扩增产物,通过PCR产物的电泳条带位置就可明显区分这4 种海参。该方法的灵敏度达到纳克级别,可以同时检测出混合样品中的这4 种海参物种。
实时荧光定量PCR技术(real-time quantitative PCR,qPCR)是PCR在海参物种鉴定中的另外一个应用。此技术是利用荧光信号的变化实时监测PCR过程,模板Ct值与每个模板起始拷贝数的对数存在线性关系,起始拷贝数越多,Ct值就越小。通过Ct值和拷贝数建立的标准曲线可对样品进行定性和定量分析[41]。qPCR的常见方法主要包括染料法和探针法两种。染料法是在PCR体系中加入荧光染料如SYBR Green I,PCR反应产物通过熔解曲线进一步分析,此方法适用性广、成本较低,但每孔只能检测一个目标基因;探针法体系中除了特异性的引物还需要特异性的探针才能释放荧光信号,此方法可同时扩增多个目标基因,但合成探针价格较高,而且不同引物探针体系的选择、验证会产生庞大的工作量。目前TaqMan探针qPCR技术已经用于仿刺参的鉴定。曹际娟等[42]基于仿刺参线粒体COI基因、NAD 4基因分别设计特异性引物和探针,所建立的qPCR方法的变异系数小于2%,重复性良好,可应用于仿刺参的快速鉴定。此外,该研究扩增的靶基因片段长度均小于150 bp,因此建立的方法也适用于经过高压或高温等处理过的DNA容易降解的深加工海参制品的物种来源鉴定。随着多重qPCR的发展,将qPCR和多重PCR结合,实现多成分的检测也将是建立海参快速鉴别方法研究的方向之一。
2.3.2 分子指纹图谱技术
2.3.2.1 限制性片段长度多态性
限制性片段长度多态性(restriction fragment length polymorphism,RFLP)利用通用引物扩增目标DNA片段,采用不同的限制性内切酶将PCR产物进行酶切,根据酶切产物的凝胶电泳条带鉴别不同基因型[43]。PCR-RFLP技术最关键的步骤是限制性内切酶的选择,要考虑不同地理分布个体的目标片段序列差异[44]。文菁等[8]采用3 种核酸内切酶(Dde I、Hae III和Sty I)对16 种海参570 bp左右的16S rRNA基因片段进行酶切,分别产生10、5 种和5 种单倍型,通过单倍型的组合可有效区分16 种海参的种类;将此方法应用于19 种商品海参冻品及干品的鉴定中,发现9 种海参产品出现标签错误的现象。PCR-RFLP技术应用于海参物种鉴定的另一个靶序列对象是COI基因,Lü Yingchun等[45]用限制性内切酶BamH I、Kpn I、Pst I、Xba I 和Eco31 I对692 bp的COI基因片段进行酶切,根据酶切DNA图谱结果成功鉴别出仿刺参、大西洋瓜参、梅花参、加州拟刺参和子安辐肛参这5 种海参。
虽然PCR-RFLP技术操作简单、成本低,但此技术也存在一些缺点:此方法只适用于单一成分的样品鉴定,混合成分的样品会干扰实验结果;种内变异可能使目标序列某一特定的酶切位点消失或产生新的酶切位点,PCR产物酶切片段长度差异不大,可能造成结果假阴性[46];此外,当PCR-RFLP技术应用于深加工食品鉴定时,由于深加工产品的DNA可能发生断裂,只能提取到较短的DNA,选择合适的限制性酶切位点较难。
2.3.2.2 随机扩增多态性DNA
随机扩增多态性DNA(random amplified polymorphic DNA,RAPD)的技术原理是将随机多态核苷酸序列作为引物进行PCR扩增,通过电泳检测扩增产物多态性来反映基因组DNA的多态性。目前PCR-RAPD技术已经用于海参遗传多样性分析和产地溯源的研究中。Yun Zhenyu等[47]利用PCR-RAPD技术和基因片段测序技术评估辽宁大连、山东烟台和威海这3 个产地的仿刺参的遗传关系和DNA多态性。结果发现不同海域的海参各自具有独特的基因特征,RAPD指纹图谱CA结果显示除了个别样本外,大部分来自同一海域的样本都聚集在同一分支中,大连仿刺参单独聚为一个分支,烟台和威海的仿刺参样品地理位置相近,聚为一个分支。研究表明根据PCR-RAPD指纹图谱可区分开这3 个产地的大部分仿刺参,个别不能区分的样品还需要进一步鉴别。
对于基因组序列信息不了解的物种鉴定,PCR-RAPD技术可以作为一种简便、快速的分子生物学鉴定方法。但该方法的主要缺点在于反应结果容易受反应体系各参数的影响,可重复性差[48]。当目标DNA的量少或发生降解时,物种特异性的长片段无法有效扩增或两个不同物种的PCR产物长度类似,都会导致假阳性结果的产生[49]。
2.3.3 DNA条形码技术
随着DNA测序成本的降低,利用DNA序列鉴别物种的方法逐渐成熟。2003年,Herbert等[50]最早提出DNA条形码的概念。DNA条形码是利用一段或几段标准的、较短的DNA序列作为标记实现物种鉴定的技术[51]。其技术核心是利用通用引物扩增目的基因片段,测序后通过数据库比对确定物种信息。随着全球物种基因条形码的发展,结合已有的生命条形码联盟(Consortium for the Barcode of Life,CBOL)数据库和美国国立生物技术信息中心(National Center for Biotechnology Information,NCBI)数据库,为食品的真伪鉴别提供了新的工具。
DNA条形码技术应用于海参物种鉴定的靶基因主要包括线粒体的COI、16S rRNA以及18S rRNA[68]和12S rRNA[69]。表2总结了利用DNA条形码技术进行海参物种鉴定的相关研究。由于CBOL和NCBI数据库中部分海参的12S rRNA和18S rRNA基因序列缺乏,使12S rRNA和18S rRNA基因作为基因条形码对海参的物种鉴定具有一定的局限,这也说明仍然需要大量的研究工作才能建立一个完善、可靠的海参DNA条形码数据库。目前常用COI和16S rRNA基因序列作为海参的条形码区域。李颖等[52]对11 个海参3 段线粒体基因(IrRNA-COI、16S rRNA和COI)片段进行扩增与测序,通过比较后发现COI基因保守性较好,其没有插入或缺失位点,而且多态位点比例较低,适合作为海参分类的分子标记。随后梁君妮等[53]也证实了利用COI基因片段可以有效鉴定海参物种,通过对海参线粒体COI基因进行测序可区分不同种类的海参,并将此方法成功地应用于海参营养液的物种鉴定中。Uthicke等[54]在分析商业海参物种的基因信息时,发现了96 种新的COI基因序列,使得利用基因信息鉴定商用海参的种类更加广泛。最近,Amin等[55]对从印度尼西亚东爪哇收集到的一种海参进行物种鉴定,结合形态学鉴定和基于COI基因的DNA条形码鉴定结果判定此物种为尻参科的海地瓜(Acaudina molpadioides)。此外,DNA条形码技术已经在市售海参产品标签的调查研究中得到应用。Wen Jing等[57]采用DNA条形码技术基于COI基因对市售海参科样品的种类进行鉴定,发现有7 个海参种类鉴定结果与商业标签名称不一致,样品标签不符率为63.6%。随后又基于16S rRNA序列对市售刺参科海参的种类进行鉴定,发现其中一个商业标签为刺参(仿刺参属)的物种其DNA条形码鉴定结果为暗刺参属[58]。
表2 DNA条形码技术在海参物种鉴定中的相关研究
Table 2 Summary of previous esearch on species identification of sea cucumber by DNA barcoding
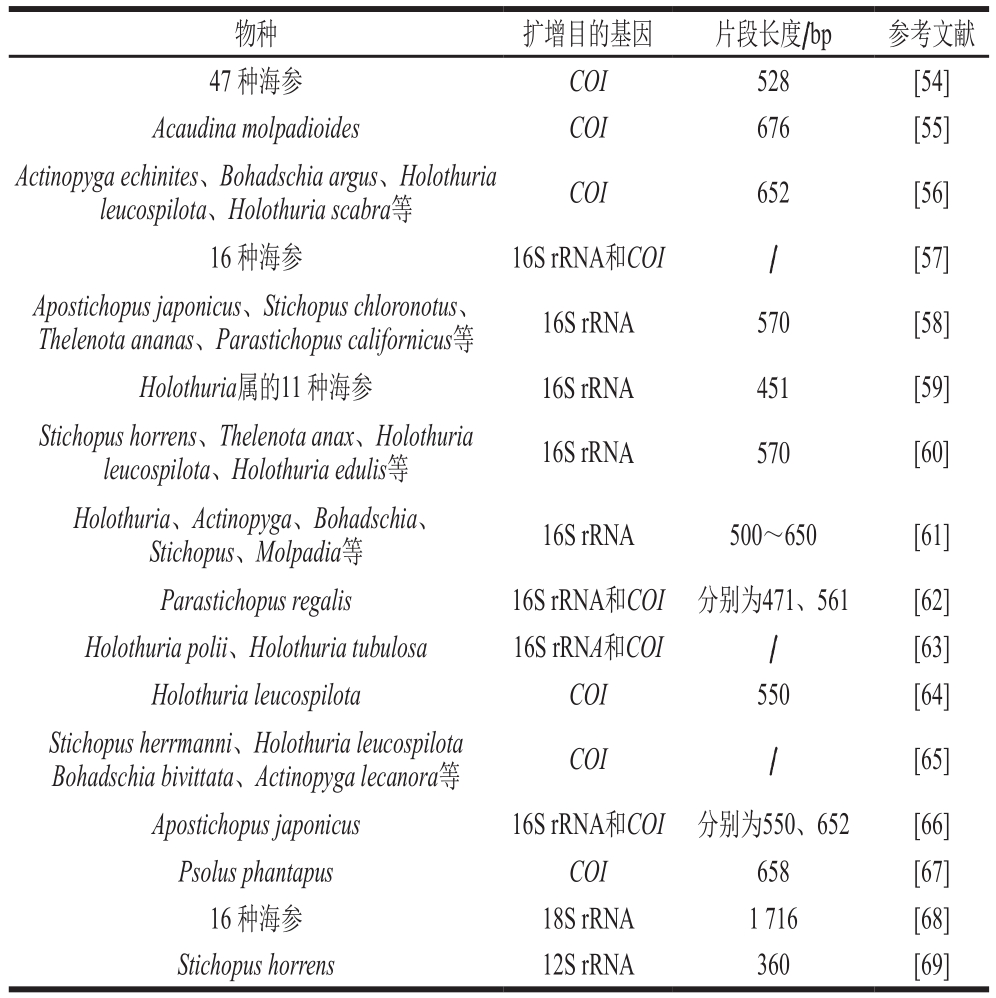
注:/.未知。
物种 扩增目的基因 片段长度/bp 参考文献47 种海参COI 528 [54]Acaudina molpadioides COI 676 [55]Actinopyga echinites、Bohadschia argus、Holothuria leucospilota、Holothuria scabra等COI 652 [56]16 种海参 16S rRNA和COI / [57]Apostichopus japonicus、Stichopus chloronotus、Thelenota ananas、Parastichopus californicus等 16S rRNA 570 [58]Holothuria属的11 种海参 16S rRNA 451 [59]Stichopus horrens、Thelenota anax、Holothuria leucospilota、Holothuria edulis等 16S rRNA 570 [60]Holothuria、Actinopyga、Bohadschia、Stichopus、Molpadia等 16S rRNA 500~650 [61]Parastichopus regalis 16S rRNA和COI 分别为471、561 [62]Holothuria polii、Holothuria tubulosa 16S rRNA和COI / [63]Holothuria leucospilota COI 550 [64]Stichopus herrmanni、Holothuria leucospilota Bohadschia bivittata、Actinopyga lecanora等COI / [65]Apostichopus japonicus 16S rRNA和COI 分别为550、652 [66]Psolus phantapus COI 658 [67]16 种海参 18S rRNA 1 716 [68]Stichopus horrens 12S rRNA 360 [69]
标准的DNA条形码技术要求从样品中能提出比较完整的DNA片段,一般长度在500~700 bp,可适用于新鲜或初级加工食品的鉴定中。但对于深加工产品例如罐头等,加工过程中的高温、高压和辐射以及酸碱、添加剂的加入等均会使原料DNA片段发生不同程度的断裂、降解或变性等,给标准DNA条形码技术的应用带来很大困难,这就需要采用DNA条形码(小于300 bp)来提高检测成功率[70-71]。随着市场需求推出的海参深加工制品越来越多,针对深加工海参制品寻找条形码用以区分海参物种是目前DNA条形码在海参真伪鉴别应用研究中的方向之一。此外,DNA条形码技术应用于海参物种鉴定依赖于测序仪,不利于现场检测。最近,针对食品掺假结合纳米技术和DNA条形码技术开发的一种纳米示踪剂,简化了标准DNA条形码相关的分析步骤(即DNA提取、条码扩增和物种鉴定),可方便携带,不依赖于专业实验室的测序就可实现物种鉴别[72],使DNA条形码技术在海参真伪鉴别中有更广阔的应用前景。
3 海参鉴别方法比较
上述用于海参鉴别的三大类方法各有其优缺点。形态学鉴定操作简单、费用低,适用于初步的海参物种鉴定,对于形态学特征消失的海参加工制品无法准确鉴定。理化鉴定技术结合化学计量学方法主要实现的是对海参产地的鉴别。在我国不同产地的仿刺参质量和价格不同,产地造假时常发生,色谱、红外光谱、质谱等技术为海参产地溯源提供了技术手段。但是,理化鉴定技术在鉴别准确性、稳定性等方面还有待提高。基于核酸分析的分子生物学检测方法不受季节、加工方法、产品形态的影响,鉴别结果准确度较高,在海参的物种鉴定中更具有优势,对产地的鉴别有一定的局限性。但根据不同产地的物种遗传多样性与地理来源是否有相关性,也可以在一定程度上区分海参产地。表3对三大类方法的主要特点、应用和局限性进行比较分析。没有哪一种技术在食品鉴别上是完美的,每一种技术均为解决食品掺假造假问题提供不同层次的技术支撑。根据适用条件以及鉴别目的需求选择适合的方法才能达到最有效的鉴定。此外,食品鉴伪研究正逐渐由单一技术的应用转变为多个技术辅助应用的方向发展。因此,鉴别研究不能局限于单一方法,必须要构建一个完善的鉴别技术体系。
表3 海参不同鉴别技术的比较
Table 3 Comparison of different techniques used for identification of sea cucumber

方法类别 方法 主要特点 目前在海参鉴别中的应用 局限性形态学鉴定 显微镜观察 方便、快捷、价格低廉 物种鉴定依赖有丰富鉴别经验的专业人员,对于加工过的海参制品无法准确鉴定HPLC 操作简单,较高的准确性、精密度和可重复性 产地鉴别可实现无损检测 产地鉴别 需要大量测量值已知的样品建立数据分析模型NMR 高重现性和灵敏度 物种鉴定和产地鉴别 灵敏度低、费用高IRMS 高准确性和灵敏度 产地鉴别 易受实验外界环境条件的影响,结果不稳定GC-MS 操作简单、高灵敏度 产地鉴别 难气化的化学组分鉴别易受干扰ICP-MS 超痕量检测限、高灵敏度 产地鉴别 样品的前处理要求高,对环境要求高,费用较高DAPCI-MS 快速、无污染,不利于海参复杂加工制品的鉴定,化学组分的分离纯化较难IR 快速、简单、价格低廉,理化鉴定可实现无损检测 产地鉴别 稳定性差,受样品表面粗糙度干扰强、高准确性和灵敏度 物种鉴定 引物的设计和反应体系的优化较难PCR-RFLP 重复性好,共显性 物种鉴定 鉴别位点受内切酶种类限制,易产生假象普通PCR 操作简单、省时、特异性分子生物学鉴定PCR-RAPD 无需专门设计引物,所需DNA量极少,简单省时 产地鉴别 不稳定、重复性差DNA条形码技术不受形态和生物个体发育的影响,准确、高效,易标准化物种鉴定近缘物种难以区分,依赖数据库的数据,不适于混合多组分的定性
4 结 语
科学、高效的海参鉴别技术是建立和完善海参鉴伪技术标准体系的基础。通过对上述多项海参鉴伪技术的深入思考发现,快速、简便、高通量、精准化仍然是海参鉴别技术的发展趋势。在今后海参产地鉴别研究中还需在一定程度上提高鉴别的准确度,增加样本数量,进一步开展化学计量学统计方法的有效性研究,筛选出最适的化学计量学方法,进而建立不同产地、不同种类海参的判别模型和相应的数据库。市场上海参深加工制品的不断推出也给海参的分子生物学鉴定带来了一定的挑战。加工制品中可能是多物种混合成分,从高度加工制品中提取出的DNA微量或DNA片段发生断裂,这些都会给物种鉴别带来困难。因此寻找较短的靶序列可应用于深加工制品的鉴定,并能在混合样本中定性检测以及定量分析是DNA检测技术的研究工作之一。新兴的高通量二代测序技术可同时对大量多物种混合样本分析,实现对降解、微量的或低质量的DNA样品进行检测,对未知食品、复杂食品或者深加工食品来说有着非常重要的作用,已经应用于食品物种鉴定领域[73],为分析食品中的物种成分提供新的研究工具。另一种高通量技术DNA微阵列可同时识别多种物种,显著提高了检测通量和速度,也将是海参DNA检测技术的发展方向之一。除此之外,快速的现场鉴别技术是目前食品检测的需求。环介导等温PCR、与荧光结合的DNA纳米技术等检测方法可以直接通过肉眼观察结果,对建立海参现场鉴别方法具有一定的启发。
参考文献:
[1] 郭盈莹, 丁燕, 徐飞飞, 等. 海参中主要生物活性成分研究进展[J].食品科学, 2014, 35(15): 335-344. DOI:10.7506/spkx1002-6630-201415066.
[2] AMININ D L, MENCHINSKAYA E S, PISLIAGIN E A, et al.Anticancer activity of sea cucumber triterpene glycosides[J]. Marine Drugs, 2015, 13(3): 1202-1223. DOI:10.3390/md13031202.
[3] 王婷, 刘京熙, 张健, 等. 海参精多糖提取工艺优化及其体外抗肿瘤活性[J]. 食品工业科技, 2016, 37(17): 68-74. DOI:10.13386/j.issn1002-0306.2016.17.005.
[4] 何丽霞, 陈启贺, 刘睿, 等. 海参寡肽: 免疫调节作用及机制研究[J]. 科技导报, 2016, 34(11): 42-47. DOI:10.3981/j.issn.1000-7857.2016.11.007.
[5] LI Rongfeng, YU Huahua, YUE Yang, et al. Sulfated polysaccharides with antioxidant and anticoagulant activity from the sea cucumber Holothuria fuscogliva[J]. Chinese Journal of Oceanology and Limnology, 2017, 35(4): 763-769. DOI:10.1007/s00343-017-5339-7.
[6] ROBINSON G, LOVATELLI A. Global sea cucumber fisheries and aquaculture FAO’s inputs over the past few years[J]. FAO Aquaculture Newsletter, 2015, 53: 55-57.
[7] PURCELL S, CHOO P S, AKAMINE J, et al. Alternative product forms, consumer packaging and extracted derivatives of tropical sea cucumbers[J]. SPC Beche-de-mer Information Bulletin, 2014, 34: 47-52.
[8] 文菁, 胡超群, 张吕平, 等. 16 种商品海参16S rRNA的PCR-RFLP鉴定方法[J]. 中国水产科学, 2011, 18(2): 451-457. DOI:10.3724/SP.J.1118.2011.00451.
[9] PURCELL S W, SAMYN Y, CONAND C. Commercially important sea cucumbers of the world[M]. Rome: Food and Agriculture Organization, 2012: 150.
[10] PURCELL S W, HAIR C A, MILLS D J. Sea cucumber culture,farming and sea ranching in the tropics: progress, problems and opportunities[J]. Aquaculture, 2012, 368/369: 68-81. DOI:10.1016/j.aquaculture.2012.08.053.
[11] 赵欣涛. 世界渔业中现有商品海参的生物分类学名称归类概述[J]. 农业开发与装备, 2016(12): 38. DOI:10.3969/j.issn.1673-9205.2016.12.031.
[12] 廖玉麟. 中国动物志, 棘皮动物门, 海参纲[M]. 北京: 科学出版社,1997: 65-284.
[13] 姚旺, 顾炎斌, 吴古远, 等. 西沙群岛6 种海参骨片的超显微结构研究[J]. 中国海洋大学学报(自然科学版), 2017, 17(1): 76-81.DOI:10.16441/j.cnki.hdxb.20160012.
[14] KAMARUDIN K R, REHAN M M. Morphological and molecular identification of Holothuria (Merthensiothuria) leucospilota and Stichopus horrens from Pangkor Island, Malaysia[J]. Tropical Life Sciences Research, 2015, 26(1): 87-99.
[15] KAMARUDIN K R, REHAN M M, BAHAMAN N A. Morphological and molecular identification of sea cucumber species Holothuria scabra, Stichopus horrens and Stichopus ocellatus from Kudat, Sabah,Malaysia[J]. Pertanika Journal of Tropical Agricultural Science, 2017,40(1): 161-172.
[16] 文菁, 曾玲, 王雁, 等. 运用形态学和分子生物学方法鉴定市场中伪劣海参产品[J]. 湛江师范学院学报, 2012, 33(3): 95-99.DOI:10.3969/j.issn.1006-4702.2012.03.018.
[17] 孙昭, 刘贞, 云振宇, 等. 海参分类鉴定技术及标准化现状[J].农产品加工(学刊), 2014(24): 72-75. DOI:10.3969/jissn.1671-9646(X).2014.12.052.
[18] 李梅阁, 吴亚君, 杨艳歌, 等. 浆果果汁真伪鉴别技术研究进展[J].食品科学, 2016, 37(13): 243-250. DOI:10.7506/spkx1002-6630-201613044.
[19] WU Zhongchen, TAO Lin, ZHANG Pengyan, et al. Diffuse reflectance mid-infrared Fourier transform spectroscopy (DRIFTS)for rapid identification of dried sea cucumber products from different geographical areas[J]. Vibrational Spectroscopy, 2010, 53(2): 222-226.DOI:10.1016/j.vibspec.2010.03.008.
[20] 陶琳, 武中臣, 张鹏彦, 等. 近红外光谱法快速鉴定干海参产地[J]. 农业工程学报, 2011, 27(5): 364-366. DOI:10.3969/j.issn.1002-6819.2011.05.063.
[21] 邹小波, 薛瑾, 石吉勇, 等. 基于近红外光谱技术的海参品质快速检测[J]. 食品安全质量检测学报, 2017, 8(9): 3431-3437.
[22] WU L, DU B, VAN DER HEYDEN Y, et al. Recent advancements in detecting sugar-based adulterants in honey: a challenge[J].Trends in Analytical Chemistry, 2017, 86: 25-38. DOI:10.1016/j.trac.2016.10.013.
[23] 陈士国, 李国云, 尹利昂, 等. 高温1H-NMR鉴别8 种海参硫酸软骨素[J]. 分析测试学报, 2010, 29(8): 782-786. DOI:10.3969/j.issn.1004-4957.2010.08.005.
[24] WU Nian, CHEN Shiguo, YE Xingqian, et al. Identification of fucans from four species of sea cucumber by high temperature1H-NMR[J].Journal of Ocean University of China, 2014, 13(5): 871-876.DOI:10.1007/s11802-014-2287-0.
[25] 刘先德. 高效液相色谱在食品质量检测中的应用[J]. 中国高新技术企业, 2015(18): 50-52. DOI:10.13535/j.cnki.11-4406/n.2015.18.026.
[26] 邹峥嵘, 易杨华, 张淑瑜, 等. 海参皂苷研究进展[J]. 中国海洋药物,2004, 23(1): 46-53.
[27] 于林芳, 薛长湖, 董平, 等. 八种海参体内皂苷类成分的SPE-HPLC分析比较[J]. 分析试验室, 2011, 30(1): 13-15. DOI:10.13595/j.cnki.issn1000-0720.2011.0004.
[28] YANG Jie, WANG Yuanhong, ZHANG Ran, et al. Determination of the triterpene glycosides in sea cucumbers by liquid chromatography with evaporative light scattering and mass spectrometry detection[J].Journal of Separation Science, 2015, 38(7): 1117-1122. DOI:10.1002/jssc.201401253.
[29] 于林芳, 董平, 薛长湖, 等. 仿刺参中皂苷类成分的高效液相色谱指纹图谱[J]. 色谱, 2010, 28(9): 885-888. DOI:10.3724/SP.J.1123.2010.00885.
[30] LI L, BOYD C E, SUN Z. Authentication of fishery and aquaculture products by multi-element and stable isotope analysis[J]. Food Chemistry, 2016, 194: 1238-1244. DOI:10.1016/j.foodchem.2015.08.123.
[31] 王志锐. 地理标志产品的碳氮稳定同位素指纹特征[D]. 大连: 大连海事大学, 2014: 45-46.
[32] ZHANG Xufeng, LIU Yu, LI Ying, et al. Identification of the geographical origins of sea cucumber (Apostichopus japonicus)in northern China by using stable isotope ratios and fatty acid profiles[J]. Food Chemistry, 2017, 218: 269-276. DOI:10.1016/j.foodchem.2016.08.083.
[33] 郭秋兰, 冯志强, 宋美英. 电感耦合等离子质谱在食品元素分析检测中的应用[J]. 食品与发酵科技, 2015, 51(5): 100-103.DOI:10.3969/j.issn.1674-506X.2015.05-024.
[34] LIU Xiaofang, XUE Changhu, WANG Yuming, et al. The classification of sea cucumber (Apostichopus japonicus) according to region of origin using multi-element analysis and pattern recognition techniques[J]. Food Control, 2012, 23(2): 522-527. DOI:10.1016/j.foodcont.2011.08.025.
[35] 李倩, 王姜, 陈焕文, 等. 表面解吸常压化学电离质谱法快速鉴别羊肉真伪[J]. 质谱学报, 2014, 35(6): 502-508. DOI:10.7538/zpxb.youxian.2014.0022.
[36] WU Zhongchen, CHEN Huanwen, WANG Weiling, et al.Differentiation of dried sea cucumber products from different geographical areas by surface desorption atmospheric pressure chemical ionization mass spectrometry[J]. Journal of Agricultural and Food Chemistry, 2009, 57(20): 9356-9364. DOI:10.1021/jf9018504.
[37] LIU Yu, ZHANG Xufeng, LI Ying, et al. The application of compound-specific isotope analysis of fatty acids for traceability of sea cucumber (Apostichopus japonicus) in the coastal areas of China[J].Journal of the Science of Food and Agriculture, 2017, 97(14): 4912-4921. DOI:10.1002/jsfa.8367.
[38] WEN Jing, HU Chaoqun, ZHANG Lüping, et al. Identification of 11 sea cucumber species by species-specific PCR method[J]. Food Control, 2012, 27(2): 380-384. DOI:10.1016/j.foodcont.2012.04.007.
[39] WEN Jing, ZENG Ling. Use of species-specific PCR for the identification of 10 sea cucumber species[J]. Chinese Journal of Oceanology and Limnology, 2014, 32(6): 1257-1263. DOI:10.1007/s00343-015-4050-9.
[40] ZUO Tao, LI Zhaojie, LÜ Yingchun, et al. Rapid identification of sea cucumber species with multiplex-PCR[J]. Food Control, 2012, 26(1):58-62. DOI:10.1016/j.foodcont.2012.01.019.
[41] 姚琳, 江艳华, 李青娇, 等. 基于DNA检测技术鉴定水产加工品原料的研究进展[J]. 中国渔业质量与标准, 2013, 3(1): 33-39.
[42] 曹际娟, 徐君怡, 徐杨, 等. 实时荧光聚合酶链式反应技术快速鉴定仿刺参[J]. 食品科学, 2014, 35(10): 145-148. DOI:10.7506/spkx1002-6630-201410026.
[43] 张丽, 张良, 刘书成, 等. DNA技术在海洋食品物种鉴定中的应用[J].遗传, 2010, 32(6): 555-560.
[44] LOCKLEY A K, BARDSLEY R G. DNA-based methods for food authentication[J]. Trends Food Science & Technology, 2000, 11(2):67-77. DOI:10.1016/S0924-2244(00)00049-2.
[45] LÜ Yingchun, ZHENG Rong, ZUO Tao, et al. Identification of five sea cucumber species through PCR-RFLP analysis[J]. Journal of Ocean University of China, 2014, 13(5): 825-829. DOI:10.1007/s11802-014-2276-3.
[46] HERRERO B, VIEITES J M, ESPINEIRA M. Authentication of Atlantic salmon (Salmo salary) using real-time PCR[J]. Food Chemistry, 2011,127(3): 1268-1272. DOI:10.1016/j.foodchem.2011.01.070.
[47] YUN Zhenyu, SUN Zhao, XU Haiyan, et al. Identifying the geographical origin of protected sea cucumbers (Apostichopus japonicus) in China using random amplified polymorphic DNA polymerase chain reaction(RAPD-PCR)[J]. Food Science and Biotechnology, 2017, 26(2): 357-362.DOI:10.1007/s10068-017-0048-8.
[48] REGO I, MARTINEZ A, GONZALEZ-TIZON A, et al. PCR technique for the identification of mussel species[J]. Journal of Agricultural and Food Chemistry, 2002, 50(7): 1780-1784. DOI:10.1021/jf0110957.
[49] LIU Z J, CORDES J F. DNA marker technologies and their applications in aquaculture genetics[J]. Aquaculture, 2004, 238(1):1-37. DOI:10.1016/j.aquaculture.2004.05.027.
[50] HEBERT P D N, RATNASINGHAM S, DE WAARD J R. Barcoding animal life: cytochrome c oxidase subunit I divergences among closely related species[J]. Proceedings of the Royal Society of London B:Biological Sciences, 2003, 270: 96-99. DOI:10.1098/rsbl.2003.0025.
[51] MONIZ M B J, KACZMARSKA I. Barcoding of diatoms: nuclear encoded ITS revisited[J]. Protist, 2010, 161(1): 7-34. DOI:10.1016/j.protis.2009.07.001.
[52] 李颖, 刘萍, 孙慧玲, 等. 仿刺参(Apostichopus japonicus) mtDNA三个基因片段的序列分析[J]. 海洋与湖沼, 2006, 37(2): 143-151.
[53] 梁君妮, 薛长湖, 赵玉然, 等. 海参营养液 DNA 高效快速提取及种类鉴定方法[J]. 食品科学, 2008, 29(5): 269-272.
[54] UTHICKE S, BYRNE M, CONAND C. Genetic barcoding of commercial Bêche-de-mer species (Echinodermata: Holothuroidea)[J].Molecular Ecology Resources, 2010, 10(4): 634-646. DOI:10.1111/j.1755-0998.2009.02826.x.
[55] AMIN M H F, PIDADA I B R, SUGIHARTO, et al. Sea cucumber species identification of family Caudinidae from Surabaya based on morphological and mitochondrial DNA evidence[C]// International Conference and Workshop on Basic and Applied Sciences: Surabaya,Indonesia, October 16-17. AIP Conference Proceedings, 2016,1718(1): 030001. DOI:10.1063/1.4943311.
[56] ARNDT A, MARQUEZ C, LAMBERT P, et al. Molecular phylogeny of eastern Pacific sea cucumbers (Echinodermata: Holothuroidea)based on mitochondrial DNA sequence[J]. Molecular Phylogenetics and Evolution, 1996, 6(3): 425-437. DOI:10.1006/mpev.1996.0091.
[57] WEN Jing, HU Chaoqun, ZHANG Lüping, et al. Genetic identification of global commercial sea cucumber species on the basis of mitochondrial DNA sequences[J]. Food Control, 2011, 22(1): 72-77.DOI:10.1016/j.foodcont.2010.06.010.
[58] WEN J, HU C, ZHANG L, et al. The application of PCR-RFLP and FINS for species identification used in sea cucumbers (Aspidochirotida:Stichopodidae) products from the market[J]. Food Control, 2010,21(4): 403-407. DOI:10.1016/j.foodcont.2009.06.014.
[59] KAMARUDIN K R, REHAN M M, NOOR H M, et al. 16S rRNA barcoding technique for species identification of processed sea cucumbers from selected Malaysian markets[J]. Journal of Science and Mathematics Letters, 2016, 4(1): 10-23.
[60] KAMARUDIN K R, REHAN A M, HASHIM R, et al. Phylogenetic relationships within the genus holothuria inferred from 16S mitochondiral rRNA gene sequences[J]. Sains Malaysiana, 2016,45(7): 1079-1087.
[61] KAMARUDIN K R, HASHIM R, USUP G. Phylogeny of sea cucumber (Echinodermata: Holothuroidea) as inferred from 16S mitochondrial rRNA gene sequences[J]. Sains Malaysiana, 2010,39(2): 209-218.
[62] MAGGI C, GONZÁLEZ-WANGÜEMERT M. Genetic differentiation among Parastichopus regalis populations in the Western Mediterranean Sea: potential effects from its fishery and current connectivity[J]. Mediterranean Marine Science, 2015, 16(3): 489-501.DOI:10.12681/mms.1020.
[63] GONZÁLEZ-WANGÜEMERT M, VALENTE S, AYDIN M. Effects of fishery protection on biometry and genetic structure of two target sea cucumber species from the Mediterranean Sea[J]. Hydrobiologia,2015, 743(1): 65-74.
[64] IDRIS M Y, AHMAD H F, HASHIM R, et al. Phylogeny of Holothuria leucospilota (Echinodermata: Holothuroidea) as inferred from cytochrome c oxidase I mtDNA gene sequences[J]. Borneo Journal of Resource Science and Technology, 2016, 1(1): 52-54.
[65] MADDUPPA H, TAURUSMAN A A, SUBHAN B, et al. DNA barcoding reveals vulnerable and not evaluated species of sea cucumbers (Holothuroidea and Stichopodidae) from Kepulauan Seribu reefs, Indonesia[J]. Biodiversitas, 2017, 18(3): 893-898.DOI:10.13057/biodiv/d180305.
[66] JO J, PARK C, KIM M, et al. Phylogenetic analysis of the three color variations of the sea cucumber Apostichopus japonicus[J].Journal of Aquaculture Research and Development, 2016, 7: 1-4.DOI:10.4172/2155-9546.1000418.
[67] LEE T, SHIN S. A newly recorded sea cucumber of the genus psolus(Holothuroidea: dendrochirotida: Psolidae) from the East Sea of Korea[J]. Animal Systematics, Evolution and Diversity, 2017, 33(3):195-199. DOI:10.5635/ASED.2017.33.3.020.
[68] LACEY K M J, MCCORMACK G P, POWELL R, et al. Phylogenetic relationships within the class Holothuroidea inferred from 18S rRNA gene data[J]. Marine Biology, 2005, 147: 1149-1154. DOI:10.1007/s00227-005-0009-2.
[69] KAMARUDIN K R, REHAN M M, NOOR H M, et al. Comparisan of ossicle shape and 12S rRNA gene sequencing techniques for species identification of Gamat-based beche-demer from Langkawi Island,Kedah[J]. Pertanika Journal of Tropical Agricultural Science, 2017,40(2): 305-318.
[70] ARMANI A, GUARDONE L, LA CASTELLANA R, et al. DNA barcoding reveals commercial and health issues in ethnic seafood sold on the Italian market[J]. Food Control, 2015, 55: 206-214.DOI:10.1016/j.foodcont.2015.02.030.
[71] SHOKRALLA S, HELLBERG R S, HANDY S M, et al. A DNA mini-barcoding system for authentication of processed fish products[J].Scientific Reports, 2015, 5: 15894. DOI:10.1038/srep15894.
[72] VALENTINI P, GALIMBERTI A, MEZZASALMA V, et al. DNA barcoding meets nanotechnology: development of a smart universal tool for food authentication[J]. Angewandte Chemie, 2017, 56(28):8094. DOI:10.1002/ange.201702120.
[73] BERTOLINI F, GHIONDA M C, D’ALESSANDRO E, et al. A next generation semiconductor based sequencing approach for theidentification of meat species in DNA mixtures [J]. PLoS ONE,2015, 10(4): e0121701. DOI:10.1371journal.pone.0121701.
Advances in Identification of Sea Cucumber
HU Ranran1,2, XING Ranran1, ZHANG Jiukai1, GE Yiqiang2,3, CHEN Ying1,*
(1. Chinese Academy of Inspection and Quarantine, Beijing 100123, China;2. College of Food Science and Nutritional Engineering, China Agricultural University, Beijing 100083, China;3. China Rural Technology Development Center, Beijing 100045, China)
Abstract: Sea cucumber is broadly favored by consumers due to its high nutritional value and great taste. There are many kinds of sea cucumber products on the market, and the quality and price of various sea cucumber products are different.Nowadays, driven by economic benefits, a number of people mislabel and adulterate high valued sea cucumber products,which has greatly harmed the interests of consumers. Beginning with the current status of adulteration of sea cucumber,this review summarizes the recent progress in the identification of sea cucumber from morphological, physicochemical and molecular biological perspectives. Finally, we discuss future directions in this area of research.
Keywords: sea cucumber; identification technique; morphological identification; physicochemical technique; molecular biology technology
收稿日期:2017-11-30
基金项目:“十三五”国家重点研发计划重点专项 (2016YFD0401104)
第一作者简介:胡冉冉(1994—)(ORCID: 0000-0002-1394-740X),女,硕士研究生,研究方向为食品真伪鉴别。E-mail: huranran61@163.com
*通信作者简介:陈颖(1972—)(ORCID: 0000-0002-8433-3341),女,研究员,博士,研究方向为食品安全。E-mail: chenyingcaiq@163.com
DOI:10.7506/spkx1002-6630-20171130-373
中图分类号:TS207.3
文献标志码:A
文章编号:1002-6630(2019)07-0304-10
引文格式:胡冉冉, 邢冉冉, 张九凯, 等. 海参鉴别技术研究进展[J]. 食品科学, 2019, 40(7): 304-313. DOI:10.7506/spkx1002-6630-20171130-373. http://www.spkx.net.cn
HU Ranran, XING Ranran, ZHANG Jiukai, et al. Advances in identification of sea cucumber[J]. Food Science, 2019, 40(7):304-313. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-20171130-373. http://www.spkx.net.cn


