随着人们的健康意识日益增强,益生菌因其益生功效而获得越来越多的关注。联合国粮农组织和世界卫生组织于2002年将益生菌定义为“一类达到足够数量时可对宿主产生有利作用的活的微生物”。其中植物乳杆菌是应用最为广泛的益生菌之一。临床研究表明,植物乳杆菌对胃肠道疾病(如应激性结肠综合征、炎性肠病、腹泻等)及变应性疾病(如特应性皮炎等)等方面具有积极的治疗效果[1-3]。然而,关于植物乳杆菌基因功能的解析尚显不足,且多集中在异源表达相关蛋白再进行分析。此外菌剂在应用及制备过程中,受到不良环境的胁迫。因此,需要其能够抵御胁迫环境从而保证活性,且能够在保存和运输过程中保持稳定。目前主要通过优化益生菌剂的保护壁材、添加辅剂等工程手段来提升菌株的抗胁迫能力[4-5]。关于如何从菌株自身的生理特性及基因特征角度来增强其抗性能力尚处于起步阶段。
tuf基因编码热不稳定延伸因子(elongation factor thermo unstable,EF-Tu),在细菌中是丰度最高的蛋白之一,参与蛋白质合成最关键的步骤。此外,EF-Tu还具有诸多其他功能,参与细菌其他生理行为[6-7]。研究表明,Francisella novicida的EF-Tu可以促发巨噬细胞的炎症细胞因子[8],然而Leptospira spp.的EF-Tu可以与血纤维蛋白溶原酶及补体因子H结合,从而导致组织浸润和补体失活[9]。在Vibrio parahaemolyticus中EF-Tu被认为是新的毒力因子[10],而在Staphylococcus aureus中EF-Tu因其细胞质或表面定位而表现出多种功能,与菌体对宿主组织的定植、存留和侵袭有关[11]。Gallibacterium的EF-Tu可能参与其生物被膜形成,与致病性有关。在益生菌领域,相关研究较为匮乏[12]。其中Lactobacillus reuteri的EF-Tu可以与猪胃黏液素结合,从而促进细菌黏附于黏膜表面[13]。然而,与诸如大肠杆菌、沙门氏菌、酵母菌等工程菌相比,益生菌的基因工程操作元件相对较少,技术平台相对不成熟,这也限制了益生菌的同源基因工程操作分析及基因重组菌剂的开发。
在此背景下,本研究采用同源超表达及电转化技术,构建植物乳杆菌tuf基因超表达重组菌株,对重组菌株的生长情况、抗胁迫能力、黏附能力及生物被膜形成能力进行分析,并检测tuf基因超表达对其他基因的转录水平有何影响。本研究为进一步研究乳杆菌基因功能,从基因改造角度开发更高效的菌剂提供新的思路和理论支持。
1.1.1 菌种与细胞
LR-1分离自四川发酵泡菜,经16S rDNA测序鉴定为一株植物乳杆菌(Lactobacillus plantarum),命名为植物乳杆菌LR-1;大肠杆菌(Escherichia coli)DH5α购于宝生物工程(大连)有限公司;HT-29细胞,由本实验室保存。
1.1.2 培养基
乳酸菌培养用培养基:MRS培养基,包括蛋白胨10 g/L,酵母提取物5 g/L,柠檬酸二铵2 g/L,葡萄糖20 g/L,MgSO4·7H2O 0.58 g/L,牛肉浸膏10 g/L,K2HPO4 2 g/L,乙酸钠5 g/L,吐温80 1 mL/L,MnSO4·4H2O 0.25 g/L。
大肠杆菌培养用培养基:LB培养基,包括胰蛋白胨10 g/L,酵母提取物5 g/L,NaCl 10 g/L,调节pH值至7.4,121 ℃灭菌15 min后备用。
1.1.3 试剂
R010Q PrimeSTAR HS DNA Polymerase、RR001A Ex Taq、6011 pMD 18-T Vector Cloning Kit 宝生物工程(大连)有限公司;EL0021 T4 DNA连接酶、RNasefree DNase I、RNase抑制剂以及DMEM(Dulbecoo's Modified Eagle Medium) 美国Thermo Scientific公司;DP209普通琼脂糖凝胶DNA回收试剂盒、DP107高纯度质粒小提中量试剂盒 天根生化科技(北京)有限公司;反转录试剂盒TUREscript 1st Strand cDNA Synthesis Kit和TRIpure 北京艾德莱生物科技有限公司;胰酶-0.02%EDTA消化液 上海碧云天生物技术有限公司;磷酸盐缓冲溶液(phosphate buffer saline,PBS) 北京索莱宝科技有限公司。
20%甘氨酸贮液:20 g 甘氨酸,溶于100 mL去离子水,高压灭菌,常温避光保存;MRSS(MRS,0.3 mol/L蔗糖):10.269 g蔗糖溶于100 mL MRS,灭菌待用;MRSSM(MRS,0.3 mol/L蔗糖,0.1 mol/L MgCl2):10.269 g蔗糖和 2 g MgCl2溶于100 mL MRS,灭菌待用;Washing Buffer(0.3 mol/L蔗糖,0.1 mol/L MgCl2):0.269 g蔗糖和2 g MgCl2溶于100 mL去离子水中,灭菌待用;30%聚乙二醇-1500(polyethylene glycol-1500,PEG-1500):15 g PEG-1500水浴加热溶化后,溶于50 mL去离子水中,采用0.22 μm无菌滤器除菌,4 ℃保存。
3K15低温高速离心机 德国Satorious公司;AIRTECH-SW-CJ-2FD超净工作台 苏州安泰空气技术有限公司;Tanon 1600全自动数码凝胶图像分析系统上海天能科技有限公司;KF960聚合酶链式反应(polymerase chain reaction,PCR)仪 杭州晶格科学仪器有限公司;MicroPulser电转化仪/电穿孔仪 美国Bio-Rad公司;FQD-96A实时定量聚合酶链式反应(quantification real-time polymerase chain reaction,qRTPCR)检测系统 杭州博日科技有限公司。
1.3.1 穿梭重组质粒tuf-pMD18T的构建
提取植物乳杆菌LR-1的基因组,以基因组为模板,扩增菌株LR-1的tuf基因。扩增结束后,进行琼脂糖凝胶电泳,切胶回收。将tuf基因胶回收产物与pMD18T载体进行连接,连接体系(10 μL)如下:tuf基因胶回收产物4 μL,pMD18T 1 μL,Solution I 5 μL。连接温度16 ℃,连接时间2 h。连接结束后将连接产物化学转化至大肠杆菌DH5α感受态细胞中,后培养于含有100 μg/mL氨苄青霉素的LB固体平板中,37 ℃培养至肉眼可见单克隆菌落。挑取单克隆菌落,置于含有100 μg/mL氨苄青霉素的LB液体培养基中,37 ℃振荡培养12 h,进行菌液PCR鉴定并筛选阳性单克隆菌落。将筛选的阳性菌株接种至含有100 μg/mL氨苄青霉素的LB液体培养基中,37 ℃振荡培养12 h,采用高纯度小提中量质粒提取试剂盒提取质粒tuf-pMD18T。将提取的质粒进行琼脂糖凝胶电泳检测,检测合格后置于-20 ℃冰箱保存。
1.3.2 过表达重组质粒tuf-pMG76e的构建
将重组质粒tuf-pMD18T进行XbaI/XhoI双酶切,并对tuf基因片段进行切胶回收。同时将质粒pMG76e(由中国农业大学陈尚武教授及李平兰教授友情馈赠)进行XbaI/XhoI双酶切,并对线性化的质粒pMG76e片段进行切胶回收。将二者进行连接,连接体系(10 μL)为:tuf基因片段4 μL,质粒pMG76e 1 μL,Solution I 5 μL。连接温度16 ℃,连接时间2 h。连接结束后将连接产物化学转化至大肠杆菌DH5α感受态细胞中。转化完成后将菌株均匀涂布于含有200 μg/mL红霉素的LB固体平板中,37 ℃培养48 h。挑取单克隆菌落进行培养,进行菌液PCR鉴定,使用引物为76e-F/R,扩增体系和扩增步骤同上所示。之后进行琼脂糖凝胶电泳,筛选阳性克隆。将阳性菌株接种至含有200 μg/mL红霉素的LB液体培养基中进行扩大培养,提取质粒,进行琼脂糖凝胶电泳检测。对检测合格的质粒进行再次双酶切鉴定,检测合格后置于-20 ℃冰箱保存。
1.3.3 感受态细胞的制备
将500 μL 20%甘氨酸贮液加到9.5 mL MRSS中,即为L-ZS9感受态培养基。以1%(体积分数)接种量将植物乳杆菌LR-1接种至以上培养基中,待菌生长至OD600 nm达到0.5。将培养好的菌液至于10 mL离心管中,6 000 r/min、4 ℃离心8 min,收集菌体;去上清液,加入2 mL Washing Buffer洗涤沉淀,6 000 r/min、4 ℃离心8 min,收集菌体;重复此步骤;去上清液,加入2 mL 30% PEG-1500重悬后,6 000 r/min、4 ℃离心10 min;去上清液,加入200 μL 30% PEG-1500重悬,每管40 μL,置于冰上待用。
1.3.4 tuf同源过表达重组菌株的构建及鉴定
采用电转化将构建好的含有tuf基因的重组质粒导入植物乳杆菌LR-1感受态细胞,构建EF-Tu同源过表达重组菌株。具体方法如下:将4 μL LR-1感受态细胞转移到2 mm电击杯中,点击检测是否爆杯,如果正常,作为对照组;分别将空载体pMG76e质粒和重组质粒tuf-pMG76e DNA(2 μL)与40 μL感受态细胞的混合后置于冰上(加质粒量不要超过总体积的1/20)转移到2 mm电击杯中,置于冰上5 min,并确定没气泡;设定点击程序1.5 kV,25 μL,400 Ω;进行点击后,冰上静置5 min;加入1 mL MRSSM复苏培养基,转移到1.5 mL离心管中,37 ℃培养2 h;将复苏的菌液7 000 r/min、常温离心2 min,去除850 μL上清液后,涂布于含3 μg/mL红霉素的MRS平板,37 ℃培养 36~48 h。
之后通过pMG76e鉴定引物进行PCR扩增及琼脂糖凝胶电泳对转化子进行初步鉴定。分别挑取转化子单克隆于含有3 μg/mL红霉素的MRS液体培养基中,静置培养16~18 h后,吸取100 μL培养菌液,10 000 r/min离心2 min,去除上清液,加入100 μL无菌水重悬菌泥,离心去除上清液,重复该步骤一次,之后用20 μL无菌水重悬菌泥,此为PCR扩增体系模板,置于-20 ℃备用。后进行PCR及琼脂糖凝胶电泳检测,空载体导入菌株为阴性对照,扩增引物序列为76F:5’-TTCGGTCCTCGGGATATG-3’;76R:5’-CTGTCTTGGCCGCTTCAA-3’。
1.3.5 tuf基因转录水平测定
通过qRT-PCR对tuf过表达重组菌株的tuf基因转录水平进行测定,以进一步鉴定tuf过表达重组菌株的构建是否成功。将植物乳杆菌LR-1野生株、空载体pMG76e导入菌株和tuf过表达重组菌株接种于MRS培养基中,至37 ℃静置培养8 h。培养完成12 000×g离心收集菌体,液氮研磨,加TRIpure,置于-80 ℃冻存。参照北京艾德莱生物科技有限公司的TRIpure提取步骤提取总RNA。cDNA的合成参照该公司TUREscript 1st Strand cDNA Synthesis Kit说明书进行,方法如下:总RNA 50 ng~5 μg,随机引物1 μL,5×RT Reaction Mix 4 μL,TUREscript H-RTase 1 μL,之后用RNase free H2O定容至20 μL。25 ℃孵育10 min,之后42 ℃孵育30~50 min,65 ℃加热15 min以失活TUREscript H-RTase。DNaseI处理合成的cDNA后,置于-80 ℃待用。以16S rRNA为内参基因,采用Primer 3 Input(version 0.4.0)设计引物,用于进行qRT-PCR。引物序列如下:tuf-F:5’-GACCGCTGCAATCACTAAGG-3’;tuf-R:5’-GAGCGGCACCAGTAATCATG-3’。
qRT-PCR体系(20 μL):1 μL模板cDNA,10 μL 2×Sybgreen,上游引物和下游引物各1 μL(10 μmol/L),以及7 μL无RNase水。扩增程序为:95 ℃、10 min;95 ℃、15 s,60 ℃、30 s,共40 个循环。采用Livak法进行数据分析。
1.3.6 生长曲线测定
将植物乳杆菌LR-1野生株、空载体pMG76e导入菌株和tuf过表达重组菌株以1%接种于MRS培养基中,37 ℃静置培养36 h,分别在3、6、9、12、15、18、21、24、27、30、33、36 h收集培养液,测定OD600 nm,绘制生长曲线。
1.3.7 耐热、耐酸、耐胆盐能力检测
分别将植物乳杆菌LR-1野生株、空载体pMG76e导入菌株和tuf过表达重组菌株进行活化。将收集到的菌体置于80 ℃水浴锅中,处理3 min,进行耐热实验。将收集到的菌体分别重悬于pH值为2.8的MRS培养基中,处理1 h,进行耐酸实验。将收集到的菌体分别重悬于含胆盐0.2 g/100 mL的MRS培养基中,处理0.5 h,进行耐胆盐实验。处理结束后通过平板活菌计数法计算活菌数。实验每组设3个平行,且进行3次生物学重复。分别确定以上菌株的耐热、耐酸和耐胆盐能力。存活率计算如式(1)所示:
1.3.8 黏附能力检测
将植物乳杆菌LR-1野生株、空载体pMG76e导入菌株和tuf过表达重组菌株接种于MRS培养基中进行活化,5 000 r/min离心10 min收集菌体,PBS洗涤菌体,并重悬至终浓度1×108 CFU/mL。
将HT-29细胞接种于24 孔细胞培养板中,待其长满单层后(每孔约含2.5×105 个细胞),每孔接种100 μL上述收集的细菌悬液,轻微晃匀后置于37 ℃培养1.5 h,弃培养液,PBS洗涤5 次,加100 μL的0.1%胰酶-0.02% EDTA室温处理10 min,消化细胞,再加900 μL DMEM,并用巴氏吸管吹打,取100 μL混合液进行10 倍稀释,各稀释度取100 μL接种于MRS平板,37 ℃培养,记录菌落数,实验每组设3 个平行,且进行3 次生物学重复。黏附率计算如式(2)所示:
1.3.9 生物被膜量检测
生物被膜形成能力检测采用结晶紫染色法。将活化的植物乳杆菌LR-1野生株、空载体pMG76e导入菌株和tuf过表达重组菌株接种于MRS培养基中,将培养液加至96 孔细胞培养板中,每孔200 μL,37 ℃静置培养36 h。培养结束后用PBS轻柔冲洗3 次,去除悬浮菌体,室温晾干。每孔加0.1%结晶紫溶液,室温染色30 min,无菌水轻柔冲洗至洗液无色为止,室温晾干。加100 μL无水乙醇充分溶解结晶紫,检测OD595 nm值。
1.3.10 其他基因转录水平的测定
按照1.3.5节提取LR-1野生株、空载体pMG76e导入菌株和tuf过表达重组菌株的总RNA,通过qRT-PCR对相关基因转录水平进行测定。根据前期研究选定与乳杆菌抗胁迫能力、黏附能力及生物被膜形成能力相关的目标基因,分别为果糖-二磷酸醛缩酶编码基因fba、甘油醛-3-磷酸脱氢酶编码基因gap、磷酸甘油酸变位酶编码基因pgm、核糖激酶编码基因rib和S-核糖高半胱氨酸酶编码基因luxS。
以16S rRNA为内参基因,采用Primer 3 Input(version 0.4.0)设计引物,用于进行qRT-PCR。引物名称及序列如表1所示。1.4 数据分析
表 1 荧光定量PCR引物设计
Table 1 Primers used for real-time PCR
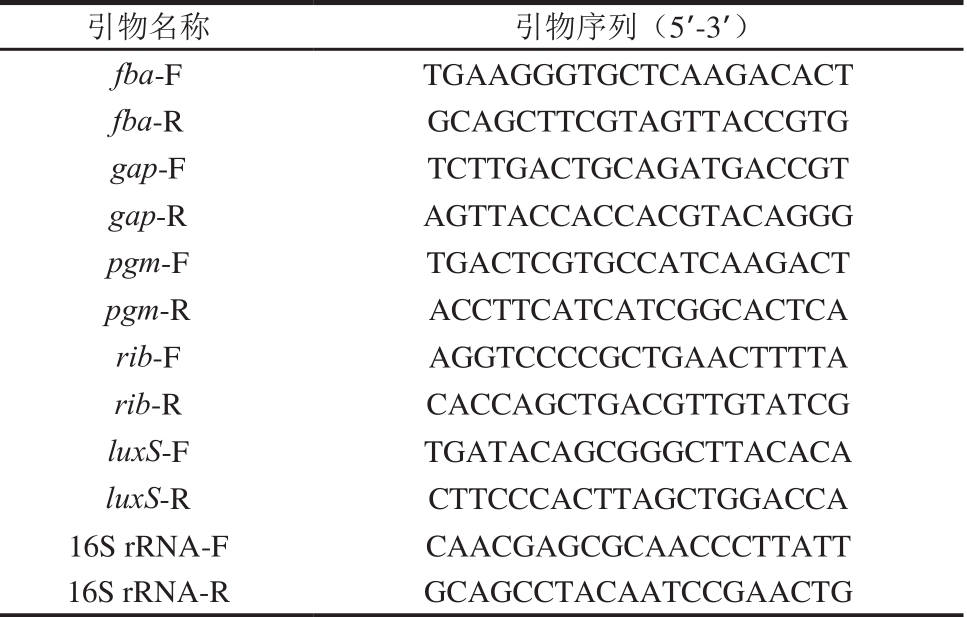
引物名称 引物序列(5’-3’)fba-F TGAAGGGTGCTCAAGACACT fba-R GCAGCTTCGTAGTTACCGTG gap-F TCTTGACTGCAGATGACCGT gap-R AGTTACCACCACGTACAGGG pgm-F TGACTCGTGCCATCAAGACT pgm-R ACCTTCATCATCGGCACTCA rib-F AGGTCCCCGCTGAACTTTTA rib-R CACCAGCTGACGTTGTATCG luxS-F TGATACAGCGGGCTTACACA luxS-R CTTCCCACTTAGCTGGACCA 16S rRNA-F CAACGAGCGCAACCCTTATT 16S rRNA-R GCAGCCTACAATCCGAACTG
所有测试重复进行平行实验3次,用GraphPad Prism 5.0软件进行统计分析(ANOVA)及作图,P<0.05,差异显著。
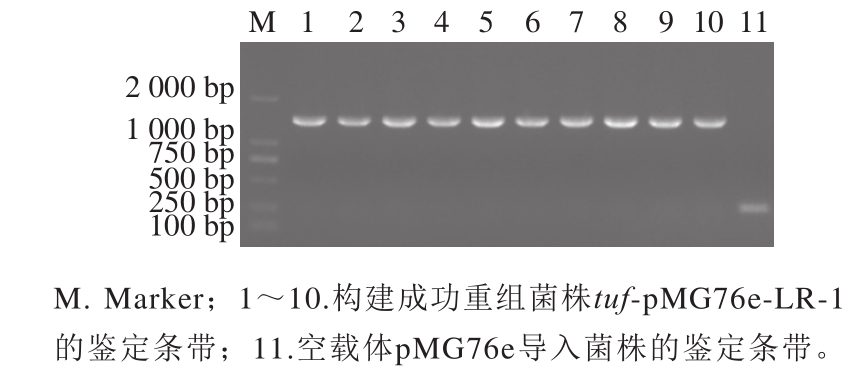
图 1 重组菌株tuf- pMG76e-LR-1的PCR及琼脂糖凝胶电泳鉴定
Fig. 1 Identification of recombinant strain tuf-pMG76e-LR-1 by PCR and agarsose gel electrophoresis
如图1所示,11号条带理论全长231 bp,tuf基因全长1 188 bp,1~10号条带理论全长1 419 bp,与Marker对比可见,实际条带大小均位于合理位置。PCR鉴定结果初步表明,重组菌株tuf-pMG76e-LR-1构建成功。
重组菌株tuf-pMG76e-LR-1的tuf基因转录水平检测结果如图2所示,空载体导入菌株pMG76e-LR-1与野生型菌株LR-1相比,tuf基因的转录水平无明显差异,重组菌株tuf-pMG76e-LR-1的tuf基因转录水平平均上约调105 倍,表明菌株tuf-pMG76e-LR-1的tuf基因转录水平显著上调。
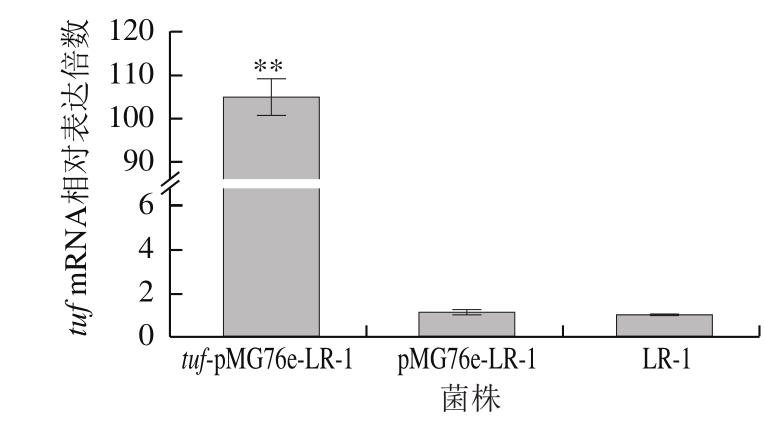
图 2 重组菌株tuf- pMG76e-LR-1的tuf基因转录水平
Fig. 2 Transcription of tuf in recombinant strain tuf-pMG76e-LR-1

图 3 重组菌株tuf- pMG76e-LR-1的生长曲线
Fig. 3 Growth curve of recombinant strain tuf-pMG76e-LR-1
由图3可知,空载体导入菌株pMG76e-LR-1与野生型菌株LR-1的生长情况较为一致,表明空载体pMG76e的导入不影响菌株LR-1的生长。同时,重组菌株tuf-pMG76e-LR-1的生长曲线表明,其早期的生长情况稍慢于空载体导入菌株pMG76e-LR-1与野生型菌株LR-1,但最终的菌悬液浓度与其余两者一致。结果表明,tuf基因超表达在早期对菌株LR-1的生长有轻微抑制,但不影响最终菌株LR-1的生长情况。
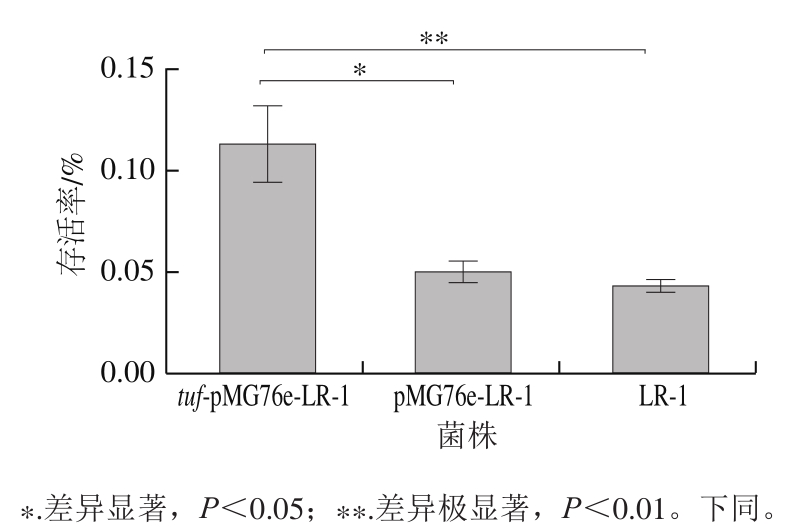
图 4 重组菌株tuf- pMG76e-LR-1的耐热能力分析
Fig. 4 Heat resistance of recombinant strain tuf-pMG76e-LR-1
通过对菌株tuf-pMG76e-LR-1、pMG76e-LR-1和LR-1的耐热能力进行检测,结果如图4所示,与野生型LR-1菌株相比,经热处理后,重组菌株tuf-pMG76e-LR-1的存活率上调至约2.75 倍。该结果说明tuf基因的超表达可增强菌株LR-1的耐热能力。此外,tuf基因超表达对菌株LR-1的耐酸能力和耐胆盐能力没有显著影响(本文中未显示该结果)。
乳酸菌在开发为发酵菌剂或益生菌剂的过程中常常要经历各种胁迫环境,如喷雾干燥法的高温环境[14-15]。有研究表明tuf基因编码的EF-Tu为耐热延伸因子,对热极其稳定,80 ℃、5 min处理原活性仅丢失50%[16]。研究表明该蛋白参与哺乳动物细胞从转录到翻译热应激的全部过程[17]。对tuf基因表达情况对菌株耐热性能的影响进行研究,结果表明tuf基因过表达会显著增强植物乳杆菌LR-1的耐热能力,与EF-Tu在其他菌株的相关研究结果一致。

图 5 重组菌株tuf- pMG76e-LR-1的黏附能力分析
Fig. 5 Adhesion ability of recombinant strain tuf-pMG76e-LR-1
由图5可知,菌株LR-1对HT-29细胞具有一定的黏附能力,且pMG76空载体的导入会对其黏附能力起抑制作用,而tuf-pMG76e重组质粒的导入会增强菌株的黏附能力,使黏附率平均上调至约1.6 倍。结果表明,tuf基因的超表达可以增强菌株LR-1的黏附性能。
有研究表明EF-Tu是一个黏附相关因子,Paracoccidioides brasiliensis的延伸因子EF-Tu与其对宿主的黏附和侵袭相关[18]。最新的研究结果发现该蛋白可作为Mycoplasma hyopneumoniae表面的黏附素,通过与纤粘蛋白结合从而发挥多种作用[19]。对于Helicobacter pylori来说,EF-Tu在致病机理中扮演者潜在黏附因子的重要角色[20]乳杆菌的相关研究发现,Lactobacillus的EF-Tu在菌株黏附时发挥重要作用,它还参与调控其对肠道病原菌的抑制作用[21]。Lactobacillus acidophilus ATCC 4356的EF-Tu也是一个黏附相关因子[22]。菌株L. plantarum 423的EF-Tu参与调控其对Caco-2细胞的黏附作用[23]。最新的研究结果发现该蛋白可作为Mycoplasma hyopneumoniae表面的黏附素,通过与纤粘蛋白结合从而发挥多种作用[19]。本研究结果与以上研究结果一致,且通过构建同源重组菌株并直接检测其黏附能力,表明tuf基因的表达情况与植物乳杆菌LR-1的黏附性能呈正相关,且超表达重组菌株tuf-pMG76e-LR-1自身的黏附能力增强。这提示研究人员益生菌tuf基因超表达重组菌株可能具有更强的体内黏附定植能力,对于其更好的益生功效具有重要价值,具体研究需要进一步动物实验进行验证。此外,空载体pMG76e导入菌株的黏附能力较野生菌株LR-1有所下降,可能是由于感受态制备及电转化过程对菌体细胞本身黏附性能造成影响,或者空载体在菌体中复制对菌体细胞黏附性能产生干扰,具体原因尚不明确,有待进一步研究。

图 6 重组菌株tuf- pMG76e-LR-1的生物被膜形成能力分析
Fig. 6 Biofilm formation of recombinant strain tuf-pMG76e-LR-1
如图6所示,菌株LR-1具有较强的被膜形成能力,与野生型菌株的被膜形成量相比,空载体导入菌株pMG76e-LR-1无显著差异,而重组菌株tuf-pMG76e-LR-1的被膜形成量增多,且在统计学意义上具有极显著差异。结合2.2节研究结果,表明菌株培养36 h后,在tuf基因超表达不会影响菌株最终生长量的情况下,会显著增强菌株LR-1的生物被膜形成能力。
益生菌的生物被膜研究处于起步阶段,前期研究曾对类植物乳杆菌L-ZS9的生物被膜态生理行为进行过分析,发现被膜态的菌体具有更强的抗胁迫能力及黏附能力[24-25],因此选择被膜形成作为一个重要检测指标。目前关于称延伸因子EF-Tu调控生物被膜的形成的研究较为少见,Gallibacterium的EF-Tu具有淀粉样性质,存在于生物被膜中[12]。前期研究曾发现类植物乳杆菌L-ZS9的EF-Tu与菌株的生物被膜形成有关[26]。本研究与之前研究结果一致,植物乳杆菌LR-1的tuf基因表达量增高,可以增强菌株的被膜形成能力。其具体作用机制尚不明确,结合2.5节研究结果进行分析,这可能是由于EF-Tu与黏附能力有关,且EF-Tu有可能是乳杆菌的一种黏附因子,其表达量增高可以增强菌株的固附着能力。
基于前期研究结果[24-26],本研究选择与乳杆菌抗胁迫能力、黏附能力及生物被膜形成能力相关的基因fba、pgm、gap、rib和luxS作为检测基因,以空载体导入菌株pMG76e-LR-1为对照菌株,对重组菌株tuf-pMG76e-LR-1以上基因的转录水平进行检测。如图7所示,重组菌株tuf-pMG76e-LR-1的基因fba、pgm、gap、rib和luxS转录水平均上调表达,其中luxS基因上调表达约47 倍,gap基因上调表达约48 倍,fba基因上调表达约56 倍,pgm基因上调表达约80 倍,rib基因上调表达约305 倍,上调倍数最为显著。

图 7 重组菌株tuf- pMG76e-LR-1和pMG76e-LR-1的基因转录水平比较
Fig. 7 Comparison of genes transcription of recombinant strain tuf-pMG76e-LR-1 and pMG76e-LR-1
糖酵解途径作为大多数生物所共有的基本代谢途径,在有氧和无氧条件下均能进行,且是糖代谢和脂类代谢的连接点,因此该代谢途径的改变对微生物生理行为具有重要影响。本研究中pgm基因编码磷酸甘油酸变位酶,fba基因编码果糖-二磷酸醛缩酶,gap基因编码甘油醛-3-磷酸脱氢酶,以上3 个基因参与微生物的糖酵解途径。前期结果表明乳杆菌中的pgm、fba和gap基因表达情况与生物被膜形成呈正相关[26]。也有研究发现菌株Saccharomyces cerevisiae的糖酵解代谢途径会影响其生物被膜形成[27],菌株Listeria monocytogenes的被膜状态中糖酵解途径相关蛋白发生上调[28],此外,生物被膜形成能力与黏附能力密切相关[29-30]。结合本研究结果可推测,tuf基因的超表达会诱导三者表达量的上调,说明tuf基因可能通过代表菌株LR-1的糖酵解效率来调控生理行为。
luxS基因编码S-核糖高半胱氨酸酶,是细菌II型群体感应系统信号分子的关键合成基因,该基因表达情况的改变常会影响细菌诸多生理行为的变化[31-33]。关于乳杆菌luxS的相关研究相较病原菌尚显不足。目前研究表明L. plantarum KLDS1.0391中luxS与菌株胁迫耐受和黏附能力有关[31]。信号分子AI-2对L. acidophilus黏附能力也有重要影响[34]。LuxS代谢对于Lactobacillus rhamnosus GG的胃肠道耐受能力非常关键[35]。luxS基因的缺失会影响Lactobacillus reuteri 100-23的一些其他生理行为[36]。Lactobacillus spp.的酸应激会诱发AI-2信号通路[37]。以上研究均表明luxS基因参与乳杆菌的抗胁迫、黏附以及其他生理行为。此外,Lactobacillus paraplantarum L-ZS9的luxS基因超表达对会显著增强菌株的生物被膜形成能力[26]。基于以上研究,本研究结果表明菌株LR-1的tuf超表达菌株相关生理行为的改变可能是通过luxS表达水平上调来调控的。rib基因编码核糖激酶,核糖激酶参与核糖代谢,与物质循环及遗传信息的合成有关,也是菌株初级代谢的重要调控基因。此外,前期研究也发现rib基因与菌株的生物被膜形成有关[26]。因此,选择以上5 个基因作为检测对象,发现tuf基因的上调会显著增强以上5 个基因的表达,以上研究与本研究2.4、2.5节研究结果结合分析,表明tuf基因有可能通过上调以上基因来增强菌株LR-1的抗胁迫能力、被膜形成能力及黏附能力。
本研究通过电转化构建植物乳杆菌LR-1的tuf基因同源超表达重组菌株,并对其生长能力、抗胁迫能力、黏附能力及被膜形成能力进行分析,结果表明tuf基因超表达对菌株LR-1的生长情况无非常的显著影响,但是可以增强其耐热性能、黏附能力及生物被膜形成能力。进一步通过qRT-PCR对糖酵解途径参与基因、II型群感关键基因及核糖激酶编码基因fba、pgm、gap、luxS和rib的转录水平进行检测,发现tuf基因超表达可诱导以上基因上调表达,为tuf基因超表达调控植物乳杆菌LR-1生理行为的分子机制提供参考。本研究为乳杆菌的基因功能分析及挖掘具有价值,同时为基因工程菌株在发酵菌剂或益生菌剂的开发应用提供了理论支持。
[1] BA H V, SEO H W, SEONG P N, et al. Lactobacillus plantarum(KACC 92189) as a potential probiotic starter culture for quality improvement of fermented sausages[J]. Korean Journal for Food Science of Animal Resources, 2018, 38(1): 189-202. DOI:10.5851/kosfa.2018.38.1.189.
[2] ATHIYYAH A F, DARMA A, RANUH R, et al. Lactobacillus plantarum IS-10506 activates intestinal stem cells in a rodent model[J]. Beneficial Microbes, 2018, 9(5): 755-760. DOI:10.3920/BM2017.0118.
[3] KIM K, LEE G, THANH H D, et al. Exopolysaccharide from Lactobacillus plantarum LRCC5310 offers protection against rotavirus-induced diarrhea and regulates inflammatory response[J].Journal of Dairy Science, 2018, 101(7): 5702-5712. DOI:10.3168/jds.2017-14151.
[4] MOKHTARI S, JAFARI S M, KHOMEIRI M, et al. The cell wall compound of Saccharomyces cerevisiae as a novel wall material for encapsulation of probiotics[J]. Food Research International, 2017, 96:19-26. DOI:10.1016/j.foodres.2017.03.014.
[5] JIANG Y, ZHENG Z, ZHANG T, et al. Microencapsulation of Lactobacillus acidophilus NCFM using polymerized whey proteins as wall material[J]. International Journal of Food Sciences and Nutrition,2016, 67(6): 670-677. DOI:10.1080/09637486.2016.1194810.
[6] WIDJAJA M, HARVEY K L, HAGEMANN L, et al. Elongation factor Tu is a multifunctional and processed moonlighting protein[J].Scientific Reports, 2017, 7(1): 11227. DOI:10.1038/s41598-017-10644-z.
[7] PENG Z, VOGEL R F, EHRMANN M A, et al. Identification and characterization of adhesion proteins in Lactobacilli targeting actin as receptor[J]. Molecular and Cellular Probes, 2018, 37: 60-63.DOI:10.1016/j.mcp.2017.08.002.
[8] SHARMA J, MISHRA B B, LI Q, et al. TLR4-dependent activation of inflammatory cytokine response in macrophages by Francisella elongation factor Tu[J]. Cellular Immunology, 2011, 269(2): 69-73.DOI:10.1016/j.cellimm.2011.03.023.
[9] WOLFF D G, CASTIBLANCO-VALENCIA M M, ABE C M, et al.Interaction of Leptospira elongation factor Tu with plasminogen and complement factor H: a metabolic leptospiral protein with moonlighting activities[J]. PLoS ONE, 2013, 8(11): e81818.DOI:10.1371/journal.pone.0081818.
[10] HE Y, WANG H, CHEN L. Comparative secretomics reveals novel virulence-associated factors of Vibrio parahaemolyticus[J]. Frontiers in Microbiology, 2015, 6: 707. DOI:10.3389/fmicb.2015.00707.
[11] PAPA R, ARTINI M, CELLINI A, et al. A new anti-infective strategy to reduce the spreading of antibiotic resistance by the action on adhesionmediated virulence factors in Staphylococcus aureus[J]. Microbial Pathogenesis, 2013, 63: 44-53. DOI:10.1016/j.micpath.2013.05.003.
[12] LOPEZ-OCHOA J, MONTES-GARCIA J F, VAZQUEZ C, et al.Gallibacterium elongation factor-Tu possesses amyloid-like protein characteristics, participates in cell adhesion, and is present in biofilms[J]. Journal of Microbiology, 2017, 55(9): 745-752.DOI:10.1007/s12275-017-7077-0.
[13] NISHIYAMA K, OCHIAI A, TSUBOKAWA D, et al. Identification and characterization of sulfated carbohydrate-binding protein from Lactobacillus reuteri[J]. PLoS ONE, 2013, 8(12): e83703.DOI:10.1371/journal.pone.0083703.
[14] DIANAWATI D, LIM S F, OOI Y, et al. Effect of type of proteinbased microcapsules and storage at various ambient temperatures on the survival and heat tolerance of spray dried Lactobacillus acidophilus[J]. Journal of Food Science, 2017, 82(9): 2134-2141.DOI:10.1111/1750-3841.13820.
[15] LAVARI L, BURNS P, PAEZ R, et al. Study of the effects of spray drying in whey-starch on the probiotic capacity of Lactobacillus rhamnosus 64 in the gut of mice[J]. Journal of Applied Microbiology,2017, 123(4): 992-1002. DOI:10.1111/jam.13567.
[16] 乔传令. 耐热蛋白质延长因子(EF-TU)的分子和功能的性质(英文)[J].实验生物学报, 1992(2): 165-172.
[17] VERA M, PANI B, GRIFFITHS L A, et al. The translation elongation of eEF1A1 couples transcription to translation during heat shock response[J]. Elife, 2014, 3: e03164. DOI:10.7554/eLife.03164.
[18] MARCOS C M, DE OLIVEIRA H C, DA S J, et al. Identification and characterisation of elongation factor Tu, a novel protein involved in Paracoccidioides brasiliensis-host interaction[J]. FEMS Yeast Research, 2016, 16(7). DOI:10.1093/femsyr/fow079.
[19] YU Y, WANG H, WANG J, et al. Elongation factor thermo unstable(EF-Tu) moonlights as an adhesin on the surface of Mycoplasma hyopneumoniae by binding to fibronectin[J]. Frontiers in Microbiology,2018, 9: 974. DOI:10.3389/fmicb.2018.00974.
[20] CHIU K H, WANG L H, TSAI T T, et al. Secretomic analysis of hostpathogen interactions reveals that elongation factor-Tu is a potential adherence factor of Helicobacter pylori during pathogenesis[J].Journal of Proteome Research, 2017, 16(1): 264-273. DOI:10.1021/acs.jproteome.6b00584.
[21] DHANANI A S, BAGCHI T. The expression of adhesin EF-Tu in response to mucin and its role in Lactobacillus adhesion and competitive inhibition of enteropathogens to mucin[J]. Journal of Applied Microbiology, 2013, 115(2): 546-554. DOI:10.1111/jam.12249.
[22] RAMIAH K, VAN REENEN C A, DICKS L M. Expression of the mucus adhesion gene mub, surface layer protein slp and adhesionlike factor EF-TU of Lactobacillus acidophilus ATCC 4356 under digestive stress conditions, as monitored with Real-Time PCR[J].Probiotics and Antimicrobial Proteins, 2009, 1(1): 91. DOI:10.1007/s12602-009-9009-8.
[23] RAMIAH K, VAN REENEN C A, DICKS L M. Surface-bound proteins of Lactobacillus plantarum 423 that contribute to adhesion of Caco-2 cells and their role in competitive exclusion and displacement of Clostridium sporogenes and Enterococcus faecalis[J].Research in Microbiology, 2008, 159(6): 470-475. DOI:10.1016/j.resmic.2008.06.002.
[24] 刘蕾, 武瑞赟, 李军, 等. 基于AI-2介导的类植物乳杆菌生物被膜态与浮游态抗胁迫能力比较与分析[J]. 食品科学, 2017, 38(22): 41-47.DOI:10.7506/spkx1002-6630-201722007.
[25] 刘蕾, 武瑞赟, 李军, 等. 类植物乳杆菌L-ZS9生物被膜态的黏附能力及形成影响因素[J]. 食品科学, 2016, 37(15): 136-143.DOI:10.7506/spkx1002-6630-201615023.
[26] LIU L, WU R, ZHANG J, et al. D-Ribose interferes with quorum sensing to inhibit biofilm formation of Lactobacillus paraplantarum L-ZS9[J]. Frontiers in Microbiology, 2017, 8: 1860. DOI:10.3389/fmicb.2017.01860.
[27] LI Z, CHEN Y, LIU D, et al. Involvement of glycolysis/gluconeogenesis and signaling regulatory pathways in Saccharomyces cerevisiae biofilms during fermentation[J]. Frontiers in Microbiology,2015, 6: 139. DOI:10.3389/fmicb.2015.00139.
[28] ZHOU Q, FENG X, ZHANG Q, et al. Carbon catabolite control is important for Listeria monocytogenes biofilm formation in response to nutrient availability[J]. Current Opinion in Microbiology, 2012, 65(1):35-43. DOI:10.1007/s00284-012-0125-4.
[29] YARAWSKY A E, ENGLISH L R, WHITTEN S T, et al. The proline/glycine-rich region of the biofilm adhesion protein aap forms an extended stalk that resists compaction[J]. Journal of Molecular Biology, 2017, 429(2): 261-279. DOI:10.1016/j.jmb.2016.11.017.
[30] BERLUTTI F, MOREA C, BATTISTONI A, et al. Iron availability influences aggregation, biofilm, adhesion and invasion of Pseudomonas aeruginosa and Burkholderia cenocepacia[J]. International Journal of Immunopathology and Pharmacology, 2005, 18(4): 661-670.DOI:10.1177/039463200501800407.
[31] JIA F F, ZHENG H Q, SUN S R, et al. Role of luxS in stress tolerance and adhesion ability in Lactobacillus plantarum KLDS1.0391[J].Biomed Research International, 2018, 2018: 4506829.DOI:10.1155/2018/4506829.
[32] JU X, LI J, ZHU M, et al. Effect of the luxS gene on biofilm formation and antibiotic resistance by Salmonella serovar Dublin[J].Food Research International, 2018, 107: 385-393. DOI:10.1016/j.foodres.2018.02.039.
[33] JIA F F, PANG X H, ZHU D Q, et al. Role of the luxS gene in bacteriocin biosynthesis by Lactobacillus plantarum KLDS1.0391:a proteomic analysis[J]. Scientific Reports, 2017, 7(1): 13871.DOI:10.1038/s41598-017-13231-4.
[34] BUCK B L, AZCARATE-PERIL M A, KLAENHAMMER T R.Role of autoinducer-2 on the adhesion ability of Lactobacillus acidophilus[J]. Journal of Applied Microbiology, 2009, 107(1):269-279. DOI:10.1111/j.1365-2672.2009.04204.x.
[35] LEBEER S, CLAES I J, VERHOEVEN T L, et al. Impact of luxS and suppressor mutations on the gastrointestinal transit of Lactobacillus rhamnosus GG[J]. Applied and Environmental Microbiology, 2008,74(15): 4711-4718. DOI:10.1128/AEM.00133-08.
[36] TANNOCK G W, GHAZALLY S, WALTER J, et al. Ecological behavior of Lactobacillus reuteri 100-23 is affected by mutation of luxS gene[J]. Applied and Environmental Microbiology, 2005, 71(12):8419-8425. DOI:10.1128/AEM.71.12.8419-8425.2005.
[37] MOSLEHI-JENABIAN S, GORI K, JESPERSEN L. AI-2 signalling is induced by acidic shock in probiotic strains of Lactobacillus spp.[J].International Journal of Food Microbiology, 2009, 135(3): 295-302.DOI:10.1016/j.ijfoodmicro.2009.08.011.
Effect of tuf Homogenous Overexpression on Physiological Behaviors of Lactobacillus plantarum LR-1