1 株T1样噬菌体vB_EcoS_IME18的分离鉴定及其受体分析
李 萍1,林 洪2,童贻刚2,*,王静雪1,*
(1.中国海洋大学食品科学与工程学院,山东 青岛 266003;2.军事科学院军事医学研究所,微生物流行病研究所病原微生物安全国家重点实验室,北京 100071)
摘 要:以大肠杆菌BL21为指示菌分离得到1 株噬菌体vB_EcoS_IME18(IME18),通过电镜观察噬菌体形态,并测定其最佳感染复数、一步生长曲线、裂解谱和体外杀菌效果;使用Illumina MiSeq测序仪完成噬菌体全基因组测序,利用比较基因组学分析噬菌体基因组特征;通过筛选噬菌体抗性株分析其耐受原因,利用受体基因沉默突变实验、噬菌体斑点实验和噬菌体吸附实验验证噬菌体受体。结果表明,噬菌体IME18具有二十面体头部和非收缩性尾部,属于长尾噬菌体科,Tunavirinae亚科,T1类噬菌体;噬菌体IME18的最佳感染复数为0.01,潜伏期为5 min,裂解期50 min,爆发量高达223 PFU/cell;在体外,噬菌体IME18能在90 min内有效杀死处于对数期的BL21;噬菌体IME18基因组全长50 354 bp,G+C含量为45.6%;蛋白FhuA、TonB在噬菌体IME18的吸附和侵染过程中发挥重要作用。
关键词:大肠杆菌;噬菌体;鉴定;噬菌体受体
应用基因工程技术生产重组蛋白被广泛应用于生物、医药、食品等领域。在重组蛋白表达系统中,大肠杆菌BL21由于其表达系统操作简单、价格廉价、目的蛋白产量大而被广泛用作受体菌[1]。值得注意的是,应用大肠杆菌BL21进行高密度发酵的生物技术实验室和工厂车间非常容易受到噬菌体污染。实验室或工厂避免噬菌体污染的预防措施主要包括控制感染源、菌株轮换、传统的基因工程策略和基于CRISPR-Cas系统的噬菌体防治策略[2-4]。预防和控制噬菌体污染策略的开发需要更多噬菌体吸附过程及噬菌体受体信息的支持。
噬菌体是一类能够侵染细菌的病毒,烈性噬菌体的生活周期通常包括吸附、注入、复制、转录和翻译、装配和释放。吸附是噬菌体感染的第一个步骤,噬菌体颗粒首先与宿主细胞表面碰撞,然后特异性识别其受体并紧密结合细胞[5]。噬菌体受体可以是蛋白质、多糖、脂多糖、菌毛、鞭毛和荚膜,在革兰氏阴性菌中,外膜蛋白和脂多糖或二者的组合经常作为噬菌体受体[6]。噬菌体和细菌存在共同进化的关系[7],为了抵抗噬菌体的袭击,细菌经常通过改变噬菌体受体来阻止噬菌体吸附,例如受体的表达下调、突变或丢失和空间阻遏[8]。在已有研究中,通过筛选噬菌体抗性突变分析噬菌体受体的方法已十分成熟[9-13]。本研究将利用筛选噬菌体抗性突变株分析噬菌体受体,为噬菌体-宿主互作研究和噬菌体污染防治提供数据支持。
1 材料与方法
1.1 材料与试剂
本实验所用大肠杆菌BL21购自北京全式金生物公司,保存于本实验室;大肠杆菌噬菌体 vB_EcoS_IME18(以下简称IME18)分离于本实验室污水;质粒pKDsgack、pCas9cr4和pKDsg-p15保存于本实验室;所使用的引物均由北京诺塞基因公司合成。
阿奇霉素(Spec)、氨苄青霉素(Amp)、L-阿拉伯糖、脱水四环素 上海生工公司;pEASYUniSeamLess Cloning and Assembly Kit、2×Trans Taq®高保真(HiFi)聚合酶链式反应(polymerase chain reaction,PCR)SuperMix、Trans 5α感受态细胞 北京全式金生物公司;Gel Extraction Kit、Plasmid Mini Kit宝生物工程(大连)有限公司;Q5®HotStart High-Fidelity 2×Master Mix 美国NEB公司;Pure PCR Template Preparation Kit 德国罗氏公司。
1.2 仪器与设备
Power Wave XS2酶标仪 美国BioTek公司;MiSeq测序仪 美国NEB公司;Bioruptor UCD-200TS超声仪美国赛默飞公司;Micro Pulser电转仪 美国Bio-Rad公司;Tannon-4200SF凝胶成像仪 上海天能科技有限公司;DNA电泳仪及相关设备 北京沃德生物医学仪器公司;9700型PCR仪 美国Applied Biosystems公司。
1.3 方法
1.3.1 噬菌体的分离与纯化
取实验室未经处理的污水,12 000 r/min离心10 min,上清液用0.22 μm的滤器过滤。将污水滤液200 μL以及对数期的BL21菌液4 mL加入2 mL 3×LB液体培养基中,37 ℃、220 r/min培养6 h。培养液用0.22 μm微孔滤膜过滤,所得滤液即为噬菌体原液。参考文献[14]所述的双层平板法进行噬菌体纯化。
1.3.2 噬菌体的电镜观察
采用磷钨酸负染法,通过透射电子显微镜观察噬菌体形态。将铜网浸于纯化的噬菌体保存液内5 min,取出后用滤纸吸干多余液体,此后将其浸于2%磷钨酸染液中染色1 min,室温干燥5 min,使用透射电镜观察噬菌体形态。
1.3.3 噬菌体基因组的提取
参考文献[15]所述蛋白酶K/SDS的方法进行噬菌体基因组的提取,将DNA于-20 ℃保存。
1.3.4 噬菌体的全基因组测序及分析
使用Illumina公司的MiSeq测序平台进行噬菌体IME18的DNA文库构建和全基因组测序。选取高质量的下机数据拼接成全基因组序列。使用NCBI的BLASTn(http://blast.ncbi.nlm.nlm.nih.gov/)进行序列相似性比对;通过在线RAST(http://rast.nmpdr.org)进行全基因组快速注释,BLASTp完善注释结果;使用MEGA6软件构建噬菌体末端酶大亚基进化树;通过Inkscape 0.92.1软件绘制噬菌体全基因组功能模式图,噬菌体IME18与T1噬菌体基因组序列ORF比对分析图。
1.3.5 噬菌体的最佳感染复数(multiplicity of infection,MOI)及一步生长曲线
参考文献[15]所述方法进行噬菌体的最佳MOI及一步生长曲线测定。取对数期(OD600 nm=0.6)的BL21菌液5 mL,按照MOI为10、1、0.1、0.01、0.001的比值依次加入噬菌体,37 ℃、220 r/min培养4 h至培养液澄清。将培养液上清用微孔滤膜过滤,使用双层平板法测定上清液中噬菌体的效价。效价最高的MOI为该噬菌体的最佳MOI。
取对数期的BL21菌液20 mL,以最佳MOI(MOI=0.01)的比值加入噬菌体,37 ℃孵育5 min,12 000 r/min离心1 min,弃上清液,沉淀用LB培养基洗涤两次并重悬于20 mL LB培养基中。将培养液置于37 ℃、220 r/min摇床中培养90 min,每隔5~10 min取样,测定噬菌体的效价。以时间为横坐标、噬菌体效价为纵坐标绘制一步生长曲线。
1.3.6 噬菌体的体外杀菌效果
取对数期的BL21菌液,按照MOI值为10、1、0、0.1、0.01、0.001依次加入噬菌体,于37 ℃、220 r/min培养4 h至培养液澄清。分别在0~2 h每隔10 min取样,测定培养液的OD600 nm值。
1.3.7 噬菌体宿主谱测定
采用噬菌体斑点实验测定噬菌体宿主谱:取300 μL对数期的待测菌液与半固体培养基混匀,平铺在含下层固体LB培养基的平板上,静止5 min待其凝固,在细菌层上滴加2 μL噬菌体保存液,37 ℃培养8 h,观察是否有噬菌斑出现。
1.3.8 噬菌体抗性株的筛选和验证
取300 μL对数期的BL21菌液与100 μL梯度稀释的噬菌体保存液(101~108 PFU/mL)混合,室温吸附5 min,按照双层平板法培养噬菌体,延长培养时间至抗性菌株形成。采用上述噬菌体斑点法测定细菌的噬菌体敏感性。选择无法形成噬菌斑的单克隆菌液为模板,进行16S rDNA鉴定。
1.3.9 噬菌体抗性株的基因组提取、高通量测序、比较基因组学分析
随机挑选3 株鉴定正确的抗性株(BL21-R),37 ℃过夜培养。使用Pure PCR Template Preparation Kit提取细菌基因组DNA,将DNA于-20 ℃保存。抗性株的全基因组序列如上所述通过MiSeq测序平台和生物信息学软件获得。使用CLC Genomics Workbench 9.0(CLC)软件将抗性株的contigs映射到参考菌株BL21(GenBank:NC_012971)的完整基因组上进行序列比对分析。
1.3.10 噬菌体受体突变株的构建
1.3.1 0.1 质粒pKDsg-tonB、pKDsg-fhuA的构建
质粒pKDsg-tonB、pKDsg-fhuA的构建利用基于PCR扩增的循环聚合酶延伸克隆技术完成[16]。选取tonB(fhuA)基因前间区序列邻近基序(protospacer adjacent motif,PAM)前20 bp序列作为向导RNA(small guide RNA,sgRNA),重新靶向质粒pKDsg-ack[17-19]。用于PCR扩增DNA片段的两对引物为:pKDsg-tonB-F(pKDsg-fhuA-F)和gamR;pKDsg-tonB-R(pKDsgfhuA-R)和gamF(表1)。使用Q5®HotStart High-Fidelity 2×Master Mix,以pKDsg-ack质粒为模板进行PCR扩增。PCR仪器设置:98 ℃预变性2 min;98 ℃变性30 s,58 ℃退火2 min,72 ℃延伸2 min,35 个循环;72 ℃延伸2 min。使用Gel Extraction Kit进行PCR产物的凝胶纯化。通过pEASY-UniSeamLess Cloning and Assembly Kit将两段PCR产物连接。取5 μL连接产物转化100 μL感受态细胞Trans 5α,涂布于含Spec(50 mg/L)的LB平板上,30 ℃过夜培养。次日,挑取单克隆于LB液体培养基中30 ℃振荡培养12 h。使用Plasmid Mini Kit提取质粒pKDsg-tonB、pKDsg-fhuA。
表 1 实验所用引物
Table 1 Primer sequences used in this study

引物名称 引物序列(5’-3’)pKDsg-tonB-F ATTTACCTCGCCGCTTCCCCGTTTTAGAGCTAGAAATAGCAAG pKDsg-tonB-R GGGGAAGCGGCGAGGTAAATGTGCTCAGTATCTCTATCACTGAG pKDsg-fhuA-F CGCAGTTGTAGTAGCCACAGGTTTTAGAGCTAGAAATAGCAAG pKDsg-fhuA-R CTGTGGCTACTACAACTGCGGTGCTCAGTATCTCTATCACTGAG gamF AGCATTCACTAACCCCCTTTCC gamR AGCTGCTGGTAGTGACGCGCCC tonB-F TGATCGTCTTGCCTTATTGA tonB-R ATAGTCAAAAGCCTCCGGTC fhuA-F TCAGGAACGCTCAGATTGC fhuA-R ACAGCCAACTTGTGAAATGG Oligo-tonB C*C*T* GGT TTT TCT ACT GAA ATG ATT ATG ACT TCA ATG ACC CTT GAT TTA CCT CGC CGC TAA TGA TAG CCG ACG TTA CTT TCG GTC TGC ATTC Oligo-fhuA G*C*T* CAG CCA AAA CAC TCA CTG CGT AAA ATC GCA GTT GTA GTA TAA TAG TGA GTT AGC GGC ATG TCT GTT TAT GCA CAG GCA GCG
1.3.1 0.2 重组
设计外源寡核苷酸(Oligo-tonB、Oligo-fhuA)用于同源重组,5’端添加3 个硫代磷酸酯键以防止其降解[19]。使用培养至对数期的BL21菌液制备感受态细胞;将2 μL pCas9cr4、pKDsg-tonB(pKDsg-fhuA)质粒转化100 μL上述感受态细胞,并涂布在含Amp(100 mg/L)、Spec(50 mg/L)的LB平板上,30 ℃过夜培养。将含质粒pCas9cr4、pKDsg-tonB(pKDsg-fhuA)的单克隆于30 ℃培养至对数期,加入终浓度为50 mmol/L的L-阿拉伯糖继续培养15~20 min以诱导λ-Red同源重组系统表达,之后制备感受态细胞;将终浓度为2 μmol/L的外源寡核苷酸转化上述感受态细胞,涂布于含Amp(100 mg/L)、Spec(50 mg/L)、脱水四环素(100 ug/L)的LB平板,30 ℃过夜培养。次日,挑取重组的突变株Δ-tonB、Δ-fhuA单克隆于LB液体培养基中,30 ℃培养至对数期。
1.3.1 0.3 突变株tonB(fhuA)基因的测序
使用Primer-BLAST工具设计BL21的tonB(fhuA)基因鉴定引物,tonB-F和tonB-R(fhuA-F和fhuA-R)(表1)。以上述突变株菌液为模板,使用2×Trans Taq®高保真(HiFi)PCR SuperMix进行PCR扩增。将PCR产物送至北京诺塞基因公司测序。通过CLC比对突变株Δ-tonB(Δ-fhuA)和BL21的tonB(fhuA)基因差异。
1.3.11 突变株的噬菌体敏感性验证
接种突变株Δ-tonB、Δ-fhuA于LB液体培养基中,37 ℃、220 r/min培养至对数期,通过噬菌体斑点实验,噬菌体吸附实验验证突变株的噬菌体敏感性。噬菌体斑点实验根据上文所述方法完成。噬菌体吸附实验按照如下所述方法[20]:取对数期的Δ-tonB(Δ-fhuA)菌液(108 CFU/mL)1 mL,12 000 r/min离心1 min,弃上清液,用磷酸盐缓冲液洗1 次并重悬于900 μL LB培养基中;将噬菌体保存液(107 PFU/mL)100 μL加入其中并混匀,37 ℃吸附5 min,12 000 r/min离心1 min。以LB代替噬菌体保存液为对照。通过双层平板的方法测定上清液中的噬菌体的效价。
2 结果与分析
2.1 噬菌体的形态
通过双层平板法分离得到1 株噬菌体,命名为IME18,该噬菌体在平板上形成圆形、透亮的噬菌斑,其边界清晰,效价约为2.4×1011 PFU/mL(图1A)。在电镜下,噬菌体IME18头部呈正多面体对称,直径约为66 nm,尾部呈非收缩性,长约150 nm(图1B)。根据2012年国际病毒分类委员会第9次报告提出的噬菌体分类与命名标准,该噬菌体属于长尾病毒科(Siphoviridae)。
2.2 噬菌体全基因组分析
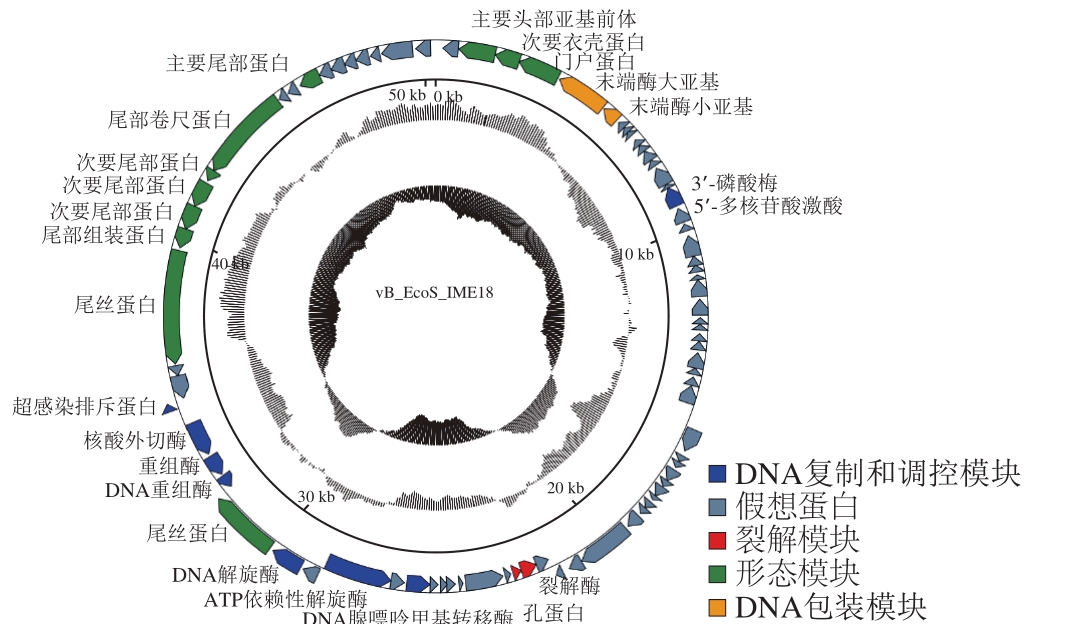
噬菌体IME18(GenBank:MH051911)基因组全长为50 354 bp,G+C含量为45.6%。在线软件tRNAscan-SE 2.0没有预测到该噬菌体含有tRNA。经RAST分析和BLASTp比对,该噬菌体共含有82 个开放阅读框(open reading frame,ORF),其中59 个ORF编码假想蛋白,23 个ORF编码功能性蛋白。功能性蛋白主要分为了4 个模块:形态模块(ORF2、ORF3、ORF4、ORF60、ORF67、ORF68、ORF69、ORF70、ORF71、ORF72、ORF75),DNA复制和调控模块(ORF16、ORF55、ORF57、ORF59、ORF61、ORF62、ORF63、ORF64),DNA包装模块(ORF5、ORF6),裂解模块(ORF47、ORF48)(图2)。
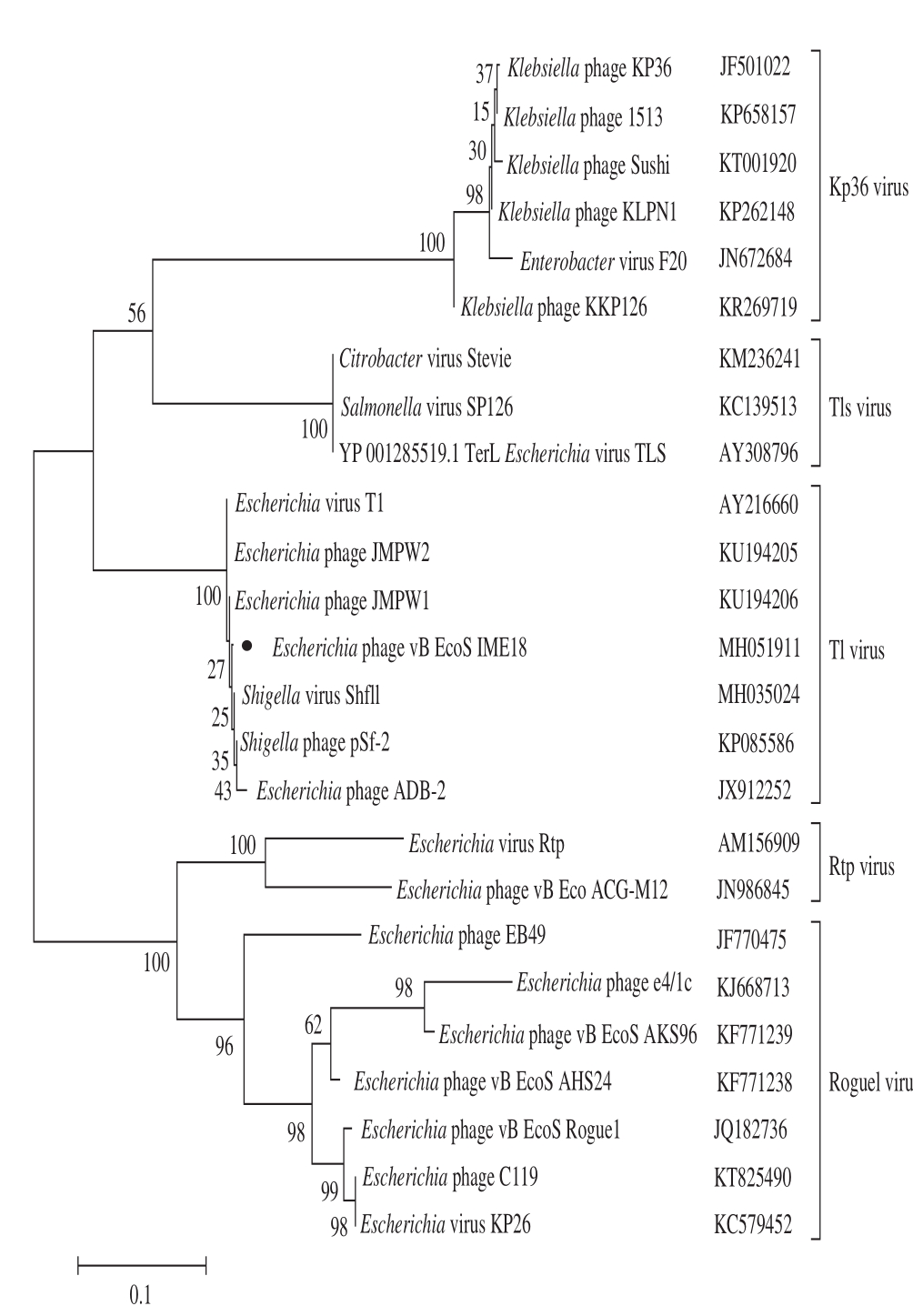
利用比较基因组学分析噬菌体IME18基因组序列ORF与T1噬菌体ORF的同源性,结果显示噬菌体IME18存在53 个(65%)ORF与T1噬菌体的同源性大于90%,有17 个(20%)ORF与T1噬菌体的同源性为50%~90%,另外12 个(15%)未知功能的ORF与T1噬菌体不存在同源性(图3)。噬菌体IME18的末端酶大亚基进化分析同样显示了噬菌体IME18与T1类噬菌体有最近的进化关系(图4)。比较基因组学分析和末端酶大亚基进化分析共同揭示了噬菌体IME18是Tunavirinae亚科,T1类的新成员。
2.3 噬菌体的最佳MOI及一步生长曲线
噬菌体IME18在MOI为0.01时具有最高效价,为2.4×1011 PFU/mL。因此,噬菌体IME18的最佳MOI为0.01。
如图5所示,噬菌体IME18感染宿主菌后5 min内效价没有明显变化,表明噬菌体IME18的潜伏期为5 min。在5~55 min内,噬菌体效价明显上升,之后趋于平稳,表明噬菌体的裂解时间为50 min。噬菌体IME18的爆发量为223 PFU/cell,表明噬菌体IME18具有很强的裂解能力。
2.4 噬菌体的体外杀菌效果
噬菌体按照不同的MOI值加入BL21的培养液中,在不同的时间点测定培养液的OD600 nm值可以反映噬菌体IME18对BL21的体外裂解能力。如图6所示,培养液的OD600 nm值呈现先还缓慢上升后极速下降趋势,MOI值越大OD600 nm值下降越快。与对照组相比,不同MOI值处理的细菌OD600 nm值在90 min内均显著下降,说明噬菌体IME18对BL21的体外裂解能力较强。
2.5 噬菌体宿主谱测定结果
噬菌体斑点实验结果显示该噬菌体对大肠杆菌BL21、DH5α、MG1655、M3110、EC600和沙门氏菌Ty21a具有裂解性。对实验室保藏的其他大肠杆菌菌株、肺炎克雷伯菌、铜绿假单胞菌均没有明显的裂解性(表2)。该噬菌体的裂解范围较窄。
表 2 噬菌体IME18的裂解谱
Table 2 Lysis spectrum of phage IME18
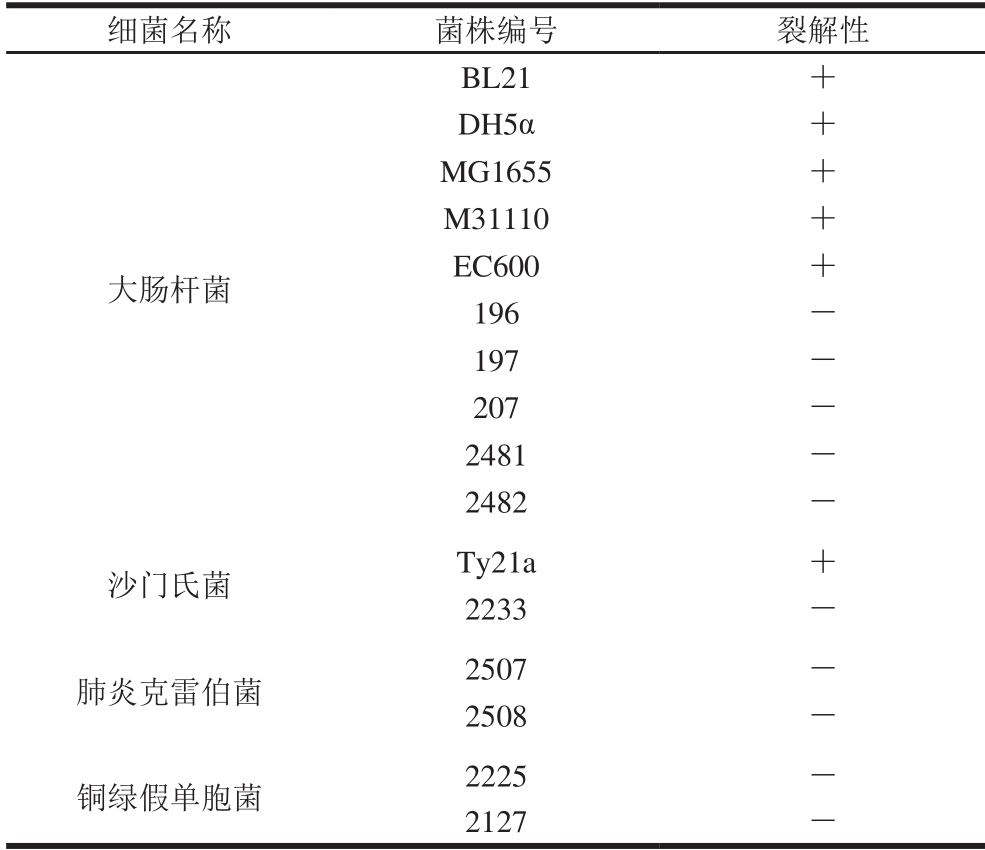
注:+.裂解;-.不裂解。
细菌名称 菌株编号 裂解性大肠杆菌BL21 +DH5α +M G1655 +M31110 +EC600 +196 -197 -207 -2481 -
G1655 +M31110 +EC600 +196 -197 -207 -2481 - 2482 -沙门氏菌 Ty21a +2233 -肺炎克雷伯菌 2507 -2508 -铜绿假单胞菌 2225 -2127 -
2482 -沙门氏菌 Ty21a +2233 -肺炎克雷伯菌 2507 -2508 -铜绿假单胞菌 2225 -2127 -
2.6 噬菌体抗性株的鉴定和基因组分析
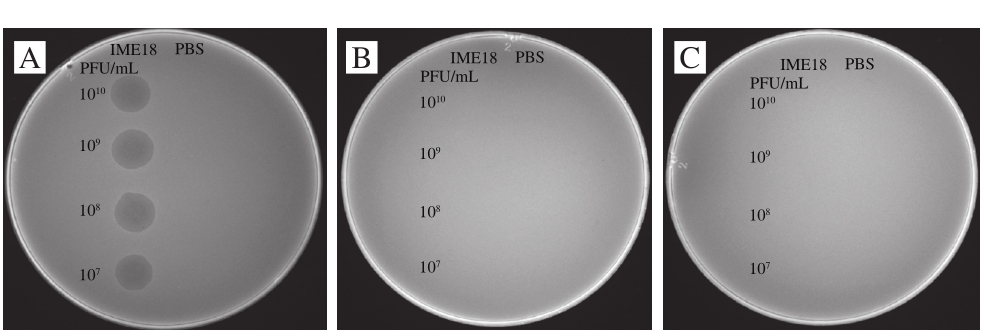
噬菌体斑点实验表明,噬菌体抗性株对噬菌体IME18产生抗性(图7B)。16S rDNA测序结果结合BLASTn比对显示,抗性株没有被其他杂菌污染。使用软件CLC将噬菌体抗性株的contigs映射到参考BL21的全基因组序列上,查找单核苷酸变异、插入、缺失。结果显示,存在15 718 bp序列在3 株耐受菌中同时缺失(表3)。比对BL21在GenBank的注释结果,噬菌体抗性株共缺失了18 个编码区序列(coding domain sequence,CDS),其中5 个CDS(CDS1、CDS3、CDS5、CDS6、CDS7)编码细菌膜蛋白,其余13 个CDS编码参与细菌生长代谢相关蛋白。该结果说明噬菌体抗性株能够通过大段基因序列的缺失来抵抗噬菌体的侵染。
表 3 BL21中与缺失片段相关的基因注释
Table 3 Gene annotation related to deletion fragments in BL21

CDS 基因注释 CDS 基因注释1 kch 10 yciG 2 ycil 11 trpA 3 tonB 12 trpB 4 yciA 13 trpC 5 yciB 14 trpD 6 yciC 15 trpE 7 ompW 16 trpL 8 yciE 17 yciV 9 yciF 18 yciO
2.7 tonB、fhuA基因的沉默
据报道,T1噬菌体使用FhuA外膜蛋白作为其第1受体,TonB蛋白作为其第2受体[21-22]。通过表3可知,选择的噬菌体抗性株没有出现fhuA基因的改变,但存在tonB基因的缺失,表明TonB蛋白在本次筛选到的抗性株中发挥重要作用。因此通过构建fhuA、tonB基因突变株来进一步研究噬菌体IME18的受体。
质粒pCas9cr4、pKDsg-tonB(pKDsg-fhuA)、外源寡核苷酸依次转入靶细胞BL21中,L-阿拉伯糖诱导重组酶的转录和翻译,脱水四环素诱导sgRNA转录和Cas9的表达;Cas9在sgRNA的指引下准确靶向目的基因,于PAM位点处切开DNA双链;含同源臂的外源寡核苷酸在重组酶的作用下整合到被切开的DNA双链上,完成基因编辑。沉默突变导致突变株Δ-tonB的TonB蛋白、Δ-fhuA的FhuA蛋白完全丧失功能。如图8所示,突变株Δ-tonB的tonB基因只表达240 个氨基酸的前8 个,Δ-fhuA的fhuA基因只能表达747 个氨基酸的前20 个。
2.8 突变株的噬菌体敏感性验证
噬菌体斑点实验和吸附实验共同测定突变株对噬菌体的敏感性。噬菌体斑点实验结果显示,突变株Δ-tonB、Δ-fhuA对噬菌体IME18产生抗性(图9)。图10噬菌体吸附实验结果显示,对照组上清液中的噬菌体效价与实验组Δ-tonB、Δ-fhuA(8.65×105 PFU/mL)基本一致,而实验组BL21上清液中噬菌体效价(5.18×105 PFU/mL)明显降低。结果表明TonB、FhuA蛋白参与噬菌体IME18的吸附和侵染。
3 讨 论
T1类噬菌体属于长尾噬菌体科,具有直径约60 nm的多面体头部,长约150 nm和直径约7 nm的非收缩性尾部的特征[23]。T1类噬菌体基因组大小约50 kb,是具有末端冗余序列的环化双链DNA[24]。本研究通过对噬菌体IME18的形态特征和全基因组序列分析,推测其属于T1类噬菌体。噬菌体IME18的效价高达2.4×1011 PFU/mL,爆发量为223 PFU/cell,能够在体外于1.5 h内显著杀死处于对数期的宿主菌,这种强裂解性表明其可能具有潜在的应用价值。
噬菌体尾丝蛋白通过与细菌表面噬菌体受体结合决定噬菌体的宿主范围。研究表明,T1噬菌体使用FhuA外膜蛋白作为其第1受体,TonB蛋白作为其第2受体[21-22]。噬菌体IME18的尾丝蛋白与T1噬菌体的尾丝蛋白存在95%的同源,推测TonB、FhuA蛋白同样在噬菌体IME18的吸附和感染中发挥关键作用,并得到验证。在大肠杆菌中,外膜蛋白FhuA主要参与铁离子配合物中铁离子的结合和转运,并且可以作为大肠杆菌素M、小菌素J25、T1噬菌体、T5噬菌体、phi80噬菌体和UC-1噬菌体的受体。TonB蛋白与外膜受体蛋白相互作用,把质子的质膜势能转化为能量,将外膜受体蛋白结合的底物运输到周质中,然后通过特定的ABC转运蛋白导入细胞质[25-27]。TonB蛋白主要参与VB12、铁离子配合物和大肠菌素M的跨膜转运,同时被证明TonB是T1噬菌体和Φ80感染的必需蛋白[25-27]。本研究通过噬菌体斑点实验和吸附实验证明了TonB、FhuA蛋白参与噬菌体IME18的吸附和侵染,为噬菌体-宿主互作研究提供了数据支持。
噬菌体一方面是引起乳品和生物发酵失败的重要因素,另一方面也在临床、食品、养殖业、工业等方面有很大的应用价值。例如,使用噬菌体治疗眼角膜脓肿和间质性角膜炎;减少肉类以及水果和蔬菜中的致病菌;减少家畜和鱼类中的细菌;检测食品和临床样品病原体[28-31]。研究噬菌体-宿主相互作用能够为更好利用噬菌体这一巨大资源提供理论支持。
参考文献:
[1] TERPE K. Overview of bacterial expression systems for heterologous protein production: from molecular and biochemical fundamentals to commercial systems[J]. Applied Microbiology and Biotechnology,2006, 72(2): 211-222. DOI:10.1007/s00253-006-0465-8.
[2] MADERA C, MONJARDIN C, SUAREZ J E. Milk contamination and resistance to processing conditions determine the fate of Lactococcus lactis bacteriophages in dairies[J]. Applied and Environmental Microbiology, 2004, 70(12): 7365-7371. DOI:10.1128/AEM.70.12.7365-7371.2004.
[3] STURINO J M, KLAENHAMMER T R. Engineered bacteriophagedefence systems in bioprocessing[J]. Nature Reviews Microbiology,2006, 4(5): 395-404. DOI:10.1038/nrmicro1393.
[4] BARRANGOU R, HORVATH P. CRISPR: new horizons in phage resistance and strain identification[J]. Annual Review of Food Science and Technology, 2012, 3(1): 143-162. DOI:10.1146/annurevfood-022811-101134.
[5] BOBAY L M, ROCHA E P, TOUCHON M. The adaptation of temperate bacteriophages to their host genomes[J]. Molecular Biology and Evolution, 2013, 30(4): 737-751. DOI:10.1093/molbev/mss279.
[6] SILVA J B, STORMS Z, SAUVAGEAU D. Host receptors for bacteriophage adsorption[J]. FEMS Microbiology Letters, 2016,363(4). DOI:10.1093/femsle/fnw002.
[7] KOSKLLA B, BROCKHURST M A. Bacteria-phage coevolution as a driver of ecological and evolutionary processes in microbial communities[J]. FEMS Microbiology Reviews, 2014, 38(5): 916-931.DOI:10.1111/1574-6976.12072.
[8] MAHONY J, SINDEREN D V. Novel strategies to prevent or exploit phages in fermentations, insights from phage-host interactions[J].Current Opinion in Biotechnology, 2015, 32: 8-13. DOI:10.1016/j.copbio.2014.09.006.
[9] DENES T, DEN BAKKER H C, TOKMAN J I, et al. Selection and characterization of phage-resistant mutant strains of listeria monocytogenes reveal host genes linked to phage adsorption[J].Applied and Environmental Microbiology, 2015, 81(13): 4295-4305.DOI:10.1128/AEM.00087-15.
[10] CHOI Y, SHIN H, LEE J H, et al. Identification and characterization of a novel flagellum-dependent Salmonella-infecting bacteriophage,IEPS5[J]. Applied and Environmental Microbiology, 2013, 79(16):4829-4837. DOI:10.1128/AEM.00706-13.
[11] LI L, PAN X, CUI X, et al. Characterization of Pseudomonas aeruginosa phage K5 genome and identification of its receptor related genes[J]. Journal of Basic Microbiology, 2016, 56(12): 1344-1353.DOI:10.1002/jobm.201600116.
[12] KIM M S, KIM Y D, HONG S S, et al. Phage-encoded colanic aciddegrading enzyme permits lytic phageinfection of a capsule-forming resistant mutant Escherichia coli strain[J]. Applied and Environmental Microbiology, 2015, 81(3): 900-909. DOI:10.1128/AEM.02606-14.
[13] KIM M, KIM S, PARK B, et al. Core lipopolysaccharide-specific phage SSU5 as an auxiliary component of a phage cocktail for Salmonella biocontrol[J]. Applied and Environmental Microbiology,2014, 80(3): 1026-1034. DOI:10.1128/AEM.03494-13.
[14] ZHANG Q, XING S, SUN Q, et al. Characterization and complete genome sequence analysis of a novel virulent Siphoviridae phage against Staphylococcus aureus isolated from bovine mastitis in Xinjiang, China[J]. Virus Genes, 2017, 53(3): 464-476. DOI:10.1007/s11262-017-1445-z.
[15] WANG Y, WANG W, LV Y, et al. Characterization and complete genome sequence analysis of novel bacteriophage IME-EFm1 infecting Enterococcus faecium[J]. Journal of General Virology, 2014,95(11): 2565-2575. DOI:10.1099/vir.0.067553-0.
[16] REISCH C R, PRATHER K L. The no-SCAR (scarless Cas9 assisted recombineering) system for genome editing in Escherichia coli[J].Scientific Reports, 2015, 5(1): 15096. DOI:10.1038/srep15096.
[17] PYNE M E, MOO-YOUNG M, CHUNG D A, et al. Coupling the CRISPR/Cas9 system with Lambda Red Recombineering enables simplified chromosomal gene replacement in Escherichia coli[J].Applied and Environmental Microbiology, 2015, 81(15): 5103-5114.DOI:10.1128/AEM.01248-15.
[18] REISCH C R, PRATHER K L J. Scarless Cas9 assisted recombineering (no-SCAR) in Escherichia coli, an easy-to-use system for genome editing[J]. Current Protocols in Molecular Biology, 2017,117: 31.8.1-31.8.20. DOI:10.1002/cpmb.29.
[19] TIAN P, WANG J, SHEN X, et al. Fundamental CRISPR-Cas9 tools and current applications in microbial systems[J]. Synthetic and Systems Biotechnology, 2017, 2(3): 219-225. DOI:10.1016/j.synbio.2017.08.006.
[20] HONG J, KIM K P, HEU S, et al. Identification of host receptor and receptor-binding module of a newly sequenced T5-like phage EPS7[J].FEMS Microbiology Letters, 2008, 289(2): 202-209. DOI:10.1111/j.1574-6968.2008.01397.x.
[21] KILLMANN H, BRAUN V. Energy-dependent receptor activities of Escherichia coli K-12: mutated TonB proteins alter FhuA receptor activities to phages T5, T1, phi 80 and to colicin M[J]. FEMS Microbiology Letters, 1994, 119(1/2): 71-76. DOI:10.1016/0378-1097(94)90394-8.
[22] KILLMANN H, VIDENOV G, JUNG G, et al. Identification of receptor binding sites by competitive peptide mapping: phages T1, T5, and phi 80 and colicin M bind to the gating loop of FhuA[J]. Journal of Bacteriology,1995, 177(3): 694-698. DOI:10.1128/jb.177.3.694-698.1995.
[23] ROBERTS M D, MARTIN N L, KROPINSKI A M. The genome and proteome of coliphage T1[J]. Virology, 2004, 318(1): 245-266.DOI:10.1016/j.virol.2003.09.020.
[24] GILL G S, MACHATTIE L A. Limited permutations of the nucleotide sequence in bacteriophage T1 DNA[J]. Journal of Molecular Biology,1976, 104(2): 505-515. DOI:10.1016/0022-2836(76)90284-9.
[25] BRAUN M, ENDRISS F, KILLMANN H, et al. In vivo reconstitution of the FhuA transport protein of Escherichia coli K-12[J]. Journal of Bacteriology, 2003, 185(18): 5508-5518. DOI:10.1128/JB.185.18.5508-5518.2003.
[26] PEACOCK R S, WELJIE A M, HOWARD S P, et al. The solution structure of the C-terminal domain of TonB and interaction studies with TonB box peptides[J]. Journal of Molecular Biology, 2005,345(5): 1185-1197. DOI:10.1016/j.jmb.2004.11.026.
[27] KHURSIGARA C M, DE CRESCENZO G, PAWELEK P D, et al. Enhanced binding of TonB to a ligand-loaded outer membrane receptor: role of the oligomeric state of TonB in formation of a functional FhuA.TonB complex[J]. Journal of Biological Chemistry, 2004, 279(9): 7405-7412. DOI:10.1074/jbc.M311784200.
[28] JEE T, VAN DOORN M B, SGMA P. Successful treatment of chronic Staphylococcus aureus-related dermatoses with the topical endolysin staphefekt SA.100: a report of 3 cases[J]. Case Reports in Dermatology, 2017, 9(2): 19-25. DOI:10.1159/000473872.
[29] MAGNONE J P, MAREK P J, SULAKVELIDZE A, et al. Additive approach for inactivation of Escherichia coli O157: H7, Salmonella,and Shigella spp. on contaminated fresh fruits and vegetables using bacteriophage cocktail and produce wash[J]. Journal of Food Protection,2013, 76(8): 1336-1341. DOI:10.4315/0362-028X.JFP-12-517.
[30] KHAIRNAR K, RAUT M P, CHANDEKAR R H, et al. Novel bacteriophage therapy for controlling metallo-beta-lactamase producing Pseudomonas aeruginosa infection in catfish[J]. BMC Veterinary Research, 2013, 9(1): 264. DOI:10.1186/1746-6148-9-264.
[31] KLUSKENS L D, FREITAS P P, AZEREDO J. A bacteriophage detection tool for viability assessment of Salmonella cells[J].Biosensors & Boelectronics, 2014, 52: 239-246. DOI:10.1016/j.bios.2013.08.053.
Isolation and Identification of a T1-Like Bacteriophage vB_EcoS_IME18 and Analysis of Its Receptor
LI Ping1, LIN Hong2, TONG Yigang2,*, WANG Jingxue1,*
(1. College of Food Science and Engineering, Ocean University of China, Qingdao 266003, China;2. State Key Laboratory of Pathogenic Microbiology Safety, Institute of Microbiological Epidemiology,Institute of Military Medicine, Academy of Military Sciences, Beijing 100071, China)
Abstract: A strain of phage, designated vB_EcoS_IME18 (IME18), was isolated using Escherichia coli BL21 as the indicator bacterium. The morphology of the phage was observed by electron microscopy and the multiplicity of infection,one-step growth curve, lysis spectrum and in vitro bactericidal effect were determined; the genomic sequencing was performed by Illumina MiSeq sequencer and genomic features were analyzed by comparative genomics; the cause of phage resistance was analyzed by screening for phage resistant strains and confirmed by receptor gene silencing mutation, phage spotting, and phage adsorption assay. The results showed that phage IME18 had an icosahedral head and a non-contractile tail, belonging to the T1-like in the Tunavirinae subfamily in the family of Siphoviridae. The multiplicity of infection of phage IME18 was 0.01, latent period 5 min, burst period 50 min and burst size 223 PFU/cell; in vitro, phage IME18 could effectively kill BL21 in the log phase in 90 min. The total length of the genome was 50 354 bp with a G + C content of 45.6%. In addition, the proteins FhuA and TonB were proved to play a key role in the adsorption and infestation of phage IME18.
Keywords: Escherichia coli; bacteriophage; identification; phage receptor
收稿日期:2018-05-18
基金项目:“十三五”国家重点研发计划重点专项(2017YFC1600703);国家重点研发计划项目(AWS16J020);山东省重点研发计划项目(2017GNC1318)
第一作者简介:李萍(1994—)(ORCID: 0000-0002-3241-7076),女,硕士研究生,研究方向为食品质量与安全。E-mail: 13126538751@163.com
*通信作者简介:童贻刚(1966—)(ORCID: 0000-0002-8503-8045),男,教授,博士,研究方向为分子微生物学、组学与生物信息学。E-mail: tong.yigang@gmail.com王静雪(1976—)(ORCID: 0000-0001-6705-815X),女,教授,博士,研究方向为食品质量与安全,应用微生物。E-mail: snow@ouc.edu.cn
DOI:10.7506/spkx1002-6630-20180518-258
中图分类号:Q939.92
文献标志码:A
文章编号:1002-6630(2019)08-0079-08
引文格式:
李萍, 林洪, 童贻刚, 等. 1 株T1样噬菌体vB_EcoS_IME18的分离鉴定及其受体分析[J]. 食品科学, 2019, 40(8): 79-86.
DOI:10.7506/spkx1002-6630-20180518-258. http://www.spkx.net.cn
LI Ping, LIN Hong, TONG Yigang, et al. Isolation and identification of a T1-like bacteriophage vB_EcoS_IME18 and analysis of its receptor[J]. Food Science, 2019, 40(8): 79-86. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-20180518-258. http://www.spkx.net.cn






 G1655 +M31110 +EC600 +196 -197 -207 -2481 -
G1655 +M31110 +EC600 +196 -197 -207 -2481 -