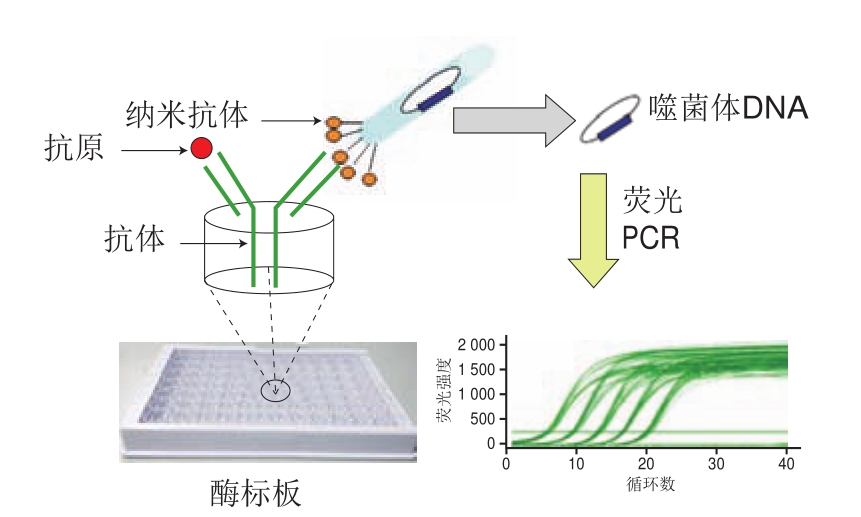
图 1 基于噬菌体展示纳米抗体的定量免疫PCR原理示意图
Fig. 1 Schematic diagram of phage displayed nanobody based real-time immuno-PCR
脱氧雪腐镰刀菌烯醇(deoxynivalenol,DON)是一种常见的真菌次级代谢产物,主要由禾谷镰刀菌(Fusarium graminearum)产生[1],此外还有尖孢镰刀菌(F. oxysporum)、串珠镰刀菌(F. moniliforme)、拟枝孢镰刀菌(F. sporotricoides)、粉红镰刀菌(F. roseum)和雪腐镰刀菌(F. nivale)等亦可产生,DON广泛存在于大麦、小麦、玉米、燕麦等作物中,是一种全球性污染的真菌毒素[2-3]。
目前,检测DON的主要方法有薄层层析法[4]、高效液相色谱法[5]、气相色谱法、气相色谱-质谱法、胶体金免疫层析法[6]、酶联免疫吸附(enzyme linked immunosorbent assay,ELISA)法[7-9]等。其中ELISA法是目前较为常用的真菌毒素检测方法,该方法是在抗原抗体免疫学反应特异性结合的基础上,利用酶的高效催化作用以放大信号实现检测,具有操作简便、特异性强和灵敏度高等优点。然而,传统的真菌毒素免疫分析体系大多基于竞争型模式,制备真菌毒素全抗原(包被抗原或竞争抗原)是建立竞争型免疫分析方法的前提和关键。目前,传统的真菌毒素全抗原需以标准品为原料,通过化学合成的方式,与大分子蛋白偶联制备而成,存在制备成本高、批次误差大且对环境具有毒害作用等缺陷。
有鉴于此,寻求制备真菌毒素全抗原替代物用于免疫学分析的测定已成为当前研究的热点。近些年来,噬菌体展示技术在真菌毒素替代物上提供了一个新的思路。该技术是将外源编码蛋白的基因插入到噬菌体衣壳蛋白基因中,然后同衣壳蛋白进行融合表达,从而实现展示外源蛋白的目的[10]。由此,可以通过生物亲和淘选得到能和靶物质特异性结合的噬菌体,而且噬菌体具有快速大批量的生产等优点。Xu Yang等[11-12]将橘霉素(citrinin,CIT)特异性单克隆抗体作为靶标,从天然羊驼抗体库及天然骆驼抗体库分别淘选得到CIT的噬菌体展示抗独特型纳米抗体,替代传统的CIT化学合成抗原应用于免疫学分析体系,实现CIT的绿色免疫分析。Wang Xianxian等[13]将玉米赤霉烯酮(zearalenone,ZEN)特异性单克隆抗体作为靶标,从天然羊驼噬菌体展示纳米抗体文库淘选得到ZEN的噬菌体展示抗独特型纳米抗体,实现了免疫学分析ZEN的无毒检测。Ji Yanwei等[14]将赭曲霉毒素A(ochratoxin,OTA)特异性单克隆抗体作为靶标,从驼源天然纳米抗体文库淘选得到OTA的噬菌体展示抗独特型纳米抗体,替代传统的OTA人工合成抗原,建立OTA环境友好型的免疫检测方法。He Zhenyun等[15]将OTA特异性单克隆抗体作为靶标,从噬菌体展示随机十二肽库和环七肽库淘选得到7 株OTA的噬菌体展示模拟表位,替代传统的OTA人工抗原,实现了OTA的无毒免疫分析。He Qinghua等[16]将ZEN特异性单克隆抗体作为靶标,从噬菌体展示十二肽库淘选得到5 株ZEN的噬菌体展示抗原模拟表位,很好地应用于ZEN的绿色免疫学分析。此外,Shu Mei[17]和Wang Yanru[18]等分别得到了伏马菌素B1(fumonisin B1,FB1)和黄曲霉毒素B(aflatoxin,AFB)的抗独特型抗体。抗独特型抗体可以模拟抗原的结合位点,同时又与天然抗原竞争,可以作为良好的抗原模拟物用于免疫学分析。
荧光定量免疫聚合酶链式反应(polymerase chain reaction,PCR)法是20世纪90年代被开发出来的一项技术,该方法结合了抗原抗体免疫学反应强特异性和PCR高扩增的敏感性,一定程度上弥补了ELISA方法不适用于小分子、示踪物质及低浓度样本的定量检测。Sano等[19]在1992年建立了一种新型的检测技术即免疫PCR,该方法是将特异性抗体和核酸分子偶联,用核酸作为标记物进行PCR扩增以达到放大信号的目的。由于PCR的超高扩增效率使得该方法的灵敏度有几个数量级的提升,很大程度上解决了很多物质的微量检测问题[20-22]。然而,该类方法由于需要特异性抗体和核酸分子的偶联过程,不仅操作耗时、繁琐,而且破坏了抗体及DNA的活性。近年来,研究者们开发出一种新型的噬菌体展示-免疫PCR方法,利用噬菌体展示技术将特异性抗体和其核酸序列直接联系在一起,可直接进行PCR扩增,省去了特异性抗体和核酸分子偶联的繁琐过程。Liu Yuanyuan等[23]基于抗Cry1Ac噬菌体展示纳米抗体建立的定量免疫PCR检测Cry1Ac方法,最低检出限为0.1 pg/mL。Lei Jiawen等[24]基于抗AFB的噬菌体展示抗独特型纳米抗体建立定量免疫PCR检测谷物和饲料中的AFB,最低检出限为0.02 ng/mL。抗独特型纳米抗体作为无毒试剂的替代和PCR的高扩增敏感的信号放大作用可以使检测的安全性和灵敏度大大提高。目前,应用该方法进行DON检测的文献鲜见报道。
本实验室在前期工作中,已从驼源天然纳米抗体库中淘选出1 株鼠源DON单克隆抗体特异性结合的噬菌体展示抗独特型纳米抗体[25]。该噬菌体颗粒既可以作为检测抗原用于免疫学分析,同时噬菌体颗粒内包裹的DNA又可以作为DNA模板进行PCR扩增。基于这个原理(图1),本研究根据鼠源DON单克隆抗体特异性结合的噬菌体展示抗独特型抗体,建立一种基于噬菌体展示纳米抗体的高灵敏、特异性检测DON的免疫荧光定量PCR方法。该方法直接使用噬菌体展示抗独特型纳米抗体作为竞争抗原的替代物,应用于免疫PCR体系,避免了使用传统的化学合成酶标抗原所带来的环境污染、操作毒性等缺陷,在食品安全尤其是在真菌毒素、致病菌[26]、农药残留[27]、转基因食品以及肿瘤标记物[28-29]的绿色灵敏检测方面具有广泛的应用前景。

图 1 基于噬菌体展示纳米抗体的定量免疫PCR原理示意图
Fig. 1 Schematic diagram of phage displayed nanobody based real-time immuno-PCR
DL2000 DNA Marker、SYBR荧光定量PCR试剂盒日本TaKaRa公司;Goldview核酸染料 北京索莱宝科技有限公司;抗DON单克隆抗体、抗DON单抗的噬菌体展示纳米抗体(P-28)由本实验室自制;DON标准品美国Sigma公司;HRP酶标记的抗M13二抗 美国GE Healthcare公司;E.coli TG1由本实验室保存;BSA、OVA 生工生物工程(上海)有限公司;96 孔聚丙烯PCR板 美国ABI公司;20% PEG/NaCl溶液(141.1 g NaCl+200 g PEG8000,去离子水定容至1 L);0.1 mol/L Gly-HCl溶液(0.75 g甘氨酸+2 mol/L HCl溶液调节pH 2.2,去离子水定容至100 mL);2 mol/L Tris-HCl溶液(48.44 g Tris+HCl调节pH 8.5,去离子水定容至200 mL);0.01 mol/L磷酸盐缓冲液(phosphate buffer saline,PBS)(KH2PO4 0.27 g,NaCl 8 g,KCl 0.2 g,Na2HPO4·12H2O 3.58 g,5 mol/L NaOH溶液调pH 7.4,加超纯水定容至1 L);0.05% PBST(1 L 0.01 mol/L PBS+0.5 mL Tween-20);TMB显色液;2 mol/L H2SO4终止液(10 mL 98%的H2SO4+80 mL超纯水);LB培养基(5 g酵母提取物,10 g细菌培养用胰蛋白胨,10 g NaCl定容至1 L超纯水);LB/Amp平板(LB培养基+15 g/mL琼脂粉+0.1 mmol/L Amp);Bind/Wash Buffer(20 mmol/L Na2HPO4·12H2O,0.15 mol/L NaCl,pH 8.0,真空抽滤);Elution Buffer(0.1 mol/L甘氨酸,pH 2.5,真空抽滤);中和Buffer(1 mol/L Tris-HCl,pH 8.5,真空抽滤)。
ECO0.9型超净工作台 美国Thermo公司;微量移液器 芬兰Labsystems公司;HQL150B大幅度恒温摇床上海智城分析仪器制造有限公司;CFX96 realtime PCR仪美国Bio-Rad公司。
1.3.1 抗DON单克隆抗体的纯化
取出Protein G亲和层析柱,竖直固定上样管于铁架台,关闭活塞;预先加入1 mL的Bind/Wash Buffer至层析柱中,充分摇匀,加入1 mL的匀浆,静置15~30 min。加入5 mL Bind/Wash Buffer到柱中,流速约为1 mL/min,流净后关闭活塞,加入用Bind/Wash Buffer 1∶1稀释的DON单克隆抗体腹水,室温静置30 min,流速约为1 mL/min,收集流穿液。加入30 mL Bind/Wash Buffer,流速约为2 mL/min,收集洗涤液,洗杂后,加入10 mL Elution Buffer流速约为1 mL/min,收集洗脱液,然后用1/10体积的中和Buffer调节pH 7.4。加入10 mL Bind/Wash Buffer洗涤柱子,关闭活塞,加入10 mL含20%乙醇溶液的Bind/Wash Buffer,4 ℃保存。
1.3.2 抗DON单抗的噬菌体展示纳米抗体(P-28)的扩增
P-28划线于LB/Amp平板培养基,37 ℃过夜培养,挑取单克隆接种于5 mL LB/Amp培养基中,37 ℃、220 r/min培养6 h。1%的接种量接种于25 mL LB/Amp培养基,37 ℃、220 r/min培养至OD值0.35~0.5左右;加入辅助噬菌体 M13KO7(感染复数1∶1),37 ℃静置孵育15 min后转至摇床,37 ℃、220 r/min培养45~60 min。培养液在4 ℃、8 000 r/min离心10 min,弃上清液,25 mL LB/Amp/Kana培养基重悬菌体,30 ℃、200 r/min培养6 h。4 ℃、8 000 r/min离心10 min,取上清液,加入1/5体积的PEG/NaCl,冰上静置4 h以上;菌液在4 ℃、8 000 r/min离心10 min,弃上清液,用1 mL 1×PBS悬浮噬菌体,加入1/5体积的PEG/NaCl ,冰上孵育1 h,4 ℃、8 000 r/min离心10 min,弃上清液,250 µL 1×PBS重悬噬菌体并测滴度。
1.3.3 引物设计
根据抗DON单抗的噬菌体展示纳米抗体P-28的VHH片段FR3-CDR3区序列设计引物,并由南京金斯瑞公司合成。引物序列如下:P28-F 5’-3’ ACT ATA TAG ACT CCG TGA AGG G(22 bp);P28-R 5’-3’ AGC ACT ATA AGG TAC TGT CGA AC(23 bp)。
1.3.4 引物验证
将合成的引物扩增抗DON单抗的噬菌体阳性克隆,以P-28为模板进行普通PCR。反应体系如下:10×PCR buffer 2 µL,dNTPs(2.5 mmol/L)1.6 µL,Mg2+ 1.2 µL,P28-F 0.5 µL,P28-R 0.5 µL,模板0.5 µL,Taq酶0.15 µL,ddH2O 13.55 µL,反应体系共计20 µL。PCR条件如下: 95 ℃变性10 min;退火、延伸(95 ℃、1 min,60 ℃、30 s,72 ℃、30 s)×30;72 ℃、10 min。将PCR产物经1%的琼脂糖凝胶电泳后,于紫外成像仪下观察结果并拍照。1.3.5 PCR退火温度的优化
以P-28噬菌体为模板,P28-F、P28-R为特异性引物,进行定量PCR,实验共设计88 孔,各行设置梯度退火温度:55~65 ℃;各列设置梯度噬菌体数量1010、109、108、107、106、0,定量PCR体系如下:2×SYBR Premix Ex Taq 10 µL,P28-F(10 µmol/L)0.5 µL,P28-R(10 µmol/L)0.5 µL,P-28噬菌体6 µL,ddH2O 3 µL,反应体系共计20 µL。PCR条件:变性95 ℃、10 min;退火95 ℃、30 s,55~65 ℃、30 s,延伸72 ℃、30 s,40 个循环;循坏后添加溶解曲线。
1.3.6 定量PCR检测DON方法的建立
用抗DON的单克隆抗体(10 µg/mL)包被96 孔酶标板,4 ℃孵育过夜,PBST洗板4 次,加入300 µL/孔4%的脱脂牛奶-PBS封闭,37 ℃孵育2 h,PBST洗板4 次。加入50 µL梯度质量浓度的DON标品(1 000、100、10、1、0.1、0 ng/mL)/待测样品和50 µL P-28(1∶3 200),PBST洗板4 次,加入100 µL 0.1 mol/L Gly-HCl(pH 2.2),水平摇床振荡孵育10 min后用移液器吸出,加入10 µL 2 mol Tris-HCl进行中和,移液器吹打数次混匀,取6 µL中和液进行定量PCR。定量PCR体系:2×SYBR Premix Ex Taq 10 µL、P28F(10 µmol) 0.5 µL、P28R(10 µmol) 0.5 µL、P-28噬菌体6 µL、加超纯水至20 µL。定量PCR条件:变性95 ℃、10 min;退火95 ℃、30 s,65 ℃、30 s,延伸72 ℃、30 s,40 个循环;循环后添加溶解曲线。
1.3.7 加标回收实验
准确称取1 g空白玉米样品,分别加入1、10、100、1 000 µg/kg的DON,然后加入10 mL PBS提取液,置于涡旋振荡器上充分振荡15 min,8 000 r/min离心20 min,收集上清液,按1.3.6节所述方法进行DON含量的测定,计算加标回收率及变异系数。
固相亲和淘选是以纯度高的抗DON单克隆抗体作为靶体,得到与之特异性结合并能与DON标准品竞争结合抗DON单抗的噬菌体展示纳米抗体P-28,因此纯度较高的抗DON单克隆抗体与噬菌体展示纳米抗体P-28结合活性较好。故本实验采用金斯瑞Protein G亲和层析柱法对抗DON单克隆抗体腹水进行纯化,纯化后抗体的SDS-PAGE分析如图2所示,在洗脱液中55 kDa和25 kDa处各有一条清晰的条带,分别为抗DON单克隆抗体的重链与轻链。抗DON单克隆抗体的纯度较高。
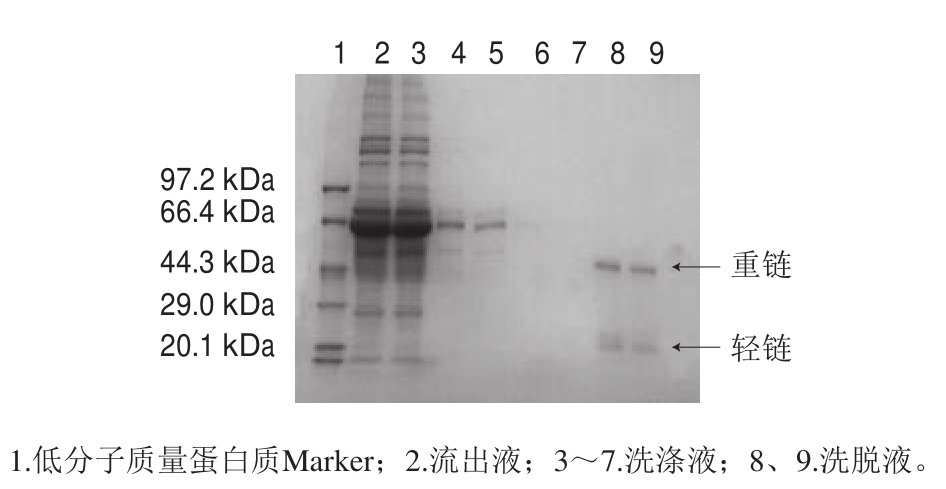
图 2 免疫亲和层析柱法纯化的抗DON单克隆抗体的SDS-PAGE分析
Fig. 2 SDS-PAGE analysis of anti-DON monoclonal antibodies purified by immuno-affinity chromatography
P-28是基于噬菌体展示文库淘选出来的抗DON单抗的噬菌体展示纳米抗体,为建立DON的荧光定量PCR检测方法,依据P-28 VHH编码基因FR3-CDR3区设计PCR引物。利用普通PCR方法对引物进行验证,将rTaq酶扩增后的反应产物经琼脂糖凝胶电泳后,于凝胶成像系统进行观察,结果如图3所示,获得目的条带为149 bp,成功扩增出目的序列,表明该引物可以进行荧光定量PCR的后续实验。

图 3 噬菌体展示纳米抗体P-28 VHH编码基因FR3-CDR3区PCR产物的琼脂糖凝胶电泳分析
Fig. 3 Agarose gel electrophoresis analysis of PCR amplified FR3-CDR3 region of the VHH encoding gen of phage displayed nanobody P-28
采用梯度PCR的方法,设置模板数梯度(106、107、108、109、1010 PFU/mL)和退火温度梯度(55~65 ℃),配制PCR体系后进行扩增。结果表明,定量PCR特异性良好,熔点曲线只存在单一峰(图4)。P-28噬菌体定量PCR扩增曲线如图5所示,作出每个退火温度下,模板数的对数(X)与Ct值(Y)的相关性曲线,如表1所示,起始模板数越多,Ct越小,并且每个模板的Ct值与模板数的对数值存在线性关系。对比不同退火温度下的相关系数R2和扩增效率,当退火温度65 ℃时,R2为1且扩增效率达96.33%,表明在退火温度65 ℃条件下,PCR的稳定性和重复性较好。因此,在后续实验中,退火温度均设为65 ℃。
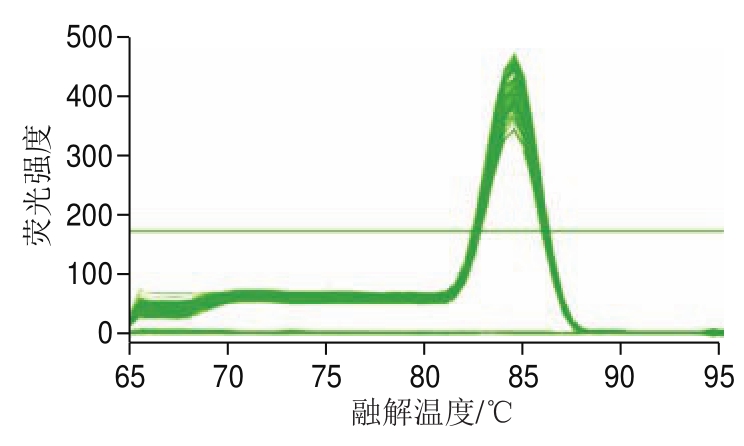
图 4 P-28噬菌体定量PCR熔点曲线峰图
Fig. 4 qPCR melting point curve for P-28 phage
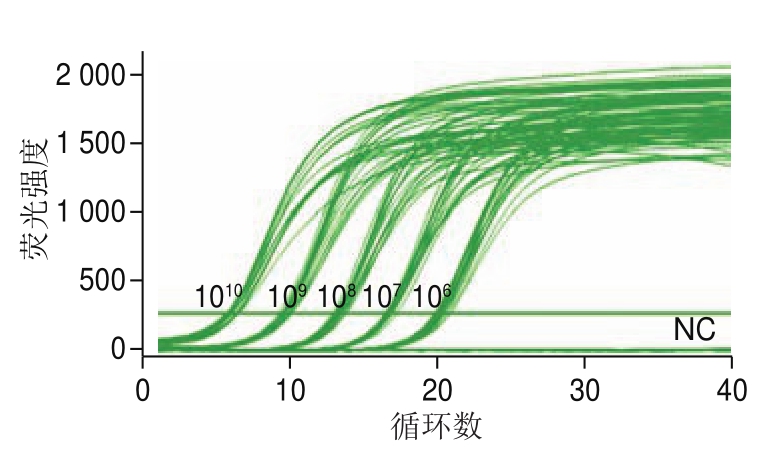
图 5 不同退火温度下噬菌体模板的定量PCR扩增曲线图
Fig. 5 qPCR amplification curves of phage template at different annealing temperatures
表 1 不同退火温度下噬菌体模板数与Ct值的线性方程
Table 1 Standard curves of phage template at different annealing temperatures

退火温度/℃ 线性回归方程 相关系数65 Y=40.76-3.41X 1.000 0 64.5 Y=41.36-3.52X 0.999 0 63.3 Y=40.91-3.49X 0.999 7 61.4 Y=40.94-3.48X 0.999 9 59 Y=41.77-3.58X 0.999 8 57 Y=41.56-3.54X 0.999 4 55.7 Y=42.61-3.66X 0.999 5 55 Y=42.29-3.58X 0.999 4
基于抗DON单抗的噬菌体展示纳米抗体P-28,建立基于间接竞争的定量免疫PCR检测DON的方法。本研究通过方正滴定方法确定最佳DON单克隆抗体包被质量浓度为10 µg/mL,噬菌体的稀释倍数为1∶3 200,抗原、抗体的反应时间为1 h。再在此条件下建立免疫荧光PCR检测DON的标准曲线,基于噬菌体展示纳米抗体P-28定量PCR检测DON的扩增曲线如图6所示,然后以DON标准品质量浓度对数值(x)为横坐标、Ct值(y)为纵坐标绘制标准曲线,线性方程为y=24.11+0.51x,相关系数R为0.995。
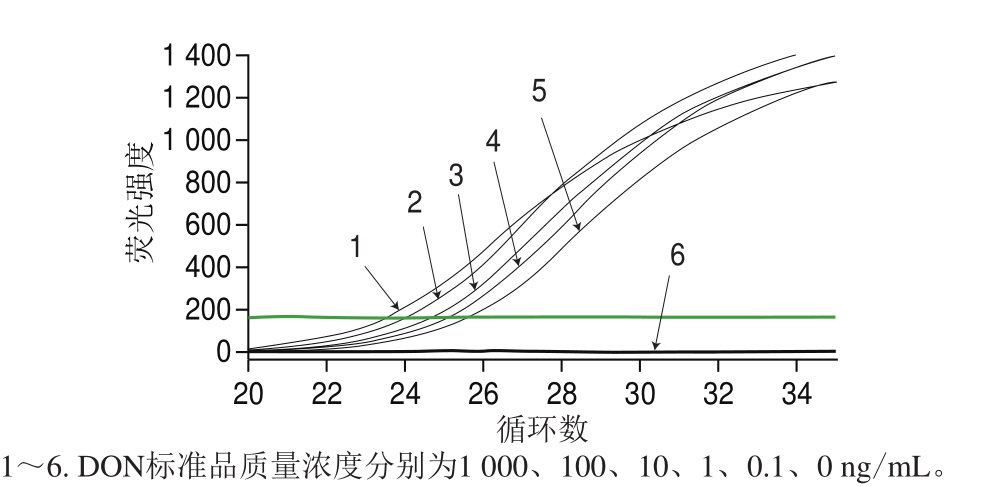
图 6 基于噬菌体展示纳米抗体定量PCR检测DON的扩增曲线图
Fig. 6 Amplification curves for qPCR detection of DON based on phage displayed nanobody
本研究建立的免疫荧光PCR方法的线性范围为0.1~1 000 ng/mL,IC50值为(3.96±2.21)ng/mL,最低检出限为0.048 ng/mL,拟合曲线相关系数R2为0.996,组间误差、批间误差、日间误差分别为4.2%、6.2%、8.9%(n=3)。采用间接竞争ELISA测定P-28的特异性,加入其他4 种常见的真菌毒素FB1、ZEN、AFB1和OTA,进行间接竞争分析,P-28与其他4 种真菌毒素均无明显交叉反应,表明该方法对DON定量检测特异性良好。
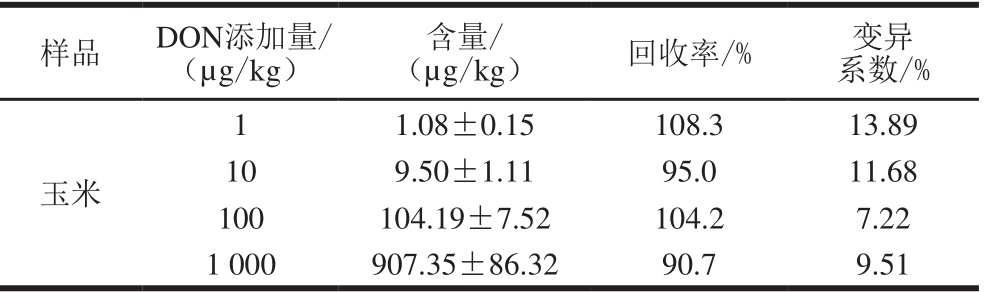
为验证所开发方法的可靠性,本实验向阴性的玉米样品中添加不同质量浓度的DON标准品,经PBS提取后,进行定量免疫PCR分析,结果如表2所示,该方法用于检测DON的加标回收率为90.7%~108.3%,变异系数为7.22%~13.89%,表明该方法具有良好的准确性及重复性。
表 2 基于噬菌体展示纳米抗体的绿色免疫PCR分析DON的加标回收实验
Table 2 Recovery of DON from spiked corn by phage displayed nanobody-mediated green immuno-PCR
注:含量为 ±s;n=3。
±s;n=3。
样品 DON添加量/(µg/kg)含量/(µg/kg)回收率/%变异系数/%玉米1 1.08±0.15 108.3 13.89 10 9.50±1.11 95.0 11.68 100 104.19±7.52 104.2 7.22 1 000 907.35±86.32 90.7 9.51
噬菌体展示-免疫PCR方法采用噬菌体粒子作为检测抗原或者抗体,同时也提供在噬菌体展示-免疫PCR扩增过程中的DNA模板。由于荧光染料的存在,随着PCR的进行,双链PCR产物呈指数增长,荧光染料与双链产物结合使得荧光信号以指数形式不断放大,这一信号放大过程使得此方法的灵敏度和线性范围得到了显著改善。
本研究针对噬菌体展示纳米抗体技术淘选出来的抗DON单抗的噬菌体展示抗独特型纳米抗体P-28,建立了一种高灵敏、特异性检测DON的荧光定量PCR的检测方法,IC50值为从传统的噬菌体ELISA值(77.89±1.77)ng/mL[25]降低至(3.96±2.21)ng/mL,最低检出限为0.048 ng/mL,线性范围从21.14~316.52 ng/mL[25]扩宽到0.1~1 000 ng/mL。相比于传统phage-ELISA检测方法,此方法提高了DON的检测灵敏度及检测范围。同时,本研究将抗DON单抗的噬菌体展示纳米抗体P-28用于替代小分子抗原用于免疫学检测,降低了因引入DON标准品制备全抗原对人体的毒害和环境的污染,提高了产品的应用价值[30],因此在食品安全尤其是真菌毒素检测领域的应用具有广泛的应用前景。
[1] MARAGOS C M, MCCORMICK S P. Monoclonal antibodies for the mycotoxins deoxynivalenol and 3-acetyl-deoxynivalenol[J].Food & Agricultural Immunology, 2000, 12(3): 181-192.DOI:10.1080/09540100050140722.
[2] LANCOVA K, HAJSLOVA J, POUSTKA J, et al. Transfer of fusarium mycotoxins and ‘masked’ deoxynivalenol (deoxynivalenol-3-glucoside) from field barley through malt to beer[J]. Food Additives &Contaminants: Part A, 2008, 25(6): 732-744.
[3] LORI G A, SISTERNA M N, HAIDUKOWSKI M, et al. Fusarium graminearum and deoxynivalenol contamination in the durum wheat area of Argentina[J]. Microbiological Research, 2003, 158(1): 29-35.DOI:10.1078/0944-5013-00173.
[4] VEGAHERRERA M, MADARIAGA R, ARANDA M, et al.Confirmation of deoxynivalenol presence in chilean wheat by highperformance thin-layer chromatography-mass spectrometry[J].Journal of the Chilean Chemical Society, 2017, 62(2): 3435-3437.DOI:10.4067/S0717-97072017000200003.
[5] KIM D H, HONG S Y, JEON M H, et al. Simultaneous determination of the levels of deoxynivalenol, 3-acetyldeoxynivalenol, and nivalenol in grain and feed samples from South Korea using a high-performance liquid chromatography-photodiode array detector[J]. Applied Biological Chemistry, 2016, 59(6): 1-7.
[6] HUANG Z B, XU Y, LI L S, et al. Development of an immunochromatographic strip test for the rapid simultaneous detection of deoxynivalenol and zearalenone in wheat and maize[J]. Food Control, 2012, 28(1): 7-12. DOI:10.1016/j.foodcont.2012.04.035.
[7] FOLLONI S, BELLOCCHI G, KAGKLI D M, et al. Development of an ELISA reverse-based assay to assess the presence of mycotoxins in cereal flour[J]. Food Analytical Methods, 2011, 4(2): 221-227.DOI:10.1007/s12161-010-9150-8.
[8] SINHA R C, SAVARD M E, LAU R. Production of monoclonal antibodies for the specific detection of deoxynivalenol and 15-acetyldeoxynivalenol by ELISA[J]. Journal of Agricultural & Food Chemistry, 1995, 43(6): 1740-1744. DOI:10.1021/jf00054a061.
[9] WANG S, DU X, HUANG Y, et al. Detection of deoxynivalenol based on a single-chain fragment variable of the antideoxynivalenol antibody[J]. FEMS Microbiology Letters, 2007, 272(2): 214-219.DOI:10.1111/j.1574-6968.2007.00765.x.
[10] SMITH G P. Filamentous fusion phage: novel expression vectors that display cloned antigens on the virion surface[J]. Science, 1985, 228:1315-1317. DOI:10.1126/science.4001944.
[11] XU Y, XIONG L, LI Y P, et al. Citrinin detection using phagedisplayed anti-idiotypic single-domain antibody for antigen mimicry[J]. Food Chemistry, 2015, 177: 97-101. DOI:10.1016/j.foodchem.2015.01.007.
[12] XU Y, XIONG L, LI Y P, et al. Anti-idiotypic nanobody as citrinin mimotope from a naive alpaca heavy chain single domain antibody library[J]. Analytical & Bioanalytical Chemistry, 2015, 407(18): 5333-5341. DOI:10.1007/s00216-015-8693-3.
[13] WANG X X, HE Q H, XU Y, et al. Anti-idiotypic VHH phage displaymediated immuno-PCR for ultrasensitive determination of mycotoxin zearalenone in cereals[J]. Talanta, 2016, 147: 410-415. DOI:10.1016/j.talanta.2015.09.072.
[14] JI Y W, HE Q H, XU Y, et al. Phage displayed anti-idiotypic nanobody mediated immuno-PCR for sensitive and environmentally friendly detection of mycotoxin ochratoxin A[J]. Analytical Methods, 2016,8(43). DOI:10.1039/C6AY01264G.
[15] HE Z Y, HE Q H, XU Y, et al. Ochratoxin A mimotope from secondgeneration peptide library and its application in immunoassay[J].Analytical Chemistry, 2013, 85(21): 10304-10311. DOI:10.1021/ac402127t.
[16] HE Q H, XU Y, HUANG Y H, et al. Phage-displayed peptides that mimic zearalenone and its application in immunoassay[J].Food Chemistry, 2011, 126(3): 1312-1315. DOI:10.1016/j.foodchem.2010.11.085.
[17] SHU M, XU Y, WANG D, et al. Anti-idiotypic nanobody: a strategy for development of sensitive and green immunoassay for fumonisin B1[J].Talanta, 2015, 143: 388-393. DOI:10.1016/j.talanta.2015.05.010.
[18] WANG Y R, LI P W, MAJKOVA Z, et al. Isolation of alpaca antiidiotypic heavy-chain single-domain antibody for the aflatoxin immunoassay[J]. Analytical Chemistry, 2013, 85(17): 8298-8303.DOI:10.1021/ac4015885.
[19] SANO T, SMITH C L, CANTOR C R. Immuno-PCR: very sensitive antigen detection by means of specific antibody-DNA conjugates[J].Science, 1992, 258: 120-122. DOI:10.1126/science.1439758.
[20] KUCZIUS T, BECKER K, FISCHER A, et al. Simultaneous detection of three CNS indicator proteins in complex suspensions using a single immuno-PCR protocol[J]. Analytical Biochemistry, 2012, 431(1): 4-10.DOI:10.1016/j.ab.2012.08.029.
[21] ASSUMPÇÃO A L F V, DA SILVA S R. Immuno-PCR in cancer and non-cancer related diseases: a review[J]. Veterinary Quarterly, 2016,36(2): 63-70. DOI:10.1080/01652176.2016.1164912.
[22] LUBELLI C, CHATGILIALOGLU A, BOLOGNESI A, et al. Detection of ricin and other ribosome-inactivating proteins by an immunopolymerase chain reaction assay[J]. Analytical Biochemistry, 2006,355(1): 102-109. DOI:10.1016/j.ab.2006.05.003.
[23] LIU Y Y, JIANG D J, LU X, et al. Phage-mediated immuno-PCR for ultrasensitive detection of Cry1Ac protein based on nanobody[J].Journal of Agricultural and Food Chemistry, 2016, 64(41): 7882-7889.DOI:10.1021/acs.jafc.6b02978.
[24] LEI J W, LI P W, ZHANG Q, et al. Anti-idiotypic nanobody-phage based real-time immuno-PCR for detection of hepatocarcinogen aflatoxin in grains and feedstuffs[J]. Analytical Chemistry, 2014, 86(21): 10841-10846. DOI:10.1021/ac5029424.
[25] QIU Y L, HE Q H, XU Y, et al. Deoxynivalenol-mimic nanobody isolated from a naïve phage display nanobody library and its application in immunoassay[J]. Analytica Chimica Acta, 2015, 887: 201-208.DOI:10.1016/j.aca.2015.06.033.
[26] DE B P, RAHAOUI H, LEER R J, et al. Real-time PCR detection of Campylobacter spp.: a comparison to classic culturing and enrichment[J].Food Microbiology, 2015, 51: 96-100. DOI:10.1016/j.fm.2015.05.006.
[27] KIM H J, MCCOY M, GEE S J, et al. Noncompetitive phage antiimmunocomplex real-time polymerase chain reaction for sensitive detection of small molecules[J]. Analytical Chemistry, 2011, 83(1): 246.
[28] NAKANO S, MORIZANE Y, MAKISAKA N, et al. Development of a highly sensitive immuno-PCR assay for the measurement of α-galactosidase A protein levels in serum and plasma[J]. PLoS ONE,2013, 8(11): e78588-e78588. DOI:10.1371/journal.pone.0078588.
[29] ZHANG H, XU Y, HUANG Q, et al. Natural phage nanoparticlemediated real-time immuno-PCR for ultrasensitive detection of protein marker[J]. Chemical Communications, 2013, 49(36): 3778-3780.DOI:10.1039/c3cc40688a.
[30] GUAN D, LI P, CUI Y, et al. A competitive immunoassay with a surrogate calibrator curve for aflatoxin M1 in milk[J]. Analytica Chimica Acta, 2011, 703(1): 64-69. DOI:10.1016/j.aca.2011.07.011.
Phage Displayed Nanobody Mediated Green Immuno-PCR for Detection of Deoxynivalenol
江东健, 罗秀儿, 何庆华. 基于噬菌体展示纳米抗体的绿色免疫PCR检测脱氧雪腐镰刀菌烯醇[J]. 食品科学, 2019,40(8): 256-261. DOI:10.7506/spkx1002-6630-20171128-337. http://www.spkx.net.cn
JIANG Dongjian, LUO Xiu’er, HE Qinghua. Phage displayed nanobody mediated green immuno-PCR for detection of deoxynivalenol[J]. Food Science, 2019, 40(8): 256-261. (in Chinese with English abstract) DOI:10.7506/spkx1002-6630-20171128-337. http://www.spkx.net.cn