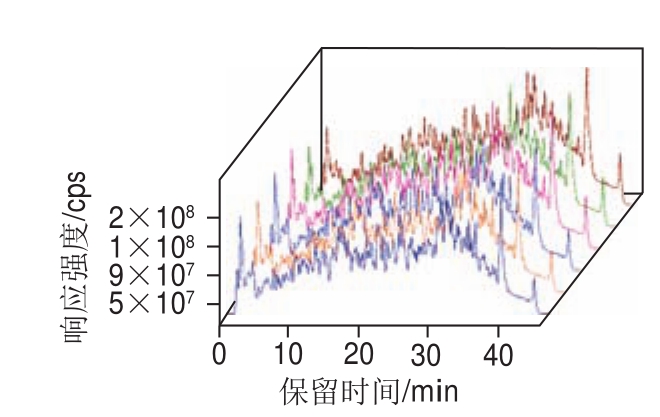
图 1 中国对虾有代表性的总离子流色谱图
Fig. 1 Representative total ion chromatogram (TIC) of F. chinensis
中国对虾(Fenneropenaeus chinensis)又称东方对虾,隶属于节肢动物门、甲壳纲、十足目、对虾科、对虾属,是我国重要的经济养殖虾类之一[1]。中国对虾主要分布于我国黄海和渤海海域[2],不仅产量丰富,而且具有营养价值高、保健功能强等特点,深受广大消费者的青睐[3-5]。中国对虾是中国和朝鲜半岛重要的水产品之一[6],同时也是我国虾类出口的主要产品[7]。随着水产养殖的发展,中国对虾发挥着越来越重要的作用。但是养殖和海捕的中国对虾在风味、口感和商业价值上存在一定差异。海捕中国对虾较养殖中国对虾鲜味浓,口感也有所不同[8]。海捕中国对虾的价格也明显高于养殖中国对虾的价格,为谋求利益,养殖对虾代替海捕对虾的现象时常发生[9-10]。因此有必要开发一种简便、快速、准确区分海捕和养殖中国对虾的方法。
目前,国内外已有一些用于区分海捕和养殖对虾的方法。20世纪80年代研究者以感官方法和解剖对比区分海捕和养殖的中国对虾[11],但对于普通消费者来说很难通过感官方法区分海捕和养殖的中国对虾。林洪等[12]研究表明肌动球蛋白溶解度、ATPase活性、疏水性可作为鉴别海捕对虾和养殖对虾的指标。薛长湖等[13]研究表明对虾肌肉蒸煮后的失水率和脂肪酸中C18:2ω6含量可用于区分海捕和养殖中国对虾。Ortea等[14]通过砷、铅、铬等重金属等多元统计分析区分海捕和养殖的对虾。Kim等[15]利用稳定性同位素性δ13C和δ15N区分海捕和养殖的对虾。
与上述方法相比,以质谱为基础的蛋白组学方法在稳健性、灵敏度、选择性、多路复用和高通量方面具有明显的优势,迅速发展成为一种强有力的食品认证工具,这一点从不断增加的物种识别、食品掺假和食品生产方法(野生/养殖)的研究中得到证明[16-19]。到目前为止,使用蛋白组学方法区分海捕和养殖对虾的研究仍然受限。但随着定量蛋白组学和质谱的发展,采集所有理论碎片离子(sequential window acquisition of all theoretical fragment ions,SWATH)技术应运而生。SWATH蛋白组学技术作为一种无标记的蛋白定量方法,可以同时对上千种蛋白进行平行量化[20],为复杂食品中蛋白定量提供更高的精确度和重现性[21-22]。由于组学研究通常涉及大量的样本和变量,因此有必要采用化学计量学方法提取有效信息并建立样本之间的关系[23]。
本研究以超高效液相色谱-飞行时间质谱(ultra-high performance liquid chromatography coupled to quadrupole time of flight-mass spectrometry,UPLC-Q TOF-MS)为分析工具,运用比较定量蛋白组学结合化学计量学寻找区分海捕和养殖中国对虾的蛋白生物标志物,为区分海捕和养殖中国对虾提供一定的技术支持。
中国对虾由中国海洋大学食品科学与工程学院提供,经鉴定为中国对虾(Fenneropenaeus chinensis)。
测序级胰蛋白酶(比活力18 523 U/mg) 美国Promega公司;质谱级甲酸(纯度>98%) 瑞士Fluka公司;乙腈 美国Fisher公司;碘代乙酰胺 美国Sigma公司;二硫苏糖醇、尿素、碳酸氢铵、盐酸 国药集团化学试剂有限公司。
AB SCIEX Triple TOF® 5600 MS仪 美国SCIEX公司;Nexera X2 30A UPLC仪 日本岛津公司;MQS50001型超纯水系统 美国Millipore公司;HH-4型数显恒温水浴锅 常州国华电器有限公司;AB135-S型精密电子分析天平 瑞士Mettler-Toledo公司。
1.3.1 中国对虾蛋白提取和酶解
海捕和养殖的中国对虾分别取6 个完整个体,用分析磨床在液氮浴中研磨成粉末。各取1 g研磨成粉末的样品,分别用10 mL蛋白提取液(8 mol/L尿素,50 mmol/L NH4HCO3溶液),垂直振荡30 min提取蛋白,高速低温离心20 min(4 ℃、15 000 r/min)。参考Wiśniewski[24]和Mi Rui[25]等方法,各取上清液100 μL,分别加入2 µL 1 mol/L二硫基苏糖醇在60 ℃反应1 h。取5 µL 1 mol/L现配的碘代乙酰胺,加入到已经冷却至室温的上述反应液中,室温避光反应1 h,实验重复3 次。采用10 kDa截留分子质量超滤离心管在15 000 r/min离心超滤20 min后,每次用200 µL 50 mmol/L NH4HCO3溶液洗涤膜上层蛋白,共洗涤3 次。最后在膜上层加入200 µL 50 mmol/L NH4HCO3溶液作为缓冲液并按酶和底物比1∶50将胰蛋白酶溶液加入到上述蛋白溶液中,混匀并37 ℃酶解16 h,使酶解彻底。超滤离心管15 000 r/min离心超滤20 min,收集下层的肽段滤液。
1.3.2 UPLC条件
色谱柱:安捷伦AdvanceBio Peptide Map column(150 mm×2.1 mm,130 Å,2.7 µm);柱温:40 ℃。流动相A:0.1%甲酸-乙腈,流动相B:0.1%甲酸溶液;流速:0.25 mL/min;进样量:30 μL;进样时间:46 min;流动相梯度:0~2 min(95% B),2~17 min(95%~80% B),27~37 min(85%~65% B),37~39 min(65%~20% B),39~42 min(20%~95% B),42~46 min(95% B)。
1.3.3 MS条件
采用正离子模式,喷雾电压5 500 V,电子电离源,扫描范围350~1 500 Da,正离子反应模式,雾化气GS1 35 psi,辅助加热器GS2 45 psi,气帘气压35 psi,喷雾电压5 500 eV,离子源温度500 ℃,解簇电压100 V,碰撞能量10 eV,信号强度阈值2×104。
样品SWATH采集:建立SWATH采集方法。在DDA模式下,在m/z 400~1 250范围内对混合多肽进行数据采集,3 次平行实验,PeakView 2.0处理获得子离子m/z和信号强度后,SWATH可变窗口计算器计算SWATH-MS动态采集窗口大小。其参数设置:目标窗口数量60 个,最小m/z 400,最大m/z 1 250,窗口重叠1 Da,CES 15,计算得数据采集系列窗口。高灵敏度模式下采集MS2谱图,用SWATH-MSALL方法对海捕和养殖中国对虾肽段样品在m/z 400~1 250范围进行SWATH数据采集,每组设置6 个生物学重复,每一样品重复采集SWATH数据2 次,采用随机数进样。
利用ProteinPilotTM软件进行数据处理和分析,选用NCBI的中国对虾数据库进行数据检索。ProteinPilot软件参数设置如下:搜库方法:Paragon method;酶切:胰蛋白酶;仪器:TripleTOF 5600;物种:无;ID Focus:Amino acid Subtitutions,Biological modifications;检索类型:ID;检测蛋白质阈值(Unused Protscore(conf)):大于0.05%(10.0%);提交错误发现率分析报告。
用PeakView和SWATH Micro App对采集的海捕和养殖中国对虾SWATH数据进行处理,对保留时间进行校正后,从鉴定蛋白库中提取可被SWATH定量的蛋白及其对应的肽段和碎片离子信息。SWATH数据处理参数设置:每一蛋白至少6 条可定量多肽,每一多肽至少6 个碎片离子。
用MarkerViewTM软件对样品进行归一化处理,设置:最小保留时间2 min,最大保留时间40 min;设置数据组的分类标志。
用SMICA软件对归一化后的数据进行主成分分析(principal component analysis,PCA)和正交偏最小二乘法判别分析(orthogonal partial least squares discriminant analysis,OPLS-DA),选取Pareto方法。

图 1 中国对虾有代表性的总离子流色谱图
Fig. 1 Representative total ion chromatogram (TIC) of F. chinensis
SWAHT-MS可采用稳健和完整的方式进行比较定量分析[26]。对海捕和养殖的中国对虾样本进行UPLC-Q TOF-MS检测,未见异常的总离子流色谱图。从图1可以看出,谱峰比较尖锐,峰形较为对称,样本的保留时间重现性好,分析系统稳定性好,为后续分析打下良好的基础。
使用ProteinPilotTM软件对得到的MS数据用中国对虾NCBI蛋白数据库进行谱库检索,共鉴定52 个蛋白、2 081 条肽段和14 775 个碎片。将检索得到的库作为数据库,并在PeakView软件将SWATH数据导入该数据库,按照1.4节的参数设置进行处理,并进行手工校正,最终保留35 个可相对定量的蛋白。

从图2A可以看出,海捕和养殖的中国对虾分居X轴的正半轴和负半轴,在t[1]上明显分离,且组间差异较大,组内差异较小。虽然样品点略显分散,但总体上可以很好地对2 个样品进行区分。从图2B可以看出,相比于PCA,OPLS-DA中样品点在X轴上的分布更加紧密,所以两两比较有监督的OPLS-DA可以更好地对海捕和养殖的中国对虾样品进行区分。
为进一步明确海捕和养殖中国对虾间具有统计学差异的蛋白,找到区分2 种不同来源中国对虾的蛋白生物标志物,利用以上建立的两两有监督的OPLS-DA模型进行识别。图3A为OPLS-DA模型对应的变量投影重要性(variable importance in projection,VIP)图,VIP值大于1可被认为是潜在生物标志物[27]。图3B为OPLS-DA模型对应的S图(S-plot),采用离原点越远表示对分类贡献越大的原则[28],选相关性p(corr)的绝对值大于0.8,寻找对样本分类具有重要贡献的蛋白生物标志物信息。图3C为OPLS-DA模型的因子载荷图,筛选标准为其Jack-knifed置信区间不应跨越0值[29-30]。通过筛选,共得到8 个候选的蛋白生物标志物,分别为肌动蛋白1、精氨酸激酶、β-肌动蛋白、血蓝蛋白、卵黄蛋白原、血蓝蛋白,部分、果糖1,6-二磷酸醛缩酶A和激活转录因子-2。

图 3 海捕和养殖中国对虾的蛋白生物标志物的筛选
Fig. 3 Screening for protein biomarkers of wild and farmed F. chinensis
此外,对海捕和养殖中国对虾的各个蛋白进行统计学分析(表1),P值小于0.05的具有显著统计学差异,在具有统计学差异的前提下,选取差异倍数大于1.2或者小于0.83的蛋白,作为最终的蛋白生物标志物。在排除精氨酸激酶、β-肌动蛋白、果糖1,6-二磷酸醛缩酶A后,肌动蛋白1、血蓝蛋白、卵黄蛋白原、血蓝蛋白,部分、激活转录因子-2被筛选为最终的蛋白生物标志物,用于区分海捕和养殖的中国对虾。
表 1 海捕和养殖的中国对虾统计分析
Table 1 Statistical analysis of wild and farmed F. chinensis

蛋白名称 P值 响应强度 差异倍数海捕 养殖胰岛素样雄激素受体 3.11×10-18 1 608.57±574.52 38 149.40±4 717.91 23.72丝氨酸/苏氨酸特异性蛋白激酶AKT 3.97×10-9 5 754.36±7 861.87 34 867.70±7 368.03 6.06逆酚氧化酶2 8.35×10-12 301 605.00±21 872.60 634 022.00±85 794.40 2.10法尼酸甲基转移酶 2.71×10-17 633 664.00±34 322.10 991 520.00±38 431.20 1.56胞浆锰超氧化物歧化酶 6.51×10-14 62 531.80±3 474.54 90 182.70±4 619.14 1.44丙糖磷酸异构酶 7.37×10-9 2 246 420.00±238 011.00 3 057 500.00±200 026.00 1.36 α2-巨球蛋白异构体3,部分 2.20×10-2 54 624.50±14 398.00 73 494.70±22 270.50 1.35血蓝蛋白 2.25×10-10 8 472 880.00±161 038.00 11 184 600.00±842 494.00 1.32精氨酸激酶 8.46×10-10 42 865 500.00±825 333.0051 126 300.00±2 682 280.00 1.19 β-肌动蛋白 1.62×10-10 24 861 800.00±840 190.00 28 758 000.00±872 055.00 1.16热休克蛋白90 7.00×10-3 110 538.00±5 555.32 120 876.00±10 682.90 1.09葡聚糖模式识别脂蛋白 2.25×10-1 3 140 720.00±353 748.00 3 409 540.00±656 776.00 1.09原肌球蛋白 8.95×10-567 504 100.00±1 663 350.0070 057 000.00±808 524.00 1.04磷酸丙酮酸水合酶 1.86×10-1 12 697 900.00±626 938.00 13 079 100.00±737 793.00 1.03肌钙蛋白I 6.61×10-2 11 639 100.00±403 024.00 11 382 300.00±221 582.00 0.98谷氨酰胺合成酶 4.96×10-1 3 960 450.00±307 081.00 3 831 610.00±566 192.00 0.97小热休克蛋白 2.79×10-1 487 998.00±63 614.90 464 358.00±37 499.60 0.95翻译调控肿瘤蛋白 9.72×10-3 210 758.00±9 287.77 191 195.00±22 061.80 0.91果糖1,6-二磷酸醛缩酶A 4.54×10-11 10 529 900.00±302 258.00 9 072 290.00±296 925.00 0.86谷氨酸脱氢酶 1.83×10-5 73 152.30±5 583.93 59 628.30±6 556.78 0.82硫氧还蛋白过氧化物酶 3.86×10-6 164 606.00±7 522.65 130 453.00±17 875.70 0.79热休克蛋白70同源蛋白 1.11×10-5 261 563.00±11 500.00 205 293.00±32 537.50 0.78 F1F0-ATP合酶β亚基 2.02×10-14 1 118 330.00±41 117.70 864 858.00±28 525.40 0.77葡萄糖调节蛋白78 7.32×10-6 95 553.20±7 300.00 69 317.40±13 784.80 0.73核小体核心组蛋白H2A,部分 3.93×10-4 146 029.00±30 805.50 101 233.00±20 783.30 0.69肌动蛋白1 1.15×10-1839 248 800.00±1 066 500.0026 399 400.00±1 186 320.00 0.67 Ras样核蛋白 3.29×10-14 53 911.50±2 817.06 35 055.20±2 568.74 0.65蛋白质二硫键异构酶2 6.81×10-10 191 643.00±13 864.80 119 123.00±20 013.20 0.62亲环素A 1.38×10-12 219 356.00±16 763.10 135 785.00±11 483.90 0.62围食膜因子 5.15×10-2 15 113.40±1 484.11 7 446.18±12 811.10 0.49核小体核心组蛋白H4,部分 2.43×10-16 426 167.00±28 212.00 203 525.00±21 648.00 0.48激活转录因子-2 1.82×10-15 1 399 870.00±180 647.00 336 586.00±48 144.60 0.24转座酶 9.41×10-22 138 529.00±6 962.48 15 507.00±8 497.74 0.11卵黄蛋白原 3.35×10-27 1 533 070.00±72 252.20 57 737.50±16 582.20 0.04血蓝蛋白,部分 1.28×10-26 1 474 900.00±78 052.30 10 308.50±5 208.850 0.01
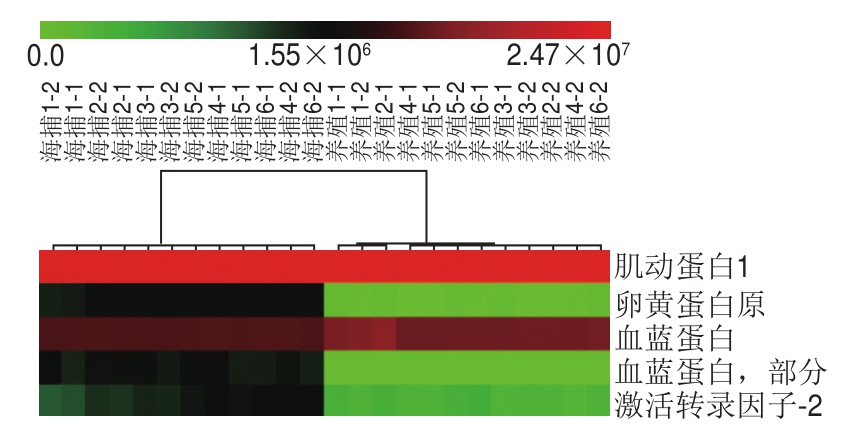
图 4 海捕和养殖中国对虾的层次聚类热力图
Fig. 4 Hierarchical cluster analysis and heatmap of wild and farmed F. chinensis
图4为海捕和养殖的中国对虾通过蛋白生物标志物筛选所得的层次聚类热力图(6 个平行实验,每个样品重复2 次),图中每个小方格代表该蛋白在该样品中的丰度,小方格的颜色越红表明蛋白丰度越大,颜色越绿表明蛋白丰度越小。从图4可以看出,海捕和养殖的中国对虾被分为2 个分支,说明通过筛选得到蛋白的生物标志物可以很好地区分这2 个样品。
本研究使用UPLC-Q TOF-MS SWATH技术为基础的蛋白组学,比较定量分析海捕和养殖中国对虾的蛋白含量。并结合化学计量学方法寻找生物标志物。OPLS-DA结果显示,该方法可以化复杂为简单,有效提取复杂变量间的主要信息,降低分析难度,并揭示海捕和养殖中国对虾样本之间蛋白丰度的差异。层次聚类分析表明,化学计量学筛选所得的生物标志物可以很好地区分海捕和养殖的中国对虾。与传统方法相比,本方法灵敏度更高、精确度更好。
本研究找到了区分海捕和养殖中国对虾的蛋白生物标志物,为区分海捕和养殖的中国对虾提供一种新的研究思路。该思路也可应用到其他食品的真实性分析、产地溯源、物种鉴定等领域,为分析高度同源的蛋白质食品提供一定的参考价值。
[1] 陈晓玲, 王君霞, 邱亮, 等. 日照地区中国明对虾急性肝胰腺坏死病(AHPND)的调查与分析[J]. 科学养鱼, 2018(1): 62-64.DOI:10.14184/j.cnki.issn1004-843x.2018.01.038.
[2] 王陌桑. 中国对虾增殖放流群体溯源分析及迁徙动态研究[D].上海: 上海海洋大学, 2016.
[3] 陈建林. 利用中国对虾加工副产物生产重组虾肉的研究[D]. 保定:河北农业大学, 2014.
[4] 侯晓荣. 中国对虾的解冻方式研究和调理制品研制[D]. 杭州: 浙江大学, 2014.
[5] 李姣, 李学鹏, 励建荣, 等. 冷藏条件下中国对虾肌肉蛋白质的生化特性[J]. 食品科学, 2011, 32(5): 16-21.
[6] CHA H K, OH C W, HONG S Y, et al. Reproduction and population dynamics of Penaeus chinensis (Decapoda: Penaeidae) on the western coast of Korea, Yellow Sea[J]. Fisheries Research, 2002, 56(1): 25-36.DOI:10.1016/S0165-7836(01)00310-1.
[7] 陈丽花, 肖作兵, 周培根. 中国对虾蛋白质的营养价值评价[J]. 上海海洋大学学报, 2009, 18(1): 88-93.
[8] 楼伟风, 李爱杰, 徐家敏. 中国对虾(Penaeus orientalis Kishiouys)粗蛋白、氨基酸含量的比较分析[J]. 中国海洋大学学报(自然科学版), 1989(S2): 69-79.
[9] PASCOAL A, BARROS-VELÁZQUEZ J, ORTEA I, et al. Molecular identification of the black tiger shrimp (Penaeus monodon), the white leg shrimp (Litopenaeus vannamei) and the Indian white shrimp (Fenneropenaeus indicus) by PCR targeted to the 16S rRNA mtDNA[J]. Food Chemistry, 2011, 125(4): 1457-1461. DOI:10.1016/j.foodchem.2010.10.053.
[10] WANNA W, CHOTIGEAT W, PHONGDARA A. Sequence variations of the first ribosomal internal transcribed spacer of Penaeus species in Thailand[J]. Journal of Experimental Marine Biology &Ecology, 2006, 331(1): 64-73. DOI:10.1016/j.jembe.2005.10.008.
[11] 崔维兴. 人工养殖与海捕对虾(Penaeus orientalis Kishinouye)鉴别特征初探[J]. 河北渔业, 1986(3): 12-19
[12] 林洪, 姜凤英, JAMIL K, 等. 养殖和海捕对虾加热后蛋白特性变化的比较研究[J]. 水产学报, 1998, 22(2): 143-147.
[13] 薛长湖, 曹杨, 陈修白, 等. 养殖对虾与海捕对虾鉴别方法初探[J]. 中国海洋大学学报(自然科学版), 1993(1): 101-106.
[14] ORTEA I, GALLARDO J M. Investigation of production method,geographical origin and species authentication in commercially relevant shrimps using stable isotope ratio and/or multielement analyses combined with chemometrics: an exploratory analysis[J]. Food Chemistry, 2015, 170(1): 145-153. DOI:10.1016/j.foodchem.2014.08.049.
[15] KIM H, KUMAR K S, HWANG S Y, et al. Utility of stable isotope and cytochrome oxidase I gene sequencing analyses in inferring origin and authentication of hairtail fish and shrimp[J]. Journal of Agricultural& Food Chemistry, 2015, 63(22): 5548-5556. DOI:10.1021/acs.jafc.5b01469.
[16] ORTEA I, O’CONNOR G, MAQUET A. Review on proteomics for food authentication[J]. Journal of Proteomics, 2016, 147(6): 212-225.DOI:10.1016/j.jprot.2016.06.033.
[17] NICOLA G, DI GIUSEPPE A M A, MARTINA L, et al. Myoglobin as marker in meat adulteration: a UPLC method for determining the presence of pork meat in raw beef burger[J]. Food Chemistry, 2013,141(3): 1814-1820.
[18] PIOVESANA S, CAPRIOTTI A L, CARUSO G, et al. Labeling and label free shotgun proteomics approaches to characterize muscle tissue from farmed and wild gilthead sea bream (Sparus aurata)[J].Journal of Chromatography A, 2016, 1428: 193-201. DOI:10.1016/j.chroma.2015.07.049.
[19] BUCKLEY M, FRASER S, HERMAN J, et al. Species identification of archaeological marine mammals using collagen fingerprinting[J].Journal of Archaeological Science, 2014, 41(2): 631-641. DOI:10.1016/j.jas.2013.08.021.
[20] SIMBRGER J M B, DETTMER K, OEFNER P J, et al. Optimizing the SWATH-MS-workflow for label-free proteomics[J]. Journal of Proteomics, 2016, 145: 137-140. DOI:10.1016/j.jprot.2016.04.021.
[21] GILLET L C, NAVARRO P, TATE S, et al. Targeted data extraction of the MS/MS spectra generated by data-independent acquisition: a new concept for consistent and accurate proteome analysis[J]. Molecular & Cellular Proteomics, 2012, 11(6): 1-7. DOI:10.1074/mcp.O111.016717.
[22] HU L P, ZHANG H W, ZHANG X M, et al. Identification of peptide biomarkers for discrimination of shrimp species through SWATH-MS-based proteomics and chemometrics[J]. Journal of Agricultural and Food Chemistry, 2018, 66(40): 10567-10574. DOI:10.1021/acs.jafc.8b04375.
[23] 张玥. 吉林省大米产地溯源分析的研究[D]. 长春: 吉林农业大学, 2015.
[24] WISNIEWSKI J R, ZOUGMAN A, NAGARAJ N, et al. Universal sample preparation method for proteome analysis[J]. Nature Methods,2009, 6(5): 359-362. DOI:10.1038/nmeth.1322.
[25] MI R, SUN Y, LI J, et al. Immune-related proteins detected through iTRAQ-based proteomics analysis of intestines from Apostichopus japonicus in response to tussah immunoreactive substances[J].Fish & Shellfish Immunology, 2018, 74: 436-443. DOI:10.1016/j.fsi.2018.01.002.
[26] VOWINCKEL J, CAPUANO F, CAMPBELL K, et al. The beauty of being (label)-free: sample preparation methods for SWATH-MS and next-generation targeted proteomics[J]. F1000 Research, 2013, 2: 272.DOI:10.12688/f1000research.2-272.v1.
[27] 赵小梅, 刘歆颖, 续畅, 等. 基于LC-MS代谢组学的雷公藤多苷致肝毒性生物标志物的初步筛查[J]. 中国中药杂志, 2015, 40(19): 3851-3858.
[28] 牟菲. 丹参-降香对大鼠心肌缺血/再灌注损伤的保护作用及机制的代谢组学研究[D]. 西安: 第四军医大学, 2017.
[29] YU Q, WU W, TIAN X, et al. Unraveling proteome changes of Holstein beef M. semitendinosus and its relationship to meat discoloration during post-mortem storage analyzed by label-free mass spectrometry[J]. Journal of Proteomics, 2017, 154: 85-93. DOI:10.1016/j.jprot.2016.12.012.
[30] WIKLUND S, JOHANSSON E, SJÖSTRÖM L, et al. Visualization of GC/TOF-MS-based metabolomics data for identification of biochemically interesting compounds using OPLS class models[J].Analytical Chemistry, 2008, 80(1): 115-122. DOI:10.1021/ac0713510.
Discrimination of Wild and Farmed Fenneropenaeus chinensis Using Proteomics and Chemometrics
薛长湖(1964—)(ORCID: 0000-0002-6626-2447),男,教授,博士,研究方向为水产品加工技术。E-mail: xuech@ouc.edu.cn
张晓梅(1981—)(ORCID: 0000-0002-1778-0504),女,高级工程师,博士,研究方向为蛋白组学及食品真实性分析。E-mail: zhangxm@aoacchina.org
胡玲萍, 张鸿伟, 张峰, 等. 利用蛋白组学和化学计量学区分海捕和养殖的中国对虾[J]. 食品科学, 2019, 40(8): 270-274.