沙门氏菌(Salmonella)是一种无芽孢、无荚膜的肠杆菌科革兰氏阴性直杆菌,广泛存在于人和动物肠道中,是全球范围导致食源性疾病爆发的重要病原菌之一[1-3]。在美国等发达国家,沙门氏菌感染的年发病率高达15.4%[4],由沙门氏菌引起的疾病爆发和住院治疗均高于其他食源性细菌[5]。在中国,每年约3亿人次因感染沙门氏菌而患病,达病原菌食源性疾病总数的70%~80%,给食品安全和消费者健康带来严重危害[6-7]。
质粒是独立于染色体外、可自主复制、通常携带多种抗性和毒力基因的闭合环状双链DNA,质粒的存在可使宿主菌产生耐药性和致病性等附加特性[8]。其中,接合质粒是一种与耐药基因水平传递和细菌耐药性获得的重要可移动元件[9]。在众多质粒中,IncI1型质粒有助于blaCTX-M-1基因在禽类和人之间散播[10],IncN型质粒已被欧洲多个国家的研究者报道携带blaCTX-M-1基因[11-12]。此外,研究还发现IncI1质粒有时还可与IncN质粒共同参与blaCTX-M-1耐药基因的转移,无论是IncI1还是IncN质粒,在分离自禽类粪便的大肠杆菌、分离自零售肉类和动物性产品的沙门氏菌中均有检出[13-14],且该2 种不相容型质粒往往被证明与某种特定耐药基因的传播密切相关。然而,我国关于此方面的研究还相对较少。
本研究以分离于我国部分省市的食源、人源和动物源沙门氏菌为材料,筛选携带IncI1和IncN质粒的菌株,阐明IncI1和IncN质粒在沙门氏菌中的流行状况,质粒阳性菌株的药敏性、与(氟)喹诺酮抗生素耐药相关基因和超广谱β-内酰胺酶(extended spectrum β-lactamases,ESBLs)编码基因的检出情况以及该2 种质粒通过水平转移传播耐药基因和耐药性的水平,以期为保障微生物食品安全问题提供参考。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株
表1 菌株信息
Table 1 Information about the isolates used in this study
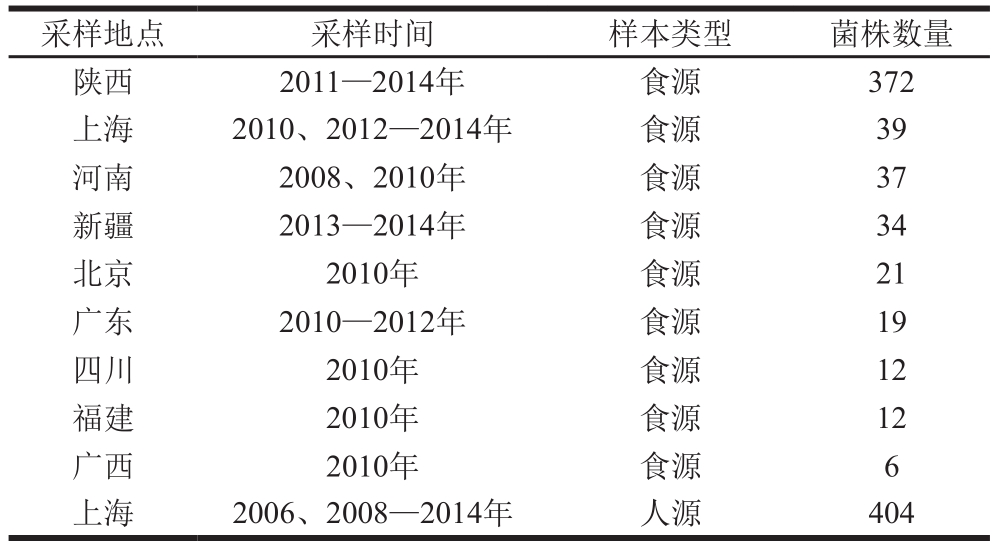
采样地点 采样时间 样本类型 菌株数量陕西 2011—2014年 食源 372上海 2010、2012—2014年 食源 39河南 2008、2010年 食源 37新疆 2013—2014年 食源 34北京 2010年 食源 21广东 2010—2012年 食源 19四川 2010年 食源 12福建 2010年 食源 12广西 2010年 食源 6上海 2006、2008—2014年 人源 404
所用菌株为实验室保存的分离于北京、上海、福建、河南、四川、广东、广西、陕西和新疆等地各类零售食品、临床病人和食品性动物的沙门氏菌(n=956)以及香港理工大学惠赠的携带IncI1(n=12)和IncN(n=2)的人源性沙门氏菌[15-18](表1)。药敏性测定用标准质控菌株Escherichia coli ATCC 25922和Enterococcus faecalis ATCC 29212为中国药品生物制品检定研究院惠赠。
1.1.2 培养基
Luria-Bertani(LB)琼脂、LB肉汤、Mueller Hinton琼脂、麦康凯琼脂 北京陆桥技术股份责任公司;XLT4培养基及其补充液 美国BD公司。
1.1.3 抗生素
药敏性测定用抗生素:萘啶酮酸(nalidixic acid,NAL)、环丙沙星(ciprofloxacin,CIP)、阿米卡星(amikacin,AMK)、硫酸庆大霉素(gentamicin,GEN)、链霉素(streptomycin,STR)、卡那霉素(kanamycin,KAN)、头孢曲松(ceftriaxone,CRO)、头孢西丁(cefoxitin,FOX)、头孢噻呋(ceftiofur,TIO)、头孢哌酮(cefoperazone,PER)、氨苄西林(ampicillin,AMP)、阿莫西林/克拉维酸(amoxicillin-clavulanic acid,AMC)、氯霉素(chloramphenicol,CHL)、四环素(tetracycline,TET)、复方新诺明(trimethoprim/sulfamethoxazole,SXT)和多黏菌素B(polymyxin B,PB)(均为分析纯) 美国Sigma公司。
1.1.4 聚合酶链式反应(polymerase chain reaction,PCR)引物
ESBLs编码基因(blaTEM、blaCTX-M、blaOXA、blaACC和blaPSE)和质粒介导的(氟)喹诺酮耐药相关基因(qnrA、qnrB、qnrS、acc(6’)-Ib和qepA)扩增引物,IncN和IncI1不相容群质粒PCR复制子分型用引物均由北京奥科鼎盛生物科技有限公司合成(表2)。
表2 PCR引物序列和产物大小
Table 2 Primers sequences used for PCR amplification and size of corresponding PCR products

目标基因 上游引物(5’-3’) 下游引物(5’-3’) 大小/bp IncI1 TCCTGATTTCATCCACGGCAA AGGGACATTATTTGCCCGCT 418 IncN CCAGAATGCCGACCCGAATG CATACGGCCAAATTTCGGGA 238 qnrA AGAGGATTTCTCACGCCAGG TGCCAGGCACAGATCTTGAC 580 qnrB GGMATHGAAATTCGCCACTG TTTGCYGYYCGCCAGTCGAA 264 qnrS GCAAGTTCATTGAACAGGGT TCTAAACCGTCGAGTTCGGCG 428 acc(6')-Ib TTGCGATGCTCTATGAGTGGCTA CTCGAATGCCTGGCGTGTTT 482 qepA CTGCAGGTACTGCGTCATG CGTGTTGCTGGAGTTCTTC 403 blaTEM ATGAGTATTCAACATTTCCG ACCAATGCTTAATCAGTGAG 964 blaCTX-M GAGTTTCCCCATTCCGTTTC CAGAATAAGGAATCCCATGGTT 909 blaOXA ACCAGATTCAACTTTCAA TCTTGGCTTTTATGCTTG 590 blaACC AGCCTCAGCAGCCGGTTAC GAAGCCGTTAGTTGATCCGG 818 blaPSE AATGGCAATCAGCGCTTCCC GGGGCTTGATGCTCACTACA 598
1.1.5 试剂
1 mol/L Tris-HCl(pH 8.0)、0.5 mol/L EDTA(pH 8.0)、5×TBE缓冲液、琼脂糖凝胶、dNTP、PCR buffer、MgCl2、rTaqTM DNA Polymerase、DL2000 DNA Marker 宝生物工程(大连)有限公司。
1.2 仪器与设备
Mycycler PCR仪、DNA电泳和凝胶成像系统 美国Bio-Rad公司;Milli-Q纯水仪 法国Millpore公司;磁力加热搅拌器 美国Fisher公司;微波炉 广州美的微波炉制造有限公司;-40 ℃低温冰箱、-80 ℃低温冰箱日本Sanyo公司;百分之一天平、万分之一天平 德国赛多利斯公司;立式压力蒸汽灭菌器 上海申安高压仪器设备有限公司;隔水式恒温培养箱 北京科伟实验仪器有限公司;移液器、高速离心机 德国Eppendorf公司;生物安全柜 美国Labgard公司;超净工作台苏州净化设备有限公司;浊度仪 美国DADE Behring公司;数显恒温水浴锅 北京科伟试验仪器有限公司;恒温摇床 上海智诚分析仪器制造有限公司。
1.3 方法
1.3.1 IncI1和IncN质粒分型
基于PCR复制子分型方法对质粒进行不相容群分析[19],用单一PCR法筛选携带IncI1和IncN不相容群质粒的菌株。采用煮沸法制备DNA模板[20]。PCR条件:94 ℃预变性10 min;94 ℃变性1 min,相应退火温度条件下1 min、72 ℃延伸1 min,35 个循环;72 ℃延伸10 min。退火温度由相应基因的引物序列决定。2 μL PCR产物经凝胶电泳和溴化乙锭染色后于凝胶成像系统下检测。在低温条件下将PCR粗产物送至上海桑尼生物科技有限公司测序,采用在线比对软件BLAST(http://www.ncbi.nlm.nih.gov/BLAST/)进行比对,确定质粒类型。
1.3.2 药敏性测定
采用美国国家临床实验室标准化委员会(Clinical and Laboratory Standards Institution,CLSI)[21]推荐使用的琼脂稀释法,测定供试抗生素对沙门氏菌的最小抑菌浓度(minimum inhibitory concentrations,MIC),参考CLSI的标准解读药敏性测定结果。
1.3.3 耐药基因检测
采用普通PCR检测相关耐药基因,引物如表2所示,PCR模板(菌株总DNA)、扩增体系和具体条件如1.3.1节所示。
1.3.4 质粒膜接合实验
表3 接合用菌株信息
Table 3 Information about the isolates used for conjugation
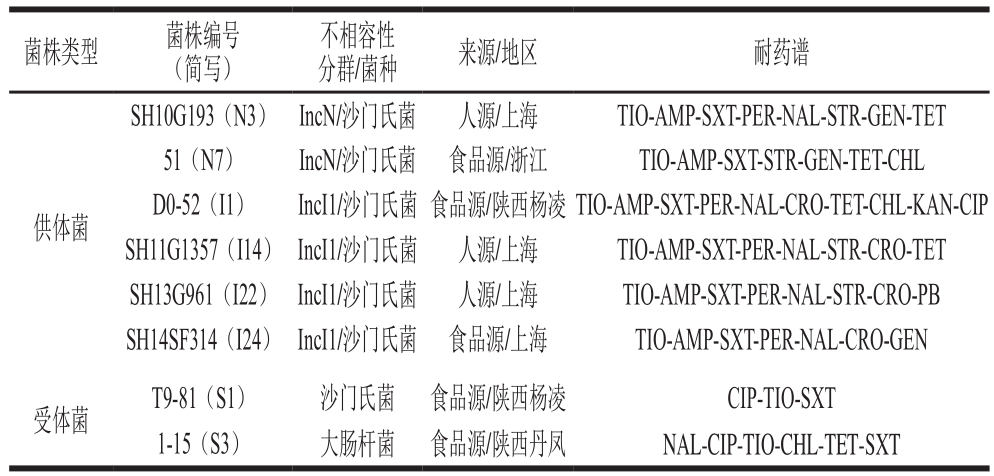
菌株类型 菌株编号(简写)不相容性分群/菌种 来源/地区 耐药谱供体菌SH10G193(N3) IncN/沙门氏菌 人源/上海 TIO-AMP-SXT-PER-NAL-STR-GEN-TET 51(N7) IncN/沙门氏菌 食品源/浙江 TIO-AMP-SXT-STR-GEN-TET-CHL D0-52(I1) IncI1/沙门氏菌 食品源/陕西杨凌 TIO-AMP-SXT-PER-NAL-CRO-TET-CHL-KAN-CIP SH11G1357(I14) IncI1/沙门氏菌 人源/上海 TIO-AMP-SXT-PER-NAL-STR-CRO-TET SH13G961(I22) IncI1/沙门氏菌 人源/上海 TIO-AMP-SXT-PER-NAL-STR-CRO-PB SH14SF314(I24) IncI1/沙门氏菌 食品源/上海 TIO-AMP-SXT-PER-NAL-CRO-GEN受体菌 T9-81(S1) 沙门氏菌 食品源/陕西杨凌 CIP-TIO-SXT 1-15(S3) 大肠杆菌 食品源/陕西丹凤 NAL-CIP-TIO-CHL-TET-SXT
基于IncI1和IncN质粒阳性菌株及受体菌药敏性结果,选择分别携带IncI1质粒和IncN质粒的沙门氏菌作为供体菌,不含该2 种质粒的大肠杆菌和沙门氏菌为受体菌,采用膜过滤法进行接合,接合用菌株相关信息如表3所示。接合方法如下:取OD600 nm为0.6的供体菌和受体菌菌液各1 mL于5 mL LB肉汤中混匀,转入孔径0.22 μm的滤器过滤,过滤后将滤膜紧贴于不含抗生素的LB琼脂培养基表面,有菌的一面向上,37 ℃过夜培养。用5 mL LB肉汤清洗过夜培养的滤膜,梯度稀释后分别涂布于同时含有CRO(32 μg/mL)和CIP(16 μg/mL)或同时含有STR(64 μg/mL)和CIP(16 μg/mL)的LB平板上,双抗平板的选择根据供体菌和受体菌的耐药表型确定,每个梯度3 个平行,37 ℃培养48 h,计数。将OD600 nm为0.6的受体菌梯度稀释后均匀涂布于不含抗生素的LB平板,每个梯度3 个平行,37 ℃培养过夜,计数,用于菌液起始浓度的确定。同时,将供体菌和受体菌分别涂布于相应的双抗平板上,每株菌3 个平行,37 ℃培养48 h,作为对照。培养结束后,确定供体菌和受体菌不能在相应的双抗平板上生长时,再在涂布有不同稀释度接合子的双抗平板上随机挑选10 个单菌落,以表2中的引物进行PCR扩增,扩增产物采用1%的琼脂糖凝胶进行电泳和测序分析,确定IncI1和IncN质粒在接合过程发生了水平转移。接合子携带的耐药基因和药敏性检测方法同1.3.1节和1.3.2节。接合频率计算公式如下:
式中:T为阳性接合子平均菌落数;R为受体菌平均菌落数。
1.4 数据统计分析
利用Minitab®18.1对实验数据进行处理,用χ2检验比较不同条件下的检出率,P<0.05,差异显著。
2 结果与分析
2.1 IncI1和IncN质粒分型
表4 IncN和IncI1质粒阳性菌株筛选结果
Table 4 Screening results of IncN and IncI1 plasmid positive isolates
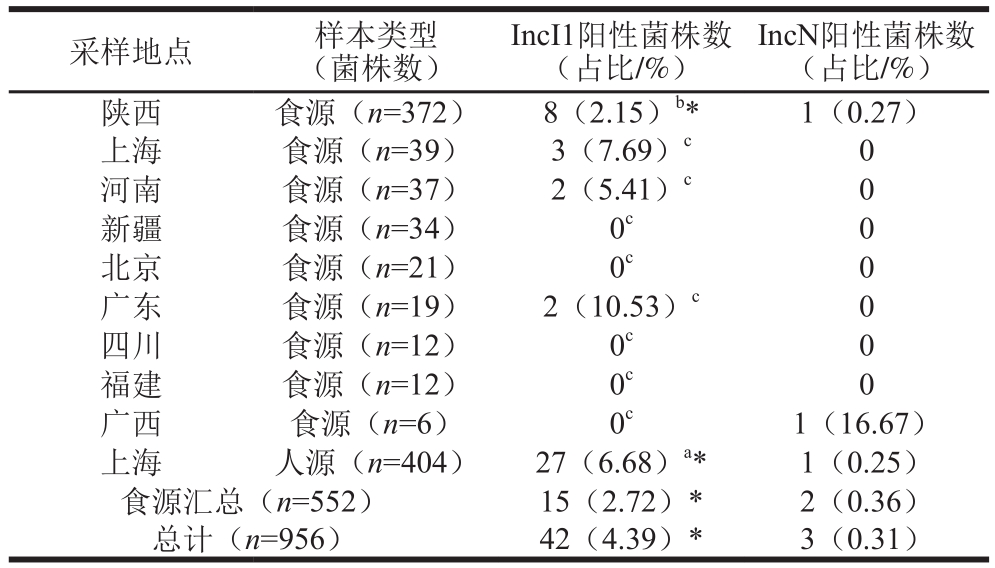
注:同列字母不同表示差异显著(P<0.05),下同;*.同行差异显著(P<0.05)。
采样地点 样本类型(菌株数)IncI1阳性菌株数(占比/%)IncN阳性菌株数(占比/%)陕西 食源(n=372) 8(2.15)b* 1(0.27)上海 食源(n=39) 3(7.69)c 0河南 食源(n=37) 2(5.41)c 0新疆 食源(n=34) 0c 0北京 食源(n=21) 0c 0广东 食源(n=19) 2(10.53)c 0四川 食源(n=12) 0c 0福建 食源(n=12) 0c 0广西 食源(n=6) 0c 1(16.67)上海 人源(n=404) 27(6.68)a* 1(0.25)食源汇总(n=552) 15(2.72)* 2(0.36)总计(n=956) 42(4.39)* 3(0.31)
956 株沙门氏菌中,IncI1质粒阳性菌的检出率(n=42,4.39%)显著(P<0.05)高于IncN质粒阳性菌的检出率(n=3,0.31%)。人源性沙门氏菌中共检出27 株(27/404,6.68%)IncI1阳性菌,显著(P<0.05)高于食源性沙门氏菌中IncI1(15/552,2.72%)阳性菌检出率。陕西食源沙门氏菌中IncI1质粒阳性菌的检出率显著(P<0.05)高于其他省市(表4)。
2.2 药敏性和耐药基因检测结果
基于不同采样时间,采样省份或同一省份不同采样地点,分离沙门氏菌的食品类型,病人的年龄、性别、患病类型和医院名称,前期通过脉冲场凝胶电泳结合XbaI限制性内切酶对IncI1和IncN质粒阳性菌株分型的DNA带谱类型结果,尽量选取来源和基因型不同的菌株作为进一步实验的材料。本研究从45 株IncI1和IncN阳性沙门氏菌中挑选出26 株代表性菌株(包括香港理工大学惠赠的2 株IncN质粒阳性沙门氏菌)用于药敏性和耐药基因检测。菌株对TIO(100.0%)耐药率最高,其次为对NAL(92.3%)、AMP(92.3%)、CFP(88.5%)、TET(84.6%)、CRO(80.8%)、SXT(76.9%)、STR(76.9%)和CHL(61.5%);对KAN(26.9%)、GEN(23.1%)、PB(23.1%)、CIP(19.2%)、AMC(15.4%)、FOX(11.5%)和AMK(3.8%)耐药率较低(表4)。除TET(85.7%)外,携带IncI1型质粒的菌株对TIO(100.0%)、NAL(100.0%)、AMP(100.0%)、CFP(95.2%)和CRO(95.2%)的耐药率显著(P<0.05)高于对其他10 种抗生素的耐药率。IncI1质粒阳性菌株比IncN质粒阳性菌株的耐药谱更宽,IncN型质粒阳性菌株均对PB、AMC、FOX和AMK敏感,对部分氨基糖苷类药物,如STR和GEN的耐药率较IncI1型质粒阳性菌株高(表5)。
表5 药敏性检测结果Table 5 Results of antimicrobial susceptibility test
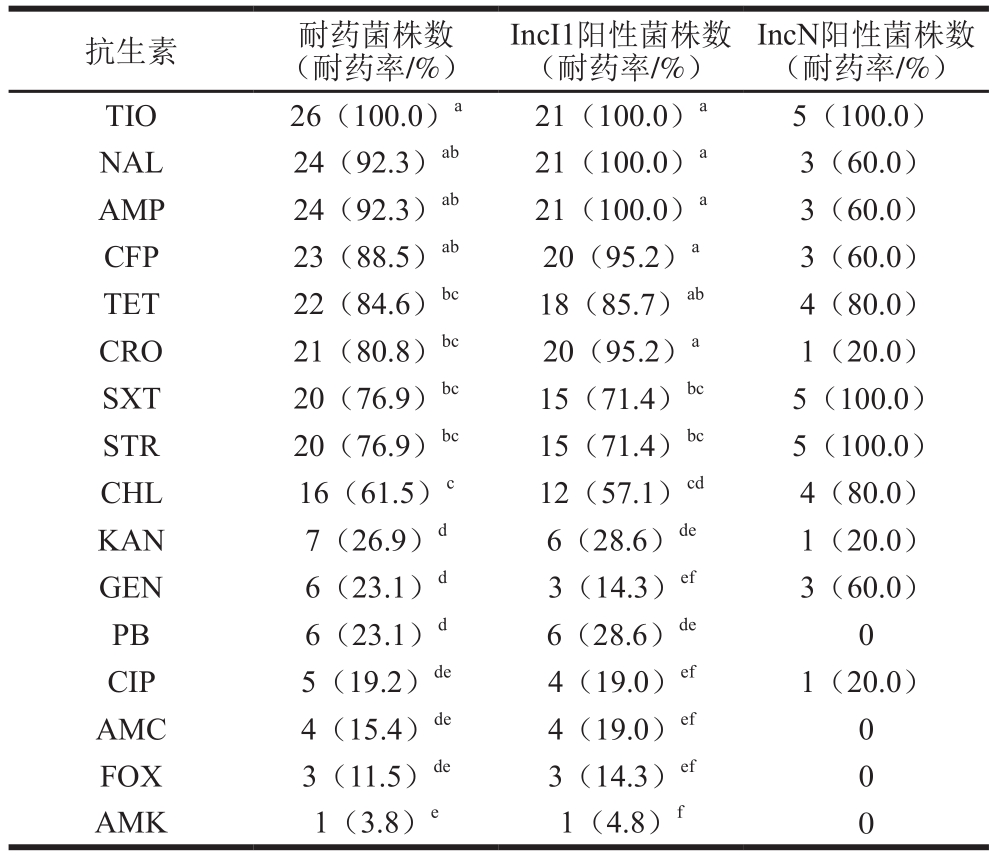
注:样本量小于5不具备统计意义,未进行差异性分析,下同。
抗生素 耐药菌株数(耐药率/%)IncI1阳性菌株数(耐药率/%)IncN阳性菌株数(耐药率/%)TIO 26(100.0)a 21(100.0)a 5(100.0)NAL 24(92.3)ab 21(100.0)a 3(60.0)AMP 24(92.3)ab 21(100.0)a 3(60.0)CFP 23(88.5)ab 20(95.2)a 3(60.0)TET 22(84.6)bc 18(85.7)ab 4(80.0)CRO 21(80.8)bc 20(95.2)a 1(20.0)SXT 20(76.9)bc 15(71.4)bc 5(100.0)STR 20(76.9)bc 15(71.4)bc 5(100.0)CHL 16(61.5)c 12(57.1)cd 4(80.0)KAN 7(26.9)d 6(28.6)de 1(20.0)GEN 6(23.1)d 3(14.3)ef 3(60.0)PB 6(23.1)d 6(28.6)de 0 CIP 5(19.2)de 4(19.0)ef 1(20.0)AMC 4(15.4)de 4(19.0)ef 0 FOX 3(11.5)de 3(14.3)ef 0 AMK 1(3.8)e 1(4.8)f 0
26 株IncI1和IncN阳性菌对5 种常见的质粒介导的喹诺酮抗性相关耐药基因和5 种ESBLs编码基因的携带状况检测结果如表6所示。未在IncN和IncI1质粒阳性菌株中检出qepA和blaACC,未在IncI1阳性菌中未检出qnrA和qnrB,未在IncN阳性菌株中检出blaCTX-M和blaPSE。IncI1质粒阳性菌株中,blaTEM和blaCTX-M基因的检出率均显著(P<0.05)高于其他基因。
表6 IncI1和IncN质粒阳性沙门氏菌耐药基因检出情况
Table 6 Prevalence of antibiotic resistance genes in IncI1 and IncN plasmids positive Salmonella
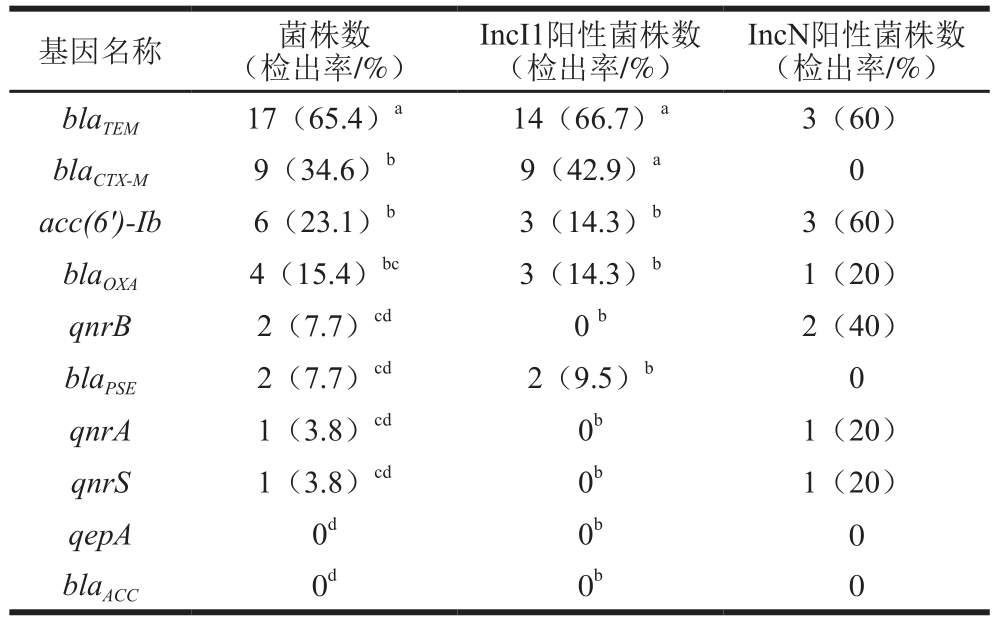
基因名称 菌株数(检出率/%)IncI1阳性菌株数(检出率/%)IncN阳性菌株数(检出率/%)blaTEM 17(65.4)a 14(66.7)a 3(60)blaCTX-M 9(34.6)b 9(42.9)a 0 acc(6')-Ib 6(23.1)b 3(14.3)b 3(60)blaOXA 4(15.4)bc 3(14.3)b 1(20)qnrB 2(7.7)cd 0 b 2(40)blaPSE 2(7.7)cd 2(9.5)b 0 qnrA 1(3.8)cd 0b 1(20)qnrS 1(3.8)cd 0b 1(20)qepA 0d 0b 0 blaACC 0d 0b 0
2.3 接合实验
4 株IncI1型质粒阳性和2 株IncN型质粒阳性供体菌与受体菌T9-81(Salmonella typhimurium)发生接合的接合频率在3.2×10-5~2.0×10—3之间,与大肠杆菌1-15的接合频率在8.7×10—7~9.6×10—5之间(表7)。接合过程中,IncI1质粒与IncN质粒均能从供体菌转移到受体,相对而言,IncI1质粒更容易通过接合作用转移。
通过PCR对供体、受体和接合子携带的耐药基因检测结果表明,除blaTEM基因在供体菌I14对应的2 种接合子I14-S1和I24-S3中未检出外,质粒携带的ESBLs编码基因(blaTEM和blaCTX-M)和(氟)喹诺酮耐药相关基因(qnrB和acc(6’)-Ib)在接合过程中均可从供体菌转移至受体菌。接合后部分接合子获得了供体菌质粒DNA编码的相应抗性表型,如KAN、STR、GEN、CRO等。部分受体菌株接受IncI1和IncN质粒后对特定抗生素的耐药表型与供体菌趋于一致。2 种不同类型的质粒发生转移后,对应的接合子对SXT和TET的耐受性变化趋势有所不同,表现出质粒的差异。从2大类接合子的药敏性变化趋势看,接合子对抗生素的耐受性变化与受体菌种属差异关系不大,受体菌种属的差异仅对接合频率有所影响。
表7 供体菌、受体菌和接合子的药敏性及耐药基因检测结果
Table 7 Antimicrobial susceptibility profiles and antibiotic resistance genes of donors, recipients and transconjugants
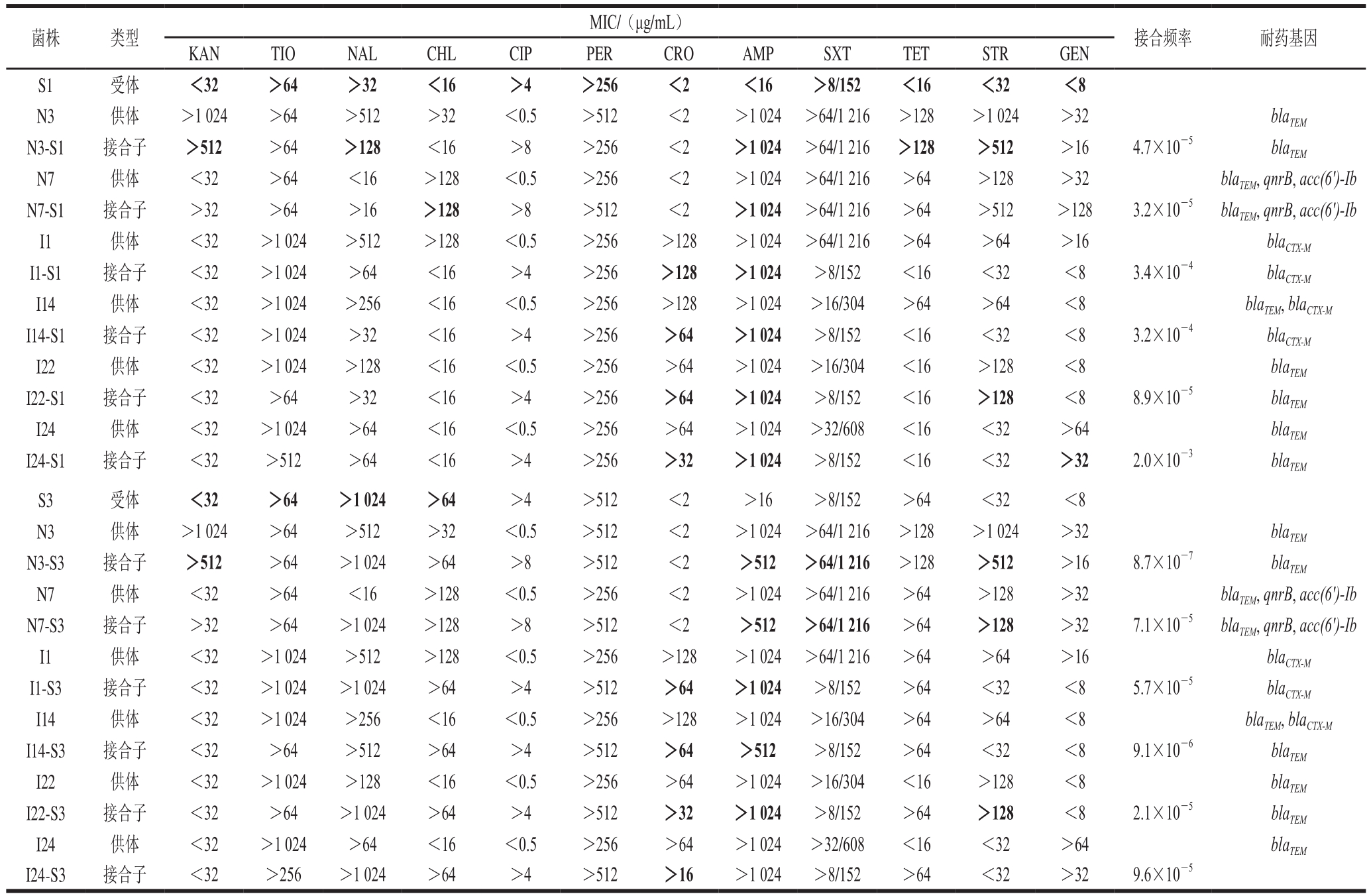
注:粗体表示菌株接合前后耐药性变化较大。
菌株 类型MIC/(μg/mL)接合频率 耐药基因KAN TIO NAL CHL CIP PER CRO AMP SXT TET STR GEN S1 受体 <32 >64 >32 <16 >4 >256 <2 <16 >8/152 <16 <32 <8 N3 供体 >1 024 >64 >512 >32 <0.5 >512 <2 >1 024 >64/1 216 >128 >1 024 >32 blaTEM N3-S1 接合子 >512 >64 >128 <16 >8 >256 <2 >1 024 >64/1 216 >128 >512 >16 4.7×10-5 blaTEM N7 供体 <32 >64 <16 >128 <0.5 >256 <2 >1 024 >64/1 216 >64 >128 >32 blaTEM, qnrB, acc(6')-Ib N7-S1 接合子 >32 >64 >16 >128 >8 >512 <2 >1 024 >64/1 216 >64 >512 >128 3.2×10-5 blaTEM, qnrB, acc(6')-Ib I1 供体 <32 >1 024 >512 >128 <0.5 >256 >128 >1 024 >64/1 216 >64 >64 >16 blaCTX-M I1-S1 接合子 <32 >1 024 >64 <16 >4 >256 >128 >1 024 >8/152 <16 <32 <8 3.4×10-4 blaCTX-M I14 供体 <32 >1 024 >256 <16 <0.5 >256 >128 >1 024 >16/304 >64 >64 <8 blaTEM, blaCTX-M I14-S1 接合子 <32 >1 024 >32 <16 >4 >256 >64 >1 024 >8/152 <16 <32 <8 3.2×10-4 blaCTX-M I22 供体 <32 >1 024 >128 <16 <0.5 >256 >64 >1 024 >16/304 <16 >128 <8 blaTEM I22-S1 接合子 <32 >64 >32 <16 >4 >256 >64 >1 024 >8/152 <16 >128 <8 8.9×10-5 blaTEM I24 供体 <32 >1 024 >64 <16 <0.5 >256 >64 >1 024 >32/608 <16 <32 >64 blaTEM I24-S1 接合子 <32 >512 >64 <16 >4 >256 >32 >1 024 >8/152 <16 <32 >32 2.0×10-3 blaTEM S3 受体 <32 >64 >1 024 >64 >4 >512 <2 >16 >8/152 >64 <32 <8 N3 供体 >1 024 >64 >512 >32 <0.5 >512 <2 >1 024 >64/1 216 >128 >1 024 >32 blaTEM N3-S3 接合子 >512 >64 >1 024 >64 >8 >512 <2 >512 >64/1 216 >128 >512 >16 8.7×10-7 blaTEM N7 供体 <32 >64 <16 >128 <0.5 >256 <2 >1 024 >64/1 216 >64 >128 >32 blaTEM, qnrB, acc(6')-Ib N7-S3 接合子 >32 >64 >1 024 >128 >8 >512 <2 >512 >64/1 216 >64 >128 >32 7.1×10-5 blaTEM, qnrB, acc(6')-Ib I1 供体 <32 >1 024 >512 >128 <0.5 >256 >128 >1 024 >64/1 216 >64 >64 >16 blaCTX-M I1-S3 接合子 <32 >1 024 >1 024 >64 >4 >512 >64 >1 024 >8/152 >64 <32 <8 5.7×10-5 blaCTX-M I14 供体 <32 >1 024 >256 <16 <0.5 >256 >128 >1 024 >16/304 >64 >64 <8 blaTEM, blaCTX-M I14-S3 接合子 <32 >64 >512 >64 >4 >512 >64 >512 >8/152 >64 <32 <8 9.1×10-6 blaTEM I22 供体 <32 >1 024 >128 <16 <0.5 >256 >64 >1 024 >16/304 <16 >128 <8 blaTEM I22-S3 接合子 <32 >64 >1 024 >64 >4 >512 >32 >1 024 >8/152 >64 >128 <8 2.1×10-5 blaTEM I24 供体 <32 >1 024 >64 <16 <0.5 >256 >64 >1 024 >32/608 <16 <32 >64 blaTEM I24-S3 接合子 <32 >256 >1 024 >64 >4 >512 >16 >1 024 >8/152 >64 <32 >32 9.6×10-5
3 讨论与结论
耐药沙门氏菌的广泛存在已经成为世界性公共卫生问题[22]。质粒介导的细菌耐药性不仅可以由亲代垂直传递给子代,而且可以在不同微生物间水平传播,成为养殖业、食品安全和人类健康的重大安全隐患[23-24]。基于PCR的复制子分型方法是研究质粒的不相容群和质粒传播耐药性的重要手段,目前,该方法已广泛应用于在人源、食源和动物源致病性细菌间传播耐药性的质粒分类研究[25-26]。
国内外关于质粒不相容群、质粒不相容群和耐药基因之间的关联性研究越来越多。表明IncI1和IncN质粒在分离自禽类粪便的大肠杆菌、零售肉类和动物产品的沙门氏菌中十分普遍[13-14],在中国流行的肠炎沙门氏菌具有头孢菌素抗性主要是由于其携带多种blaCTX-M基因的IncI1质粒传播[27]。IncN质粒在肠杆菌科细菌中也普遍存在,并被报道携带多种抗性决定簇,包括ESBLs和碳青霉烯酶等[28-29]。此外,携带不同种类耐药基因的可接合性IncN质粒也在源于世界范围内患者和环境样品的不同细菌中被广泛报道[30-32]。然而,该2 种不相容质粒大多集中在大肠杆菌和肺炎克雷伯菌中,关于沙门氏菌中IncN和IncI1质粒的流行状况及其介导的耐药性研究较少。
本研究对源自我国多个省市的人源性和食源性沙门氏菌进行IncI1和IncN不相容质粒阳性菌的筛查,但2 种质粒阳性菌的检出率均低于现有报道的结果[19,31-32]。导致结果差异的原因一方面可能由于现有报道的供试菌株基本分离于零售食品和病死的食品性动物,另一方面,研究中供试筛选菌株的数量也会导致检出率的差异。共筛查了956 株沙门氏菌,而现有研究的菌株数均较少。本研究还发现IncI1质粒在人源性沙门氏菌中的检出率(6.68%)显著(P<0.05)高于食源性沙门氏菌(2.72%),陕西食源沙门氏菌中IncI1质粒阳性菌的检出率显著(P<0.05)高于其他省市,这可能是由于其他省市菌株数量太少所导致。与此类似,其他研究在沙门氏菌和大肠杆菌中检出IncN阳性菌株的比例为18.2%~25.6%,IncI1阳性菌株的比例为7.7%~31.8%[19,33-34]。尽管IncN和IncI1质粒阳性菌株的检出率均不高,但由于该2 种质粒都是可接合性质粒,能通过细菌之间的接合作用介导耐药性在不同环境中传播[10-12],导致细菌耐药谱不断增宽,耐药性增强,因此需要引起相关部门足够的重视。
质粒通常携带多种抗性基因和毒力基因,赋予宿主耐药性和致病性等多种附加特性[8]。IncI1质粒携带β-内酰胺和IV型菌毛编码基因,可赋予菌株免受抗生素和毒力入侵的能力,比普通菌株具有更强的致病性。来自于人源和动物源的细菌已在英国、巴基斯坦和洪都拉斯等国家被多次报道携带含有blaCTX-M-15基因的IncI1质粒[35-36]。本研究在人源和食品源IncI1质粒阳性菌株中主要检出了blaTEM、blaCTX-M、acc(6’)-Ib、blaOXA和blaPSE基因。有研究表明,ESBLs常见型别编码基因通常由质粒介导,可在同种或不同种属的革兰氏阴性菌间频繁、广泛地转移[37]。方婷子[19]研究发现,沙门氏菌中IncI1质粒阳性菌(12.0%)均携带blaOXA-1;携带blaTEM-1基因的菌株携带IncHI2、IncP、IncI1和IncA/C等质粒。此外,本研究在IncN质粒阳性菌株中还检出acc(6’)-Ib、qnrB、blaOXA、qnrA和qnrS等与头孢菌素和(氟)喹诺酮类抗生素耐药相关基因。据报道,IncN型质粒是临床检出菌株中的主要质粒类型,也是携带blaCTX-M、blaNDM和qnr等耐药基因的主要质粒[38],也是促进blaCTX-M基因水平传播的常见流行质粒[11,39],IncI1质粒有时还会与IncN质粒共同参与blaCTX-M-1耐药基因的转移[13-14]。qnr、acc(6’)-Ib和ESBLs编码基因通常会位于同一质粒上,可通过接合、转座作用转移和传播,因此携带该类质粒的沙门氏菌可以被认为是一个潜在的耐药基因库,如果此类沙门氏菌在社区或食品生产链中传播,将会导致巨大的潜在危害[40]。本研究发现携带IncI1质粒的菌株比携带IncN质粒菌株的耐药谱更宽,对头孢类抗生素的耐受水平更高,而携带IncN质粒的菌株对氨基糖苷类抗生素的抗性则更高一些。由此可以推论,IncN质粒可能主要介导氨基糖苷类药物耐药,而IncI1型质粒主要介导头孢类抗生素耐药。当然,该2 类质粒介导的耐药性是否和本研究的推断结果一致,还需进行进一步研究。
通过耐药质粒传递耐药性是细菌中间最为常见也是最为重要的一种耐药机制。本研究中,供体菌与沙门氏菌的接合频率高于与大肠杆菌的接合频率。IncI1和IncN质粒的接合转移还赋予了受体菌对KAN、TIO、CRO、AMP、STR和GEN的耐药表型,与以前研究结果相似[41-42]。
已有研究表明,IncI1型质粒是人源和食品源大肠杆菌及沙门氏菌携带和传播β-内酰胺酶基因最常见的质粒之一,blaTEM、blaSHV、blaCTX-M、blaCMY和blaOXA基因通常会随IncI1质粒的转移而水平传播[43-44]。blaTEM和blaCTX-M可通过IncN和IncI1在禽类和人之间广泛传播[10,41],IncN质粒也被欧洲多个国家的研究者报道携带有blaCTX-M-1[11-12]。本研究中,blaCTX-M-55和blaCTX-M-3均被证明可随IncI1和IncN型质粒在接合过程从供体菌传递给受体菌。虽然blaTEM在大部分供体和接合子中均有检出,但在I14作为供体菌时均未在接合子中检出,表明IncI1质粒可能在转移过程没有将宿主细胞中的质粒完整地转移到受体菌,也可能在接合过程中部分基因丢失。当然,导致未能发生转移的具体原因尚待进一步通过S1-PFGE结合Southern-blot和质粒序列分析证明。除ESBLs编码基因外,qnr和acc(6’)-Ib-cr也通常位于接合质粒上,并且可以通过接合作用进行转移和传播[46],本研究在IncN质粒阳性菌株中检出的qnrB和acc(6’)-Ib基因均可通过接合传给受体菌。
研究IncI1和IncN质粒在沙门氏菌中的流行状况及其在不同种属细菌间的传播其转移规律,将有助于对细菌耐药性传播提供实验依据,对食源性疾病爆发的原因进行溯源和风险评估,降低或防止食品安全事件的发生提供依据。
[1]XU Y M, TAO S, HINKLE N, et al.Salmonella, including antibioticresistant Salmonella, from flies captured from cattle farms in Georgia,USA[J].The Science of the Total Environment, 2018, 616/617: 90-96.DOI:10.1016/j.scitotenv.2017.10.324.
[2]KIRK M D, PIRES S M, BLACK R E, et al.World health organization estimates of the global and regional disease burden of 22 foodborne bacterial, protozoal, and viral diseases, 2010: a data synthesis[J].PLoS Medicine, 2015, 12(12): e1001921.DOI:10.1371/journal.pmed.1001921.
[3]PAN H, PAUDYAL N, LI X L, et al.Multiple food-animal-borne route in transmission of antibiotic-resistant Salmonella newport to humans[J].Frontiers in Microbiology, 2018, 9: 23.DOI:10.3389/fmicb.2018.00023.
[4]MARDER E P, CIESLAK P R, CRONQUIST A B, et al.Incidence and trends of infections with pathogens transmitted commonly through food and the effect of increasing use of culture-independent diagnostic tests on surveillance: foodborne diseases active surveillance network,10 US Sites, 2013-2016[J].Morbidity and Mortality Weekly Report,2017, 66(15): 397.DOI:10.15585/mmwr.mm6615a1.
[5]Centers for Disease Control and Prevention.Surveillance for foodborne disease outbreaks United States, 2015: annual report[R/OL].Atlanta: CDC, 2017.https://www.cdc.gov/foodsafety/pdfs/2015FoodBorneOutbreaks_508.pdf.
[6]World Health Organization.Overcoming antimicrobial resistance[M].Geneva: WHO, 2000.
[7]刘思洁, 赵薇, 孙景昱, 等.2014—2015年吉林省食源性致病菌监测结果分析[J].食品安全质量检测学报, 2018, 9(4): 947-950.
[8]ZHANG F Y, HUO S Y, LI Y R, et al.A survey of the frequency of aminoglycoside antibiotic-resistant genotypes and phenotypes in in broilers with septicaemia in Hebei, China[J].British Poultry Science,2014, 55(3): 305-310.DOI:10.1080/00071668.2014.891096.
[9]LOPATKIN A J, MEREDITH H R, SRIMANI J K, et al.Persistence and reversal of plasmid-mediated antibiotic resistance[J].Nature Communication, 2017, 8(1).DOI:10.1038/s41467-017-01532-1.
[10]HALL L V, DIERIKX C M, STUART J C, et al.Dutch patients, retail chicken meat and poultry share the same ESBL genes, plasmids and strains[J].Clinical Microbiology and Infection, 2011, 17(6): 873-880.DOI:10.1111/j.1469-0691.2011.03497.x.
[11]MOODLEY A, GUARDABASSI L.Transmission of IncN plasmids carrying blaCTX-M-1 between commensal Escherichia coli in pigs and farm workers[J].Antimicrobial Agents and Chemotherapy, 2009,53(4): 1709-1711.DOI:10.1128/AAC.01014-08.
[12]SPENGLER G, MOLNÁR A, SCHELZ Z, et al.The mechanism of plasmid curing in bacteria[J].Current Drug Targets, 2006, 7(7): 823-841.DOI:10.2174/138945006777709601.
[13]PEZZELLA C, RICCI A E, LUZZI I, et al.Tetracycline and streptomycin resistance genes, transposons, and plasmids in Salmonella enterica isolates from animals in Italy[J].Antimicrobial Agents and Chemotherapy, 2004, 48(3): 903-908.DOI:10.1128/AAC.48.3.903-908.2004.
[14]TOMOEDA M, INUZUKA M, KUBO N, et al.Effective elimination of drug resistance and sex factors in Escherichia coli by sodium dodecyl sulfate[J].Journal of Bacteriology, 1968, 95(3): 1078-1089.
[15]QIAO J, ZHANG Q, ALALI W Q, et al.Characterization of extendedspectrum beta-lactamases (ESBLs)-producing Salmonella in retail raw chicken carcasses[J].International Journal of Food Microbiology,2017, 248: 72-81.DOI:10.1016/j.ijfoodmicro.2017.02.016.
[16]YIN M Y, YANG B W, WU Y, et al.Prevalence and characterization of Salmonella enterica serovar in retail meats in market place in Uighur,Xinjiang, China[J].Food Control, 2016, 64: 165-172.DOI:10.1016/j.foodcont.2015.12.029.
[17]YANG B W, CUI Y, SHI C, et al.Counts, serotypes, and antimicrobial resistance of Salmonella isolates on retail raw poultry in the People’s Republic of China[J].Journal of Food Protection, 2014, 77(6): 894-902.DOI:10.4315/0362-028X.JFP-13-439.
[18]YANG B W, XI M L, WANG X, et al.Prevalence of Salmonella on raw poultry at retail markets in China[J].Journal of Food Protection,2011, 74(10): 1724-1728.DOI:10.4315/0362-028X.JFP-11-215.
[19]方婷子.沙门氏菌耐药质粒的筛查与特征分析[D].上海: 上海交通大学, 2015.
[20]黄培堂, 王恒墚, 周晓.分子克隆实验指南[M].3版.北京: 科学出版社, 2002: 36-42.
[21]Clinical and Laboratory Standards Institute.Methods for dilution antimicrobial susceptibility tests for bacteria that grow aerobically approved standard: CLSI M07-A10-2015[S].2nd ed.Wayne, PA,USA: Clinical and Laboratory Standards Institute, 2003.
[22]EDDRA A, FILALI F R, KARRAOUAN B, et al.Prevalence,molecular and antimicrobial resistance of Salmonella isolated from sausages in Meknes, Morocco[J].Microbial Pathogenesis, 2017, 105:340-345.DOI:10.1016/j.micpath.2017.02.042.
[23]MATHUR S, SINGH R.Antibiotic resistance in food lactic acid bacteria: a review[J].International Journal of Food Microbiology,2005, 105(3): 281-295.DOI:10.1016/j.ijfoodmicro.2005.03.008.
[24]马婧嘉.沙门氏菌耐药质粒及其耐药基因的筛查[D].上海: 上海交通大学, 2013.
[25]WAGNER S, GALLY D L, ARGYLE S A.Multidrug-resistant Escherichia coli from canine urinary tract infections tend to have commensal phylotypes, lower prevalence of virulence determinants and ampC-replicons[J].Veterinary Microbiology, 2014, 169(3/4): 171-178.DOI:10.1016/j.vetmic.2014.01.003.
[26]CARATTOLI A.Resistance plasmid families in Enterobacteriaceae[J].Antimicrobial Agents and Chemotherapy, 2009, 53(6): 2227-2238.DOI:10.1128/AAC.01707-08.
[27]WONG M H Y, KAN B, CHAN E W-C, et al.IncI1 plasmids carrying various blaCTX-M genes contribute to ceftriaxone resistance in Salmonella enterica serovar enteritidis in China[J].Antimicrobial Agents and Chemotherapy, 2016, 60(2): 982-989.DOI:10.1128/AAC.02746-15.
[28]SCHWEIZER C, BISCHOFF P, BENDER J K, et al.Plasmidmediated transmission of KPC-2 carbapenemase in Enterobacteriaceae in critically ill patients[J].Frontiers in Microbiology, 2019, 10: 276.DOI:10.3389/fmicb.2019.00276.
[29]CHMELNITSKY I, SHKLYAR M, LEAVITT A, et al.Mix and match of KPC-2 encoding plasmids in Enterobacteriaceae-comparative genomics[J].Diagnostic Microbiology & Infectious Disease, 2014,79(2): 255-260.DOI:10.1016/j.diagmicrobio.2014.03.008.
[30]PEREIRA P S, BORGHI M, ALBANO R M, et al.Coproduction of NDM-1 and KPC-2 in Enterobacter hormaechei from Brazil[J].Microbial Drug Resistance, 2015, 21(2): 234-236.DOI:10.1089/mdr.2014.0171.
[31]EILERTSON B, CHEN L, CHAVDA K D, et al.Genomic characterization of two KPC-producing Klebsiella isolates collected in 1997 in New York city[J].Antimicrobial Agents and Chemotherapy,2017, 61(4): e02458-16.DOI:10.1128/AAC.02458-16.
[32]WEINGARTEN R A, JOHNSON R C, CONLAN S, et al.Genomic analysis of hospital plumbing reveals diverse reservoir of bacterial plasmids conferring carbapenem resistance[J].MBio, 2018, 9(1):e02011-17.DOI:10.1128/mBio.02011-17.
[33]李静怡, 崔可琦, 冯赛祥, 等.不同源鼠伤寒沙门氏菌耐药性质粒不相容性分群的初步研究[J].中国畜牧兽医, 2014, 41(3): 54-58.
[34]冯建昆, 韩志华, 吴华, 等.携带四环素耐药基因的鸭源大肠杆菌质粒不相容群分析[J].江西农业学报, 2012, 24(1): 131-133.
[35]REDDY E A, SHAW A V, CRUMP J A.Community-acquired bloodstream infections in Africa: a systematic review and metaanalysis[J].The Lancet Infectious Diseases, 2010, 10(6): 417-432.DOI:10.1016/S1473-3099(10)70072-4.
[36]DJEGHOUT B, SAHA S, SAJIB M S I, et al.Ceftriaxone-resistant Salmonella typhi carries an IncI1-ST31 plasmid encoding CTXM-15[J].Journal of Medical Microbiology, 2018, 67(5): 620-627.DOI:10.1099/jmm.0.000727.
[37]杜雄伟, 李叶, 王晓辉.沙门氏菌耐药机制的研究进展[J].江苏农业科学, 2010(6): 487-490.
[38]BRUCE H, THOMSON N R, THOMAS C M, et al.Fitness of Escherichia coli strains carrying expressed and partially silent IncN and IncP1 plasmids[J].BMC Microbiology, 2012, 12(1): 1-9.DOI:10.1186/1471-2180-12-53.
[39]DOLEJSKA M, VILLA L, HASMAN H, et al.Characterization of IncN plasmids carrying blaCTX-M-1 and qnr genes in Escherichia coli and Salmonella from animals, the environment and humans[J].Journal of Antimicrobial Chemotherapy, 2013, 68(2): 333-339.DOI:10.1093/jac/dks387.
[40]蒋琰.肠杆菌科细菌质粒介导喹诺酮耐药机制研究[D].杭州: 浙江大学, 2009.
[41]CHEN S, ZHAO S, WHITE D G, et al.Characterization of multipleantimicrobial-resistant Salmonella Serovars isolated from retail meats[J].Applied & Environmental Microbiology, 2004, 70(1): 1-7.DOI:10.1128/AEM.70.1.1-7.2004.
[42]王晓泉, 焦新安, 刘晓文, 等.江苏部分地区食源性和人源沙门氏菌的多重耐药性研究[J].微生物学报, 2007, 47(2): 221-227.
[43]CLOECKAERT A, PRAUD K, DOUBLET B, et al.Dissemination of an extended-spectrum-beta-lactamase blaTEM-52 gene-carrying IncI1 plasmid in various Salmonella enterica serovars isolated from poultry and humans in Belgium and France between 2001 and 2005[J].Antimicrobial Agents and Chemotherapy, 2007, 51(5): 1872-1875.DOI:10.1128/AAC.01514-06.
[44]HOPKINS K L, LIEBANA E, VILLA L, et al.Replicon typing of plasmids carrying CTX-M or CMY β-lactamases circulating among Salmonella and Escherichia coli isolates[J].Antimicrobial Agents and Chemotherapy, 2006, 50(9): 3203-3206.DOI:10.1128/AAC.00149-06.
[45]RODRIGUES C, MACHADO E, PEIXE L, et al.IncI1/ST3 and IncN/ST1 plasmids drive the spread of blaTEM-52 and blaCTX-M-1/-32 in diverse Escherichia coli clones from different piggeries[J].Journal of Antimicrobial Chemotherapy, 2013, 68(10): 2245-2248.DOI:10.1093/jac/dkt187.
[46]GAY K, ROBICSEK A, STRAHILEVITZ J, et al.Plasmidmediated quinolone resistance in non-typhi serotypes of Salmonella enterica[J].Clinical Infectious Diseases, 2006, 43(3): 297-304.DOI:10.1086/505397.