乙醛酸是醛酸家族中分子质量最小的物质,是生物体三羧酸(tricarboxylic acid cycle,TCA)循环和乙醛酸循环中重要的有机酸,影响生物体的代谢平衡,其衍生品多达数十种,其中典型的有香兰素、乙基香兰素、尿囊素、对羟基苯丙氨酸、对羟基苯乙酰胺等[1]。这些衍生物被广泛应用于食品添加剂、糕点、糖果、烘烤食品、烟酒、香料、化妆品以及医药等行业[2-3]。随着乙醛酸需求量的增加,化学法生产乙醛酸带来的污染日益严重,因此,利用生物法合成乙醛酸成为重要途径。
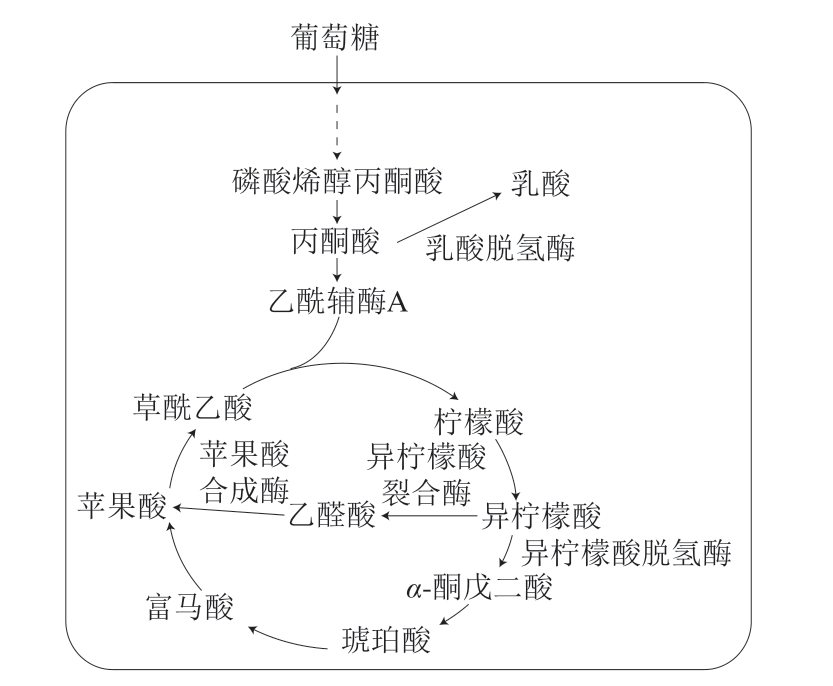
图1 C. glutamicum ATCC 13032代谢途径
Fig. 1 C. glutamicum ATCC 13032 metabolic pathways
谷氨酸棒状杆菌(Corynebacterium glutamicum)作为食品安全级的微生物,具有易培养、不产孢子、产率高、生物安全[4]等优点。随着C. glutamicum ATCC 13032全基因组测序的完成[5]和组学数据的积累[6],对其进行工程化改造得以实现[7-8]。在C. glutamicum糖酵解途径和TCA循环过程中,丙酮酸、乳酸、柠檬酸、异柠檬酸、乙醛酸和苹果酸之间存在复杂的网络系统,其代谢途径如图1所示,它们错综复杂并且相互关联[9],研究人员对其进行定向改造,使之具有更高的生产效率。常规的代谢工程技术一般是加强合成代谢途径,阻断竞争代谢途径及支流代谢途径,从而提高产品合成效率,但是敲除一些竞争途径的关键基因常常会影响细胞生长[10-11]。基于RNA引导的DNA核酸内切酶II型规律间隔成簇短回文重复序列(clustered regularly interspaced short palindromic repeats,CRISPR)/Cas9系统的CRISPRi技术,可以根据需要调节基因转录和蛋白表达水平,进而优化代谢网络,提高合成效率。Cleto等[12]应用CRISPRi技术,对C. glutamicum中pgi、pck、pyk三个基因表达强度进行调控,使pgi、pck的表达强度下调97%,pyk基因的表达强度下调98%,发酵后得到的代谢产物L-赖氨酸和L-谷氨酸的产量与基因敲除时的水平相当。刘旭峰等[13]应用CRISPRi技术对大肠杆菌(Escherichia coli)中3 个关键基因zwf、pfkA、gltA的表达强度分别进行下调,最终使苏氨酸产率均提升了约15%,而菌体的生长并没有受到显著影响。CRISPRi体系由失去核酸内切酶活性的dCas9蛋白和sgRNA两个组件构成[14],其中sgRNA由3 部分组成:20 nt目标基因特定核苷酸区域的识别序列、42 nt dCas9蛋白结合序列以及40 nt转录终止子序列[15-17]。CRISPRi工作机制为sgRNA引导dCas9蛋白结合在DNA的特定位置时,三者形成的复合物能够形成空间位阻,实现调节基因表达水平的目的。CRISPRi技术具有灵活高效的特点,被广泛应用于代谢工程和合成生物学等领域[18-24],现如今,CRISPRi已被证明能在多个物种间发挥抑制作用,例如大肠杆菌、分歧杆菌、酿酒酵母以及动植物细胞等[25-27]。
本研究以C. glutamicum ATCC 13032为出发菌株,敲除其支流代谢关键酶乳酸脱氢酶合成基因lldh,并建立CRISPRi调控体系,利用该体系下调C. glutamicum支流代谢中的关键酶异柠檬酸脱氢酶合成基因icd和苹果酸合成酶合成基因ms的表达强度,同时过表达异柠檬酸裂合酶合成基因icl,使乙醛酸得到积累,减少副产物的生成,以期为C. glutamicum工业生产乙醛酸研究提供一定参考依据。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株与质粒
表1 本实验所用的菌株、质粒
Table 1 Information about the strains and plasmids used in this study

菌株与质粒 说明 来源pK18mobsacB 含sacB蔗糖致死基因和卡那霉素抗性基因的整合载体 本实验室保藏dCas9-pK18mobsacB 整合了dCas9基因的pK18mobsacB载体 本实验构建icd-ms-pK18mobsacB 整合了icd和ms的sgRNA转录单元组的pK18mobsacB载体 本实验构建pXMJ19GZ 表达质粒 本实验室保藏icl-pXMJ19GZ 整合了icl的表达质粒 本实验构建E. coli Trans T1 大肠杆菌 北京全式金生物技术有限公司C. glutamicum ATCC13032 谷氨酸棒状杆菌野生型 本实验室保藏C. glutamicum/dCas9Δaldh 敲除aldh并整合dCas9的工程菌 本实验构建C. glutamicum/icd-msΔlldh 上一步工程菌基础上敲除lldh并整合icd和ms的sgRNA转录单元组 本实验构建C. glutamicum/icl 上一步工程菌的基础上整合icl表达质粒 本实验构建
1.1.2 试剂
High-Fidelity DNA聚合酶 北京全式金生物制品公司;T4 DNA连接酶、限制性内切酶 NEB(北京)有限公司;基因组提取试剂盒、质粒小提中量试剂盒、超薄DNA产物纯化试剂盒、琼脂糖凝胶DNA回收试剂盒 天根生化科技(北京)有限公司;聚合酶链式反应(polymerase chain reaction,PCR)引物由生工生物工程(上海)股份有限公司合成。
1.2 仪器与设备
T100 Thermal Cycler PCR扩增仪、Gel DOC XR全自动凝胶成像仪、Centrifuge 5418R高速冷冻离心机 美国伯乐公司;PowerWave XS2酶标仪 美国伯腾仪器有限公司;1200 Infinity高效液相色谱仪 美国安捷伦公司;MiniSpin台式高速离心机 德国艾本德公司。
1.3 方法
1.3.1 培养条件与培养基
E. coli培养条件:LB培养基,37 ℃培养,氨苄青霉素工作质量浓度为100 μg/L,卡那霉素工作质量浓度为50 μg/L;C. glutamicum ATCC 13032培养条件:37 ℃培养,卡那霉素工作质量浓度为30 μg/L。
LB培养基配方:10 g/L胰蛋白胨,10 g/L NaCl,5 g/L酵母提取物,调pH 7左右;固体培养基则添加琼脂粉20 g/L。孵育培养基配方:10 g/L胰蛋白胨,10 g/L NaCl,5 g/L酵母提取物,1 g/L乙酸钠,调pH 7左右。种子培养基配方:10 g/L玉米浆,25 g/L葡萄糖,20 g/L琼脂。发酵培养基配方:50 g/L葡萄糖,5 g/L乙酸钠,0.1 g/L NH4NO3,1.5 g/L KH2PO4,0.3 g/L CaCl2,0.25 g/L MgSO4,5 g/L (NH4)2SO4。
1.3.2 打靶质粒的构建
dCas9基因打靶质粒的构建:为将dCas9基因整合到基因组上,选择C. glutamicum支流代谢中乙醛脱氢酶合成基因aldh作为其替代基因,将dCas9替换到aldh基因的相应位置,以C. glutamicum基因组为模板,分别扩增出aldh基因上下同源臂片段、启动子sod基因片段,以实验室保存的dCas9-Peasy质粒为模板,扩增出dCas9基因片段,通过融合PCR技术将其搭接,获得的搭接片段与空质粒pK18mobsacB用NheI和XhoI进行双酶切,得到的酶切产物纯化,经T4连接酶于16 ℃过夜连接,转化至E. coli Trans T1感受态细胞中,获得打靶质粒dCas9-pK18mobsacB。
icd基因和ms基因的sgRNA转录单元打靶质粒的构建:为敲除C. glutamicum支流代谢关键酶乳酸脱氢酶合成基因lldh,同时也为将icd和ms基因的sgRNA转录单元整合到染色体上,因此选取lldh基因的相应位置作为目的位置,将sgRNA转录单元替换掉lldh基因。其中,icd基因的sgRNA转录单元组件包括启动子sod基因序列、20 bp的sgRNA基因序列、Cas9蛋白结合区域基因序列以及终止子rrnB基因序列;ms基因的sgRNA转录单元组件包括启动子tuf基因序列、20 bp的sgRNA基因序列、Cas9蛋白结合区域基因序列以及终止子rrnB基因序列。以C. glutamicum基因组为模板扩增出lldh基因的上、下同源臂片段,通过延长引物,利用2 次PCR扩增出icd基因和ms基因的sgRNA组件,利用Golden Gate方法构建出打靶质粒icd-ms-pK18mobsacB。
icl基因过表达质粒的构建:以C. glutamicum基因组为模板扩增出启动子sod基因片段、icl基因片段及终止子rrnB基因片段并搭接。将搭接片段与pXMJ19GZ质粒利用HindIII和BamHI进行双酶切,按照上述方法构建获得打靶质粒icl-pXMJ19GZ。
1.3.3 感受态细胞的制备及转化
C. glutamicum感受态细胞制备参考Jang等[28]方法,将过夜培养的菌株按照10%接种量接种于种子培养基中摇瓶振荡培养,使用PowerWave XS2酶标仪测定OD值,当OD600 nm达到0.5~0.7时,4 ℃、4 000 r/min离心收集菌体,弃掉上清液后加入10%甘油轻轻洗涤2 次。吸取10 μL打靶质粒和40 μL的C. glutamicum感受态细胞于1.5 mL离心管中轻弹混合,转入电转杯,静置5 min后开始电击,电击条件为电压2.5 kV/m、电阻250 Ω、电容25 μF。电击完成后立即向电击杯中加入1 000 μL预热的孵育培养基,重悬混合后转移至1.5 mL离心管中,在恒温加热器中于48 ℃加热6 min,之后30 ℃、180 r/min培养4 h。离心弃上清液后将菌液涂布在含有卡那霉素的LB固体培养基平板上,30 ℃恒温培养48~72 h。
1.3.4 工程菌的筛选及鉴定
C. glutamicum工程菌利用蔗糖致死基因sacB反向筛选技术,通过2 次同源重组方法进行基因敲除,其原理如图2所示。
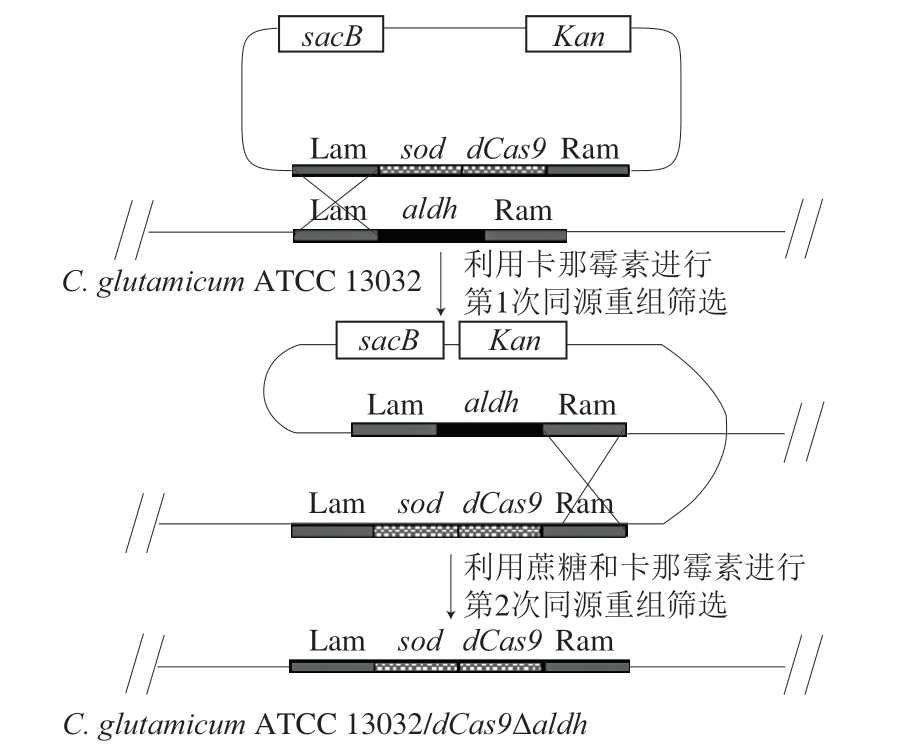
图2 同源重组原理示意图
Fig. 2 Schematic diagram of the homologous recombination principle
以突变菌株C. glutamicum/dCas9Δaldh为例,其构建及筛选过程如下:将打靶质粒dCas9-pK18mobsacB利用电转化转入到野生型C. glutamicum ATCC 13032菌株,48~72 h后,对在含卡那霉素的固体培养基上生长的单菌落进行分子验证,确定打靶质粒发生第1次同源重组,已整合到染色体上。选取验证正确的阳性单菌落转接到孵育培养基中,30 ℃、180 r/min培养12 h,让其在无抗生素条件下自由生长,使其发生第2次同源重组。将菌液稀释涂布在含10%蔗糖的LB固体培养基上,30 ℃过夜培养。将长出的转化子一一对应扩培到含10%蔗糖和卡那霉素的LB固体培养基上。在含蔗糖培养基上生长而在含卡那霉素的培养基上不生长的单菌落即为发生第2次同源重组的菌株,进一步通过菌液PCR验证获得阳性转化子。按照此方法,将质粒icd-ms-pK18mobsacB转入到C. glutamicum/dCas9Δaldh中,获得阳性工程菌C. glutamicum/icd-msΔlldh。将icl基因过表达质粒iclpXMJ19GZ转入到C. glutamicum/icd-msΔlldh中,获得阳性工程菌C. glutamicum/icl。
1.3.5 乙醛酸检测方法的建立
将获得的工程菌C. glutamicum/icl与野生菌分别接种到种子培养基中,30 ℃、180 r/min摇瓶振荡过夜培养,分别以等OD值接种到含5%葡萄糖的发酵培养基中,在相同的条件下摇瓶振荡培养48 h,定时取样,并测定菌体生长浓度,同时调节pH值,使之维持在7.0左右。将留存待测的样品经8 000 r/min离心10 min后,吸取上清液用0.22 μm醋酸纤维素滤膜过滤并稀释,利用高效液相色谱法检测发酵液中乙醛酸的含量。检测条件为:Agilent 1200高效液相色谱仪,紫外检测器,BDS HYPERSIL C18柱(4.6 mm×200 mm,5 μm),流动相为20 mmol/L KH2PO4(pH 2.8)和乙腈混合液,检测波长210 nm,柱温29 ℃,流速0.5 mL/min,进样量20 μL。
1.4 数据及图像处理
凝胶成像图利用Visio软件与PowerPoint处理并标注,生长曲线图利用Origin 8制作。
2 结果与分析
2.1 dCas9表达盒基因置换aldh基因工程菌的构建及验证
选取2 个转化子对获得的重组质粒dCas 9-pK18mobsacB进行PCR验证。如图3所示,dCas9表达盒基因与阳性对照条带大小一致,说明打靶质粒构建成功。
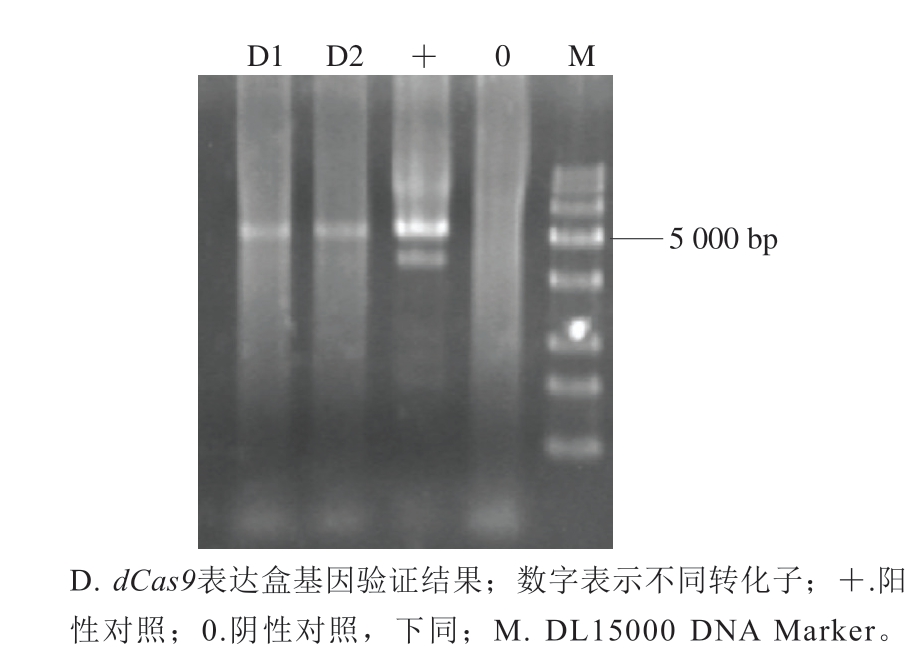
图3 质粒dCas9-pK18mobsacB PCR验证
Fig. 3 PCR verification of plasmid dCas9-pK18mobsacB
将打靶质粒dCas9-pK18mobsacB利用电转化转入到野生型C. glutamicum ATCC 13032菌株,选取4 个获得的转化子进行菌液PCR验证。如图4所示,aldh基因未能扩增出,而dCas9基因与阳性对照条带大小一致,从而说明dCas9表达盒基因置换了aldh基因的工程菌C. glutamicum/dCas9Δaldh构建成功。
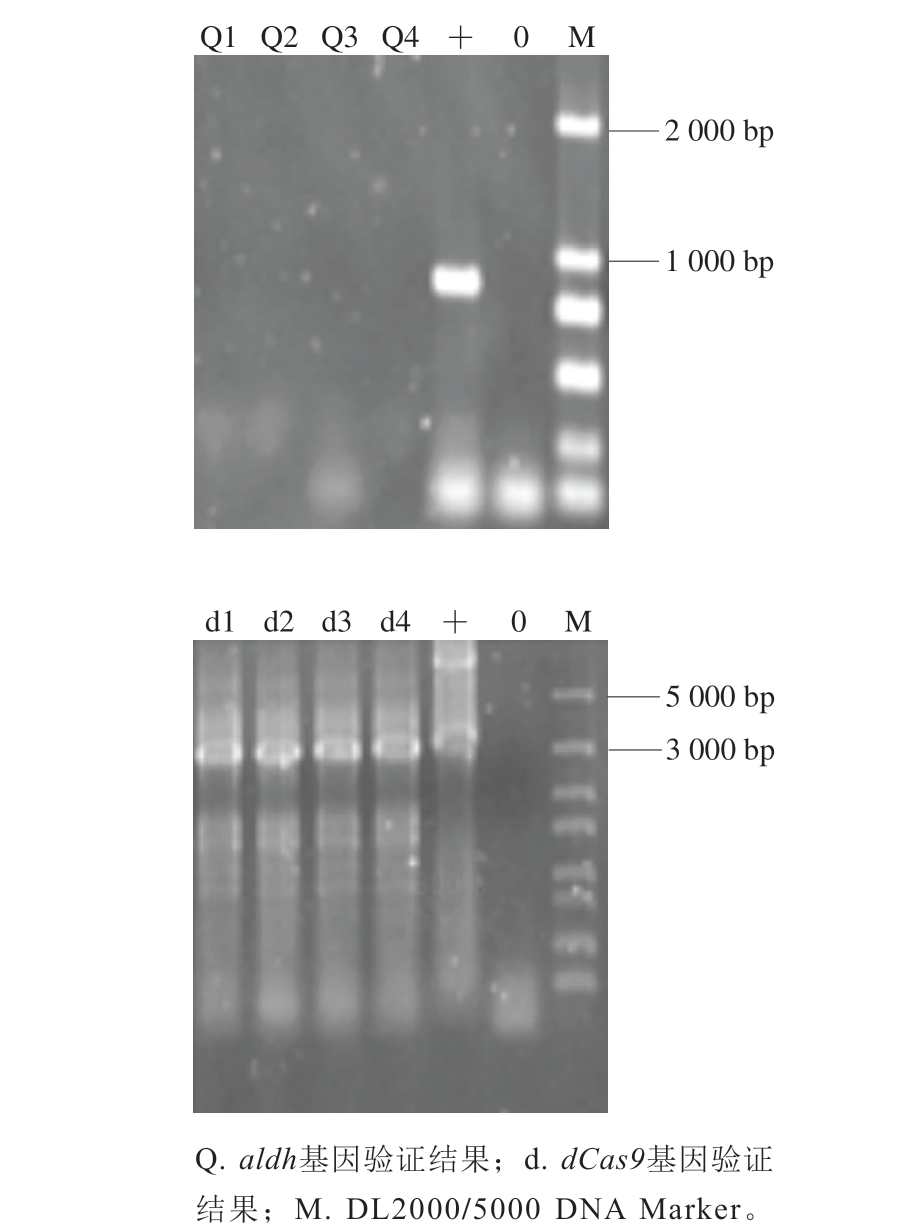
图4 工程菌C. glutamicum/dCas9Δaldh菌液PCR验证
Fig. 4 PCR verification of C. glutamicum/dCas9Δaldh
2.2 icd与ms基因sgRNA转录单元置换lldh基因工程菌构建及验证
对获得的打靶质粒icd-ms-pK18mobsacB进行酶切验证。如图5所示,经酶切后分别获得5 400 bp和3 000 bp两条片段,与pK18mobsacB质粒和icd与ms基因sgRNA转录单元片段大小一致,说明打靶质粒构建成功。
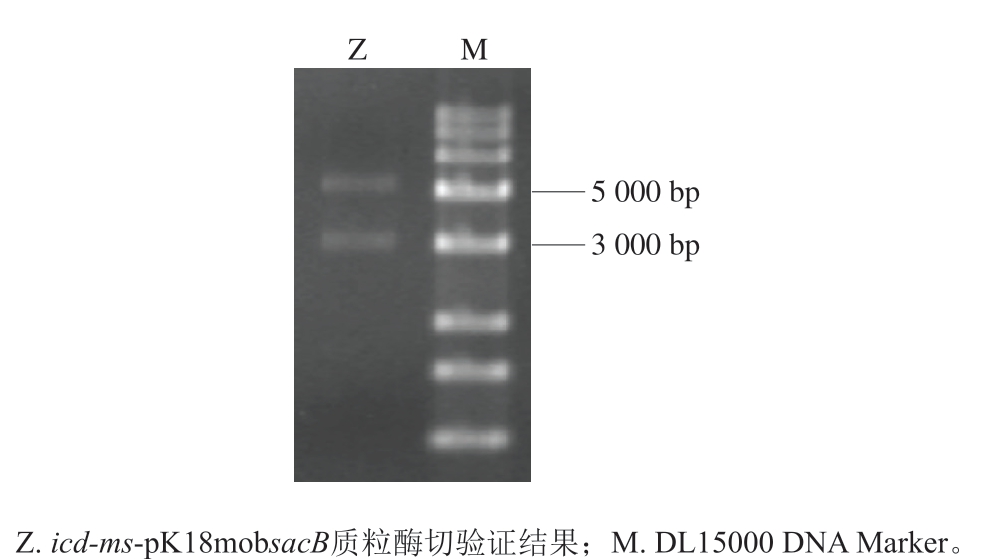
图5 质粒icd-ms-pK18mobsacB酶切验证
Fig. 5 Enzymatic digestion verification of icd-ms-pK18mobsacB
将打靶质粒icd-ms-pK18mobsacB利用电转化转入到已验证正确的工程菌C. glutamicum/dCas9Δaldh中,选取4 个获得的转化子进行菌液PCR验证。如图6所示,lldh基因未扩增出条带,而icd-sgRNA基因大小、ms-sgRNA基因大小均与阳性对照条带大小一致,从而说明icd与ms基因sgRNA转录单元基因置换lldh基因的工程菌C. glutamicum/icd-msΔlldh构建成功。
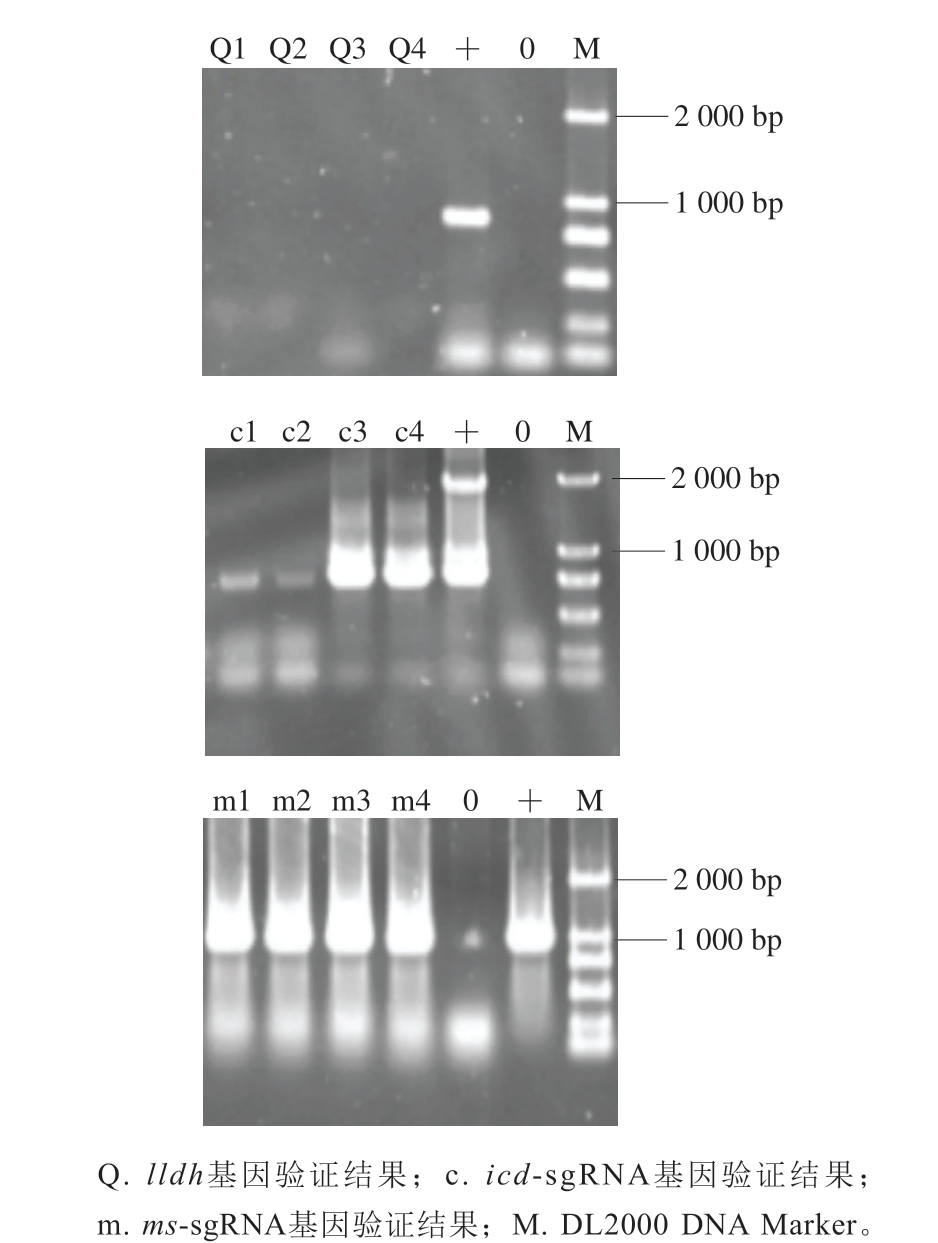
图6 工程菌C. glutamicum/icd-msΔlldh菌液PCR验证
Fig. 6 PCR verification of C. glutamicum/icd-msΔlldh
2.3 过表达icl基因的工程菌构建及验证
对获得的表达质粒icl-pXMJ19GZ进行酶切验证。如图7所示,经酶切后获得6 500 bp和1 600 bp两条片段,与pXMJ19GZ质粒和icl基因及启动子sod基因搭接片段大小一致,说明重组表达质粒构建成功。
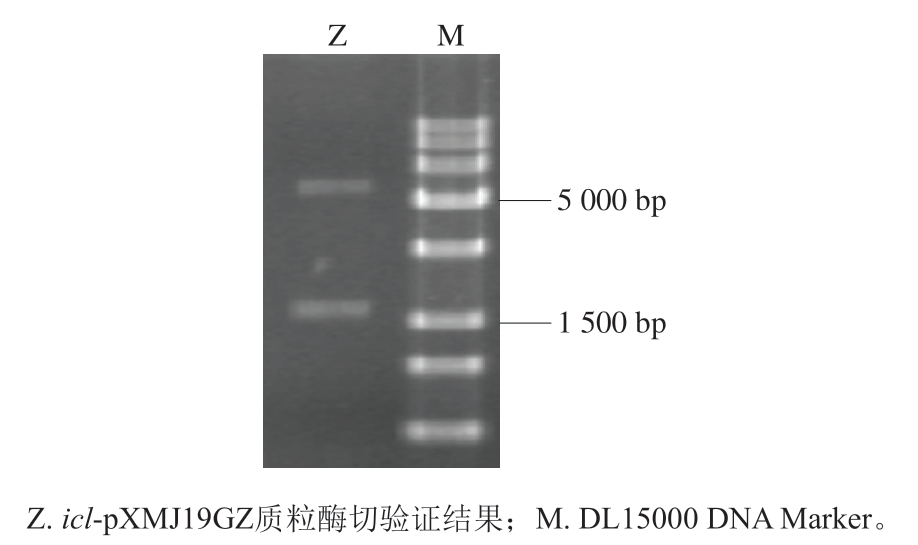
图7 质粒icl- pXMJ19GZ酶切验证
Fig. 7 Enzymatic digestion verification of icl-pXMJ19GZ
将表达质粒icl-pXMJ19GZ利用电转化转入到已验证正确的工程菌C. glutamicum/icd-msΔlldh,随机挑取4 个单菌落进行菌液PCR验证。如图8所示,扩增出的icl基因大小与阳性对照条带大小一致,说明工程菌C. glutamicum/icl构建成功。
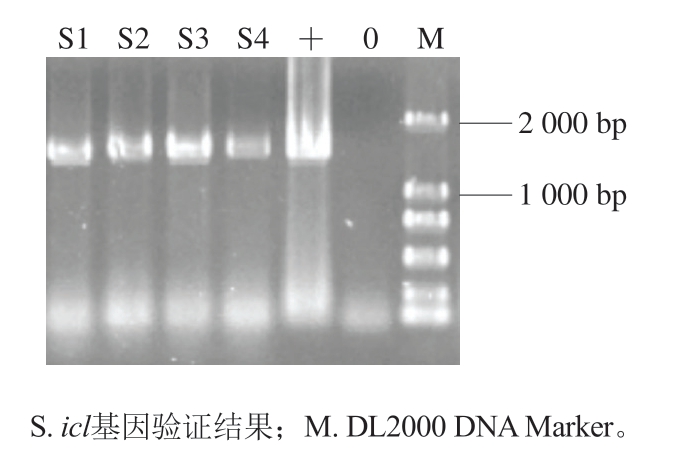
图8 工程菌C. glutamicum/icl菌液PCR验证
Fig. 8 PCR verification of C. glutamicum/icl
2.4 工程菌生长状况
工程菌与野生菌培养48 h,定时取样,并测定菌体生长浓度。如图9所示,野生菌和工程菌C. glutamicum/icl在0~24 h之间生长快速,24~48 h之间生长趋于平缓,二者生长趋势几近相同,说明敲除了aldh和lldh基因,同时沉默了icd和ms基因,并未影响菌株的糖酵解途径和TCA循环,菌株能正常生长。
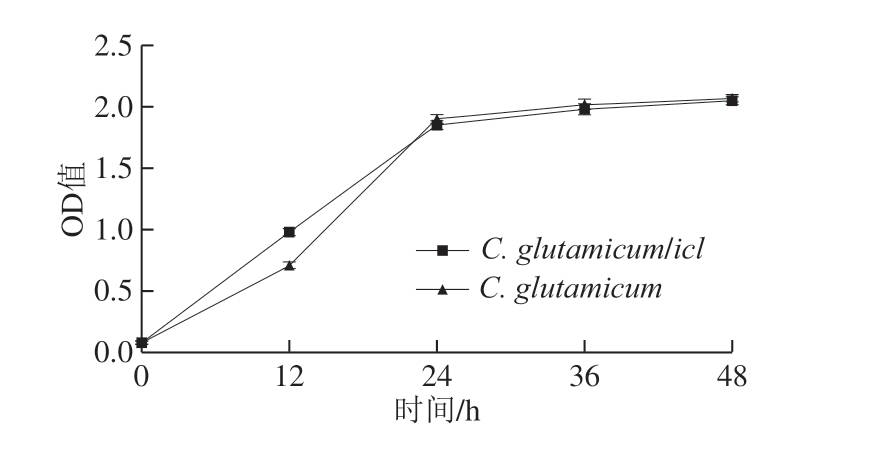
图9 野生菌与工程菌的生长情况
Fig. 9 Growth curves of wild-type and engineered bacteria
2.5 工程菌乙醛酸的发酵
为检测工程菌发酵液中乙醛酸产量,将工程菌C. glutamicum/icd-msΔlldh、C. glutamicum/icl与野生菌C. glutamicum ATCC 13032同时以5%葡萄糖作为碳源的发酵培养基进行发酵,连续培养48 h,期间每12 h取样待检测,利用高效液相色谱法检测发酵液中乙醛酸含量。经计算达到48 h发酵终点时,野生菌发酵液中乙醛酸几乎为0,工程菌C. glutamicum/icd-msΔlldh发酵液中乙醛酸质量浓度仅为1.05 mg/mL,而最终工程菌C. glutamicum/icl发酵液中乙醛酸质量浓度为5 mg/mL,实现了乙醛酸从无到有的过程。在最终工程菌C. glutamicum/icl的发酵过程中,0~36 h乙醛酸积累量很少,36 h后开始逐渐积累。野生菌C. glutamicum ATCC 13032和最终工程菌C. glutamicum/icl发酵终点时出峰结果如图10所示。
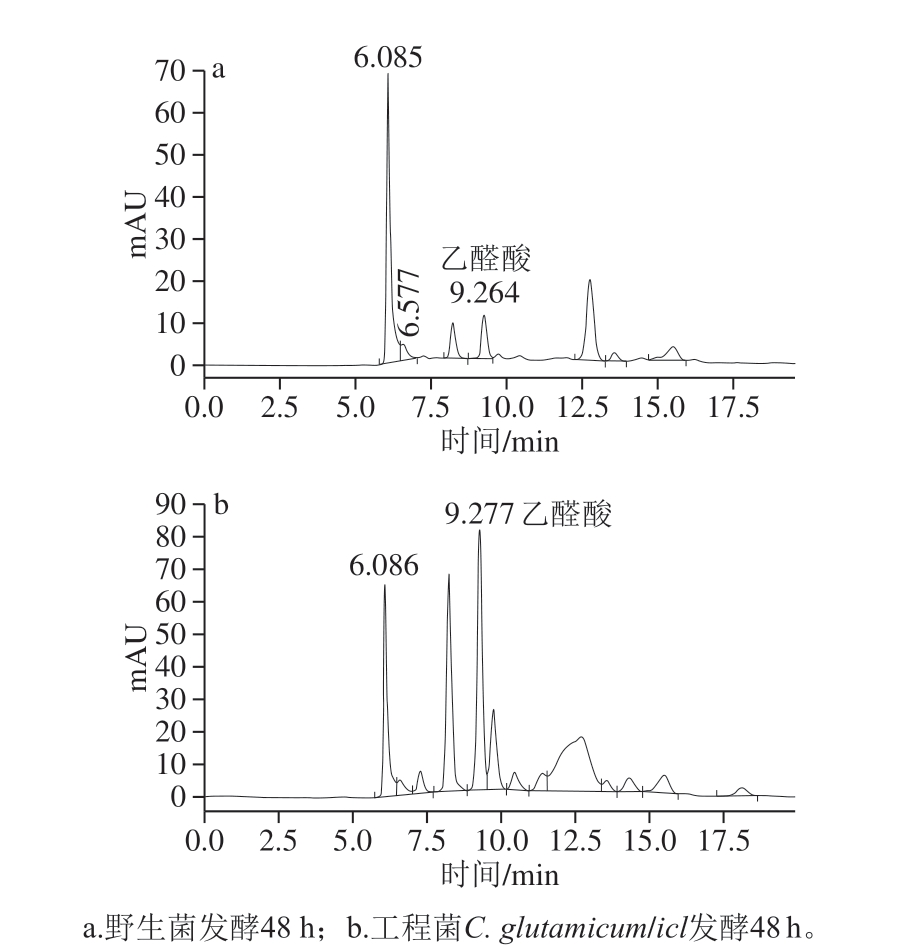
图10 野生菌与工程菌发酵液液相图谱
Fig. 10 Liquid chromatograms of fermentation broths of wild-type and engineered bacteria
3 讨 论
乙醛酸的生物合成法是近年来研究的热点。目前,生物法主要是通过酵母的静息细胞进行生物催化反应,利用乙醇酸氧化酶将乙醇酸氧化为乙醛酸[29],反应中需要以石油的衍生物乙醇酸作为底物,同时加入过氧化氢酶和黄素单核苷酸,此方法不仅操作复杂,而且生产成本高,严重限制了其产业化应用。与之相比基因工程菌发酵法更有利于可持续发展,但关于基因工程菌发酵法生产乙醛酸的报道却很少。Yang Chao等[30]发现通过提高异柠檬酸裂合酶基因icl的活性,可以增加流向乙醛酸旁路代谢通量;Toyoda等[31]通过敲除乳酸脱氢酶基因lldh,阻断了丙酮酸与乳酸的旁路代谢,使丙酮酸大量积累;Eikmanns等[32]发现,通过基因克隆敲除异柠檬酸脱氢酶基因icd后,α-酮戊二酸生成量大大减少,而乙醛酸含量增加,但是由于异柠檬酸脱氢酶是TCA循环的关键酶,阻断后造成菌体生长受到较大影响。
为解决敲除必需基因后影响菌体生长的问题,本研究借助CRISPRi技术,能够在不影响细胞生长的前提下,对目标产品的代谢途径进行优化和调整。在CRISPRi体系中,sgRNA能否高效准确表达,也关乎其能否正确引导Cas9蛋白对目的基因进行靶向编辑,因此sgRNA组件中启动子和终止子的选择也至关重要。Cleto等[12]选用诱导型启动子tac,构建了一系列sgRNA载体应用于C. glutamicum的CRISPRi体系,通过添加诱导剂IPTG诱导sgRNA表达;Liu Jiao等[33]选用C. glutamicum中组成型强启动子cspB的人工合成衍生体p11F作为其sgRNA组件的启动子,选用rrnB作为其sgRNA组件的终止子,并对其效率进行测试,发现脱靶率下降至(1.3±0.2)×10-2,并认为其更适合C. glutamicum的CRISPR/Cas9系统;Jiang Yu等[34]构建了C. glutamicum的CRISPR/Cpf1系统,选用人工合成的组成型启动子j23119作为sgRNA组件的启动子,选用rrnB作为其sgRNA组件的终止子,利用该系统对γ-谷氨酰激酶G149处进行密码子饱和突变,使其减轻对L-脯氨酸的抑制作用。本研究sgRNA组件中所采用的启动子sod、tuf和终止子rrnB是天然启动子及终止子,由于其转录范围较大,不能精确表达Cas9蛋白特定的识别序列,因而在CRISPR/Cas9系统中可能影响到Cas9蛋白的对目标位置的精准切割,影响到CRISPRi体系对目标基因的调控范围和强度。但在本研究中,通过检测工程菌发酵液,发现与野生菌相比乙醛酸得到了积累,由此可以推断本研究所选用的天然启动子及终止子能够应用于CRISPRi调控体系。为进一步提高代谢产物的产量,选择更为确切的人工合成启动子作为CRISPRi体系中sgRNA的启动子,是本实验室未来工作的重点。
本研究敲除C. glutamicum支流代谢关键酶乳酸脱氢酶合成基因lldh,并建立CRISPRi调控体系,利用该体系下调异柠檬酸脱氢酶和苹果酸合酶的表达,同时强化异柠檬酸裂合酶,使得工程菌与野生型相比较生长速度基本相同,在不影响生长的情况下,达到生物合成乙醛酸的目的,经检测工程菌培养48 h后的发酵液中乙醛酸质量浓度为5 mg/mL。由此获得的遗传资源将为乙醛酸生物合成生产奠定基础,此技术方法也为其他代谢工程产品研究提供借鉴。
[1] 周新军. 乙醛酸的制备研究进展[J]. 乙醛醋酸化工, 2017(2): 16-24.DOI:CNKI:SUN:JXHY.0.2017-02-005.
[2] 李光要, 程正载, 王涵鼎, 等. 乙醛酸的氧气-硝酸联合氧化制备及快速分析[J]. 河南科技大学学报(自然科学版), 2019, 40(2): 100-104.DOI:10.15926/j.cnki.issn1672-6871.2019.02.019.
[3] 肖铭. 乙醛酸合成技术的研究进展[J]. 精细与专用化学品, 2017,25(4): 47-49. DOI:10.19482/j.cn11-3237.2017.04.11.
[4] 张海灵, 李颜颜, 王小元. 代谢工程改造谷氨酸棒状杆菌合成及分泌途径生产-缬氨酸[J]. 生物工程学报, 2018, 34(10): 1606-1619.DOI:10.13345/j.cjb.180112.
[5] MENTZ A, NESHAT A, PFEIFER-SANCAR K, et al.Comprehensive discovery and characterization of small RNAs in Corynebacterium glutamicum ATCC 13032[J]. BMC Genomics, 2013, 14: 714-729.DOI:10.1186/1471-2164-14-714.
[6] WENDISCH V F, POLEN T. Transcriptome/proteome analysis of Corynebacterium glutamicum[J]. Corynebacterium glutamicum, 2013,23: 173-216. DOI:10.1007/978-3-642-29857-8_6.
[7] 靳海迎. 基于CRISPR-Cas9系统的谷氨酸棒状杆菌基因组编辑技术的研究[D]. 济南: 山东大学, 2017: 1-16 .
[8] 江南大学. 一种高产L-赖氨酸的重组谷氨酸棒状杆菌及其构建方法: CN201811465343.X[P]. 2019-02-22 .
[9] KIND S, BECKER J, WITTMANN C. Increased lysine production by flux coupling of the tricarboxylic acid cycle and the lysine biosynthetic pathway: metabolic engineering of the availability of succinyl-CoA in Corynebacterium glutamicum[J]. Metabolic Engineering, 2013, 15(1):184-195. DOI:10.1016/j.ymben.2012.07.005.
[10] NOH M, YOO S M, KIM W J, et al. Gene expression knockdown by modulating synthetic small RNA expression in Escherichia coli[J].Cell Systems, 2017, 5(4): 418-426. DOI:10.1016/j.cels.2017.08.016.
[11] YANG Y, LIN Y, LI L, et al. Regulating malonyl-CoA metabolism via synthetic antisense RNAs for enhanced bio-synthesis of natural products[J]. Metabolic Engineering, 2015, 29: 217-226. DOI:10.1016/j.ymben.2015.03.018.
[12] CLETO S, JENSEN J V, WENDISCH V, et al. Corynebacterium glutamicum metabolic engineering with CRISPR interference(CRISPRi)[J]. ACS Synthetic Biology, 2016, 5(5): 375-385.DOI:10.1021/acssynbio.5b00216.
[13] 刘旭峰, 王宁, 郝亚男, 等. CRISPRi干扰中心代谢基因转录对苏氨酸合成的影响[J]. 食品与发酵工业, 2019, 45(8): 1-7. DOI:10.13995/j.cnki.11-1802/ts.019489.
[14] BARRANGOU R, SIKSNYS V, HORVATH P, et al. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria[J]. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(39):15539-15540. DOI:10.1073/pnas.1208507109.
[15] SHEMIAKINA I I, ERMAKOVA G V, CRANFILL P J, et al.A monomeric red fluorescent protein with low cyto-toxicity[J]. Nature Communications, 2012, 3: 1204-1210. DOI:10.1038/ncomms2208.
[16] GARNEAU J E, DUPUIS M-È, VILLION M, et al The CRISPRCas bacterial immune system cleaves bacteriophage and plasmid DNA[J].Nature, 2010, 468: 67-71. DOI:10.1038/nature09523.
[17] MOJICA F J M, DÍEZ-VILLASEÑOR C, GARCÍA-MARTÍNEZ J,et al. Short motif sequences determine the targets of the prokaryotic CRISPR defence system[J]. Microbiology, 2009, 155(Pt 3): 733-740.DOI:10.1099/mic.0.023960-0.
[18] MARTIN J, KRZYSZTOF C, INES F, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J].Science, 2012, 337: 816-821. DOI:10.1126/science.1225829.
[19] JIANG W Y, BIKARD D, COX D, et al. RNA-guided editing of bacterial genomes using CRISPR-Cas systems[J]. Nature Biotechnology, 2013, 31(3): 233-239. DOI:10.1038/nbt.2508.
[20] QI L S, LARSON M H, GILBERT L A, et al. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression[J]. Cell, 2013, 152(5): 1173-1183. DOI:10.1016/j.cell.2013.02.022.
[21] DOMINGUEZ A A, LIM W A, QI L S. Beyond editing: repurposing CRISPR-Cas9 for precision genome regulation and interrogation[J].Nature Reviews Molecular Cell Biology, 2016, 17(1): 5-15.DOI:10.1038/nrm.2015.2.
[22] WU J J, DU G C, CHEN J, et al. Enhancing flavonoid production by systematically tuning the central metabolic pathways based on a CRISPR interference system in Escherichia coli[J]. Scientific Reports,2015, 5(1): 13477-13490. DOI:10.1038/srep13477.
[23] KIM S K, HAN G H, SEONG W, et al. CRISPR interferenceguided balancing of a biosynthetic mevalonate pathway increases terpenoid production[J]. Metabolic Engineering, 2016, 38: 228-240.DOI:10.1016/j.ymben.2016.08.006.
[24] YAO L, CENGIC I, ANFELT J, et al. Multiple gene repression in cyanobacteria using CRISPRi[J]. ACS Synthetic Biology, 2016, 5(3):207-212. DOI:10.1021/acssynbio.5b00264.
[25] AGNIESZKA P, ZAHIR A, HATOON B, et al. RNA-guided transcriptional regulation in planta via synthetic dCas9-based transcription factors[J]. Plant Biotechnology Journal, 2015, 13(4):578-589. DOI:10.1111/pbi.12284.
[26] CHOUDHARY E, THAKUR P, PAREEK M, et al. Gene silencing by CRISPR interference in mycobacteria[J]. Nature Communications,2015, 6(2): 6267-6277. DOI:10.1038/ncomms7267.
[27] SMITH J D, SURESH S, SCHLECHT U, et al. Quantitative CRISPR interference screens in yeast identify chemical-genetic interactions and new rules for guide RNA design[J]. Genome Biology, 2016, 17(1): 45-60. DOI:10.1186/s13059-016-0900-9.
[28] JANG K, BRITZ M. Improved electrotransformation frequencies of Corynebacterium glutamicum using cell-surface mutants[J].Biotechnology Letters, 2000, 22(7): 539-545. DOI:10.1023/A:1005629224109.
[29] 张建国, 杨洁. 生物法合成乙醛酸[J]. 微生物学杂志, 2003, 23(3):32-34. DOI:10.3969/j.issn.1005-7021.2003.03.011.
[30] YANG C, HAO N, YAN M, et al. Effect of overexpressing isocitrate lyase on succinate production in ldh-1 Corynebacterium glutamicum[J]. Chinese Journal of Biotechnology, 2013, 29(11): 1696-1700. DOI:10.13345/j.cjb.2013.11.001.
[31] TOYODA K, TERAMOTO H, INUI M, et al. The ldhA gene,encoding fermentative L-lactate dehydrogenase of Corynebacterium glutamicum, is under the control of positive feedback regulation mediated by LldR[J]. Journal of Bacteriology, 2009, 191(13): 4251-4258. DOI:10.1128/JB.00303-09.
[32] EIKMANNS B J, RITTMANN D, SAHM H. Cloning, sequence analysis, expression, and inactivation of the Corynebacterium glutamicum icd gene encoding isocitrate dehydrogenase and biochemical characterization of the enzyme[J]. Journal of Bacteriology,1995, 177(3): 774-782.
[33] LIU J, WANG Y, LU Y J, et al. Development of a CRISPRCas9 genome editing toolbox for Corynebacterium glutamicum[J]. Microbial Cell Factories, 2017, 16: 205-221. DOI:10.1186/s12934-017-0815-5.
[34] JIANG Y, QIAN F H, YANG J J, et al. CRISPR-Cpf1 assisted genome editing of Corynebacterium glutamicum[J]. Nature Communications,2017, 8: 15179-15189. DOI:10.1038/ncomms15179.