微生物发酵是重要的工业生产手段之一。相比于一般的化学合成和植物提取,微生物发酵条件温和,能利用易获取的廉价底物快速繁殖,具有产量高、易转化、周期短、易于提取纯化等优点。随着医药和食品领域发展,微生物发酵产品需求多元化,天然菌株中缺乏所需产物的代谢途径,或其代谢途径调控复杂,所需产物难以实现过量积累。拓宽微生物生产领域和提升微生物生产效率,是加快微生物发酵产业化的有效手段,已有大量研究人员致力于发酵优化和菌种改造。工业微生物发酵的人工代谢调控方法主要分两类:一类是基因修饰,如密码子优化、过量表达、竞争途径敲除等;另一类是发酵条件控制,如温度、pH值、供氧量、培养基碳氮比、前体物质添加等。这些技术手段相对成熟,主要作用位点针对性较强,但目前仍存在一定的局限性。传统改造多属于静态调控,相比于以动态调控为核心的合成生物学改造手段,传统改造方法不能在菌体所处环境条件发生变化时,动态地对代谢流进行调控,容易导致菌体自身代谢失衡,氧化还原平衡被破坏,进一步可能引起菌体的裂解死亡,因此亟需一种基于不同需要进行构建工程菌的思路。
合成生物学作为生物学和工程学的一个跨学科分支,对生物过程进行工程化。构建非天然存在的人工生物系统,同时重新整合天然存在的生物系统是合成生物学的出发点和归宿。合成生物学使用的最基础工具为标准化生物元件,生物元件通过逐级组装形成生物线路、生物网络和生物系统,通过对各元件的设计和组合,构建出具有特殊功能的人工系统。2000年,第一个利用遗传拨动开关构建的合成双稳态基因调节网络被建立[1],它由任意两个相互抑制的启动子组成,利用瞬态化学或热感应在稳态之间翻转,显示出接近理想的开关阈值,对生物技术和生物计算具有重要的意义。基于此设计出的人工基因调控模块构成菌体代谢活动的系统,能够更合理地设计微生物体内代谢网络,有效解决菌体代谢失衡问题。微生物作为合成生物学研究的重要载体,合成生物学技术的出现使工程菌种研究获得了全新的机遇,合成生物技术的发展更是极大地提升了被构建工程菌的生产能力。在合成生物学研究中,微生物通常被设计并改造成细胞工厂,应用于不同产品的生产。本文概述合成生物学技术,综述借助编辑工具和生物元件进行代谢通路的移植或动态调控构建工程菌,并介绍利用所构建工程菌在生产氨基酸、有机酸、芳香族化合物、糖类中的应用。
1 合成生物学技术方法
1.1 基因编辑工具
规律间隔成簇短回文重复序列(clustered regularly interspaced short palindromic repeats,CRISPR)/CRISPR关联蛋白(CRISPR-associated proteins,Cas)系统是新涌现的基因编辑工具,能够完成RNA导向的DNA识别及编辑。该工具的开发为构建更高效的基因定点修饰技术提供了全新的平台。2013年,Le Cong等[2]报道了CRISPR/Cas系统能同时编辑哺乳动物基因组中的多个位点,作为一种RNA引导的内切核酸酶系统,它可以直接通过核苷酸碱基配对靶向DNA位点。与传统基因编辑工具相比较,CRISPR系统作为一种新型编辑工具,具有省时、易构建、精度高等特点,以CRISPR系统为代表的新型基因编辑技术飞速发展,在诸多生物学领域中得到广泛应用,成为近年基因组编辑的热门工具,当前已被广泛应用于基因敲除、基因沉默和基因激活等方面,极大扩展了基因编辑技术的应用范围,如图1所示。
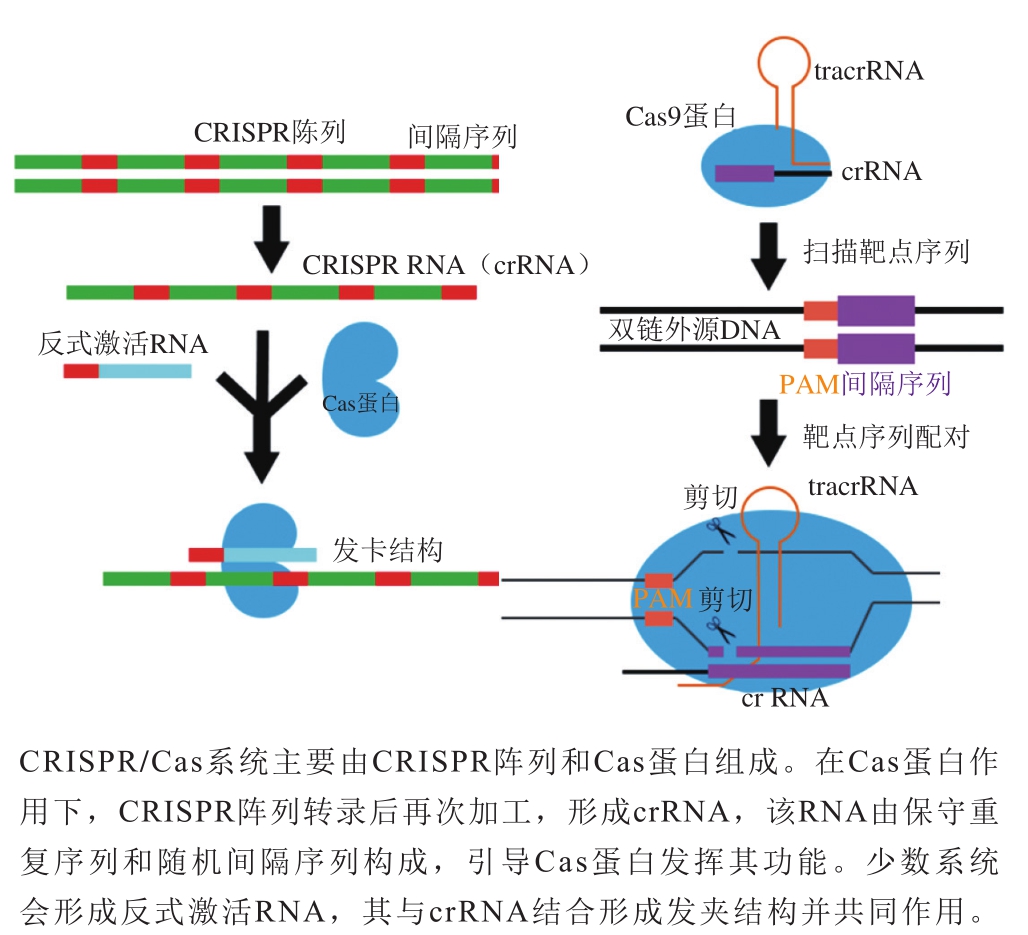
图1 CRISPR/Cas系统作用机制[3]
Fig. 1 Mechanism of action of CRISPR/Cas system[3]
CRISPR/Cas系统被广泛应用于多物种基因编辑的同时,Cas9蛋白对常用模式菌——谷氨酸棒杆菌的毒性问题也随之被发现,这一困扰阻碍了微生物改造研究进程。2015年,Bernd等[4]报道了一种具有不同于Cas9特征的更简单的Cpf1蛋白。Cpf1是一种缺乏反式激活CRISPR RNA(trans-activating CRISPR RNA,tracrRNA)的单一RNA引导的内切酶,通过DNA双链交错断裂来裂解DNA,抗干扰能力增强。与Cas9蛋白相比,Cpf1蛋白对基因组编辑活性较高,脱靶效应更低,应用范围更广,并解决了部分菌种中Cas9不适用问题。自被发现以来,CRISPR-Cpf1已被广泛应用于微生物研究。Krumbach等[5]用CRISPR/Cpf1系统在机械敏感通道MscCG的关键位置进行密码子饱和诱变,筛选到可外排L-谷氨酸的谷氨酸棒杆菌突变体。Ding Dan等[6]同时表达来自Cas9和Cpf1内含子的多个gRNA和crRNA,与常规Cpf1载体相比,这种杂合基因在同时靶向多个位点时具有更高的效率和稳定性。Ferenczi等[7]将CRISPR/Cpf1核糖核蛋白与单链DNA修复模板一步共递送,在靶向核基因编辑效率低下的莱茵衣藻中实现高效、精确的靶向DNA替换。上述研究证明了CRISPR/Cpf1系统在内源基因座的序列特异性突变和表位标记的无转基因和无选择标记中的用途。
1.2 基因组合成方法
随着近10 年合成生物学飞速发展,越来越多的科研团队都致力于用合成DNA来构建复杂的基因系统。细胞自身基因组的人工改造自然而然地进入了人们视野。2008年,Venter研究所用化学合成的DNA构建出580 000 bp支原体基因组的完整副本[8];2016年,该研究所重新设计了之前合成的支原体基因组,删除部分被认定为生长非必需的基因和DNA,被删除部分约占全基因组的一半[9]。这项工作未对基因进行任何编码,保留的基因组部分与自然序列相同,但它是目前人为改造最多的基因组,它的基因内容和布局与天然基因组相比有很大差异。
作为基因组合成领域的里程碑,人工合成酵母基因组计划旨在保证人工合成的酵母具有与野生型酵母相似活性的基础上,实现人工合成酿酒酵母的全基因组构建,并且达到酵母基因组的完全人工编写,构建全设计型、全合成型的酿酒酵母基因组。人工合成酵母基因组计划已有大量研究者参与探索,Shen Yue等[10]通过loxP介导进行合成染色体的重排和修饰,将基因组半合成酵母中与生长缺陷有关基因序列用野生型酵母序列替代,获得与天然菌株生物过程高度一致的新型酵母菌株。同时Shao Yangyang等[11]在酿酒酵母基因组结构的融合重建上取得突破,通过连续的端对端染色体融合和着丝粒缺失,从含有16 条线性染色体的酿酒酵母单倍体细胞中获得了一种功能性单染色体酵母,这项研究证实了从染色体结构和功能方面探索真核生物进化的可能。贾斌[12]设计的“与门”开关实现了合成型酿酒酵母基因组重排的精确调控。通过合成型基因组重排获得多种表型,提高了酵母中类胡萝卜素产量,成功发掘验证了相关的功能基因。
2 合成生物学设计理论
2.1 生物元件设计与动态调控
2.1.1 基础元件设计
生物元件是生命体内具有最基础生物功能的基本结构单元,例如调控基因表达的调控元件,包括启动子、终止子、核糖体结合位点(ribosomebinding site,RBS)以及特定功能的结构元件(如天然产物合成途径中酶基因)。近年来随着对启动子序列核心元件和上游激活序列认识的不断提高,人工杂合启动子的开发和应用正在迅速发展。将来有望获得序列更短、性能更强的启动子,从而有利于实现对模式菌中所引入的复杂通路进行更有效的调控。通过构建文库的方法来设计启动子或RBS等调控元件非常有效,但文库构建与元件筛选效率较低,不适用于需要大量调控元件的人工生物系统构建,此外许多元件无法快速进行文库筛选。因此建立一套完整的启动子的强度预测模型至关重要。Salis等[13]通过将描述翻译起始的生物物理模型与最优化算法相结合,提出了一套热力学预测模型用于RBS设计。该模型通过预测RBS与核糖体结合的强度,可对蛋白翻译的起始速率及蛋白表达水平进行定量调控。Shi Feng等[14]通过优化RBS序列与启动子间隔,提高苏云金芽孢杆菌来源的异亮氨酸双加氧酶表达。由于已有模型预测准确度偏低,现有的在启动子或RBS强度预测水平不足以支持基于强度预测的理性设计,难以从真正意义上实现启动子或RBS序列设计,而且缺乏通用性。借助人工神经网络建立效果和普适性更好的预测模型和方法是可进一步挖掘的方向[15],所建立的模型和方法还可用于指导合成生物学精细调控元件的理性设计[16]。不同来源不同功能的生物元件,可以通过复杂设计与其他元件或模块进行组装[17],形成更大规模的具有特定生物学功能的生物回路、装置和系统。因此,生物元件的搜寻与标准化是设计与组装更复杂的功能模块和生命系统的基础。
2.1.2 传感器与开关设计
借助生物传感器进行动态调控,可以取代常规基因敲除,从而解决因基因敲除导致的菌体生长不良影响产量这一问题。目前生物传感器主要包括荧光共振能量转移传感器、核糖开关传感器和转录因子传感器。
荧光共振能量转移传感器由于操作简单、敏感性高、反应速度快的特点常用于食品安全检测领域,例如对赭曲霉毒素A的检测[18]。核糖开关对配体的响应更为快速,结构简单易于改造,更适用于基因表达的动态调控。王兴[19]设计了对异丙基硫代半乳糖苷浓度具有高灵敏度的传感体系,包括产生信号分子LuxI蛋白表达元件和能接受信号分子的刺激产生绿色荧光蛋白的感应元件。转录因子传感器借助转录因子识别和结合基因启动子区的元件来控制下游基因表达,其调控基因表达的特性适用于建立细胞工厂,例如筛选高活性酶及高产菌、动态调控蛋白表达。如Uchiyama等[20]开发了一种基于报告基因分析的酶筛选方法,在大肠杆菌中利用转录因子Ben R控制绿色荧光蛋白基因表达,对酰胺酶活力进行筛选。Rogers等[21]设计出一种小分子响应性转录因子Acu R的遗传编码生物传感器,根据荧光强度与靶代谢产物的细胞内浓度成比的原理,能实时监测菌体内3-羟基丙酸浓度的改变。Kortmann等[22]利用赖氨酸生物传感器对荧光激活细胞进行分选,获得了有利于L-赖氨酸产量提升的丙酮酸羧化酶突变体。
生物开关作为基因表达动态调控的重要组成部分,通过感应某一条件的改变,控制基因表达“开启”或“关闭”,在代谢工程中常与传感器联动工作来完成调控。Binder等[23]使用基于tac表达模块的光响应控制元件,使用光控开关来改善谷氨酸棒状杆菌中生长抑制型倍半萜生产。Cheng Lin等[24]将温控开关用于α-酮丁酸生产,通过温度升高诱导苏氨酸脱氨酶过量表达,获得α-酮丁酸高产菌株。Zhou Libang等[25]在产赖氨酸的谷氨酸棒杆菌LP917菌株中,使用大肠杆菌和枯草芽孢杆菌的核糖开关来控制gltA基因和三羧酸循环活性,提高了进入赖氨酸合成途径的代谢通量。修宇[26]以重要中间产物柚皮素为核糖开关的小分子配体,利用能与柚皮素特异识别的适体库,以tetA双选择标记基因作为报告基因,在生物体内通过两条不同柚皮素浓度的筛选路径,富集获得具有不同最适检测适应范围的核糖开关库。由于核酸适配体对目标产物缺乏响应,基于核糖开关的代谢物传感器在代谢工程中的应用仍然有限。近年来出现了一些有助于设计核糖开关的工具,或许可以促进核糖开关在微生物工业生产上的应用。
2.2 蛋白质设计
根据目前的认知水平,功能生物元件的完全从头设计有是一项艰难的挑战,特别是在氨基酸序列与高级结构确定方面。许多蛋白质元件缺少高通量模型,无法快速进行文库筛选获得具有预期功能的突变体。因此根据掌握的元件序列与功能之间存在的特殊关系建立计算机模型,对元件的关键位点进行改造,理性设计具有预期功能和控制特性的元件是元件设计的重要方向。在蛋白质元件设计上,可以基于已知元件结构和功能间的关系,替换编码生物元件的DNA序列,或引入编码非天然氨基酸序列,获得转化的蛋白质元件;其次可以根据大规模定向进化筛选,将随机变异导入编码生物元件DNA中,用合适的筛选方法进行大规模筛选,得到期望的蛋白质生物元件;还可以利用计算模拟,定向设计与合成蛋白质中拓扑结构,利用从头设计合成出具有特殊结构的蛋白质。
在蛋白质从头设计上,蛋白质的整体结构依赖设计,但合成的蛋白质结构特征常常与预测结构不尽一致,因此迫切需要有效预测蛋白质结构折叠模式的方法。给定可以准确计算某种蛋白质构象能量的生物物理方法,同时基于快速抽样搜索的算法,就可以快速寻找新型蛋白。Kevin等[27]基于X射线衍射,设计了两个蛋白质结构的蛋白质低聚物,形成的双组分笼具有四面体对称性。两种蛋白均具有较高的可溶性。此外还使用了一个有利于氢键网络的形成计算协议,而不是完全的疏水相互作用,以稳定设计的蛋白质-蛋白质界面。Huang等[28]从头设计了四重对称、耐热的丙糖磷酸异构酶(triosephosphate isomerase,TIM)桶状蛋白质,蛋白质的X射线晶体结构与设计的TIM木桶模型几乎相同。设计序列与其他天然存在的TIM桶状蛋白超家族相距甚远,这为定制酶提供了可能。Doyle等[29]报道了用于从头设计串联重复蛋白结构的计算方法的开发和验证,应用这些方法设计了一系列闭合的类螺旋体重复结构,并验证了在蛋白质结构数据库中尚未出现的左手螺旋结构的α-类蛋白重复序列。Justin等[30]借助人工设计的能将二氧化碳转化为燃料和化学物质的酶Formolase,在细菌体内构建一种全新的代谢通路,说明现代蛋白质工程和设计工具能够促进构建全新的生物合成途径。这些人造蛋白结构可能会在生物纳米技术中得到应用,并且为蛋白质制剂、新型功能酶的合成等方面的研究作出了贡献,具有很大的应用潜力。
2.3 合成线路与合成网络设计
基于合成生物元件,对多个元件进行串联或并联,组成了具有处理信息能力和干预生命体功能的合成基因线路。合成基因线路设计首先需要构建基础功能元件,如拨动开关、感应器等,然后进行元件组装,构建复杂的功能线路。基因线路的人工合成有助于对菌体自身基因组的删减,基因组简化能提升人们对基因组功能的认知,创造精简高效的工程菌,如删除菌体代谢调节网络的重复部分、稳定插入的外源基因、优化菌体代谢途径以及加强菌体对物质和能量的利用。近年来,合成基因线路的设计取得一些进展,科学家们构建出了基因传感器和具有所需强度的调控元件、基因表达动态控制器和生物触发器等一批功能组件。基于当前的研究,在单个细胞内构建的合成基因线路,难以携带大量的人为附加元件。基因表达的竞争和噪声限制了基因线路规模化,而降低噪声通常需要增加基因表达强度,使得细胞内有限的资源和能量分配紧张。如果减少元件数和表达量来降低其资源占用量,会增加基因线路表达的不确定性。使用动态信号可以在一定程度上抵抗外源噪声带来的不确定性,通过监测单个细胞,发现细胞内基因表达差异小于细胞间差异。徐显皓[31]的研究结果表明,增加监测时间点可以提高对外源噪声的抗干扰能力。此外,针对动态信号的特点有针对性地设计基因线路也能降低基因表达噪声。
随着合成生物学领域的发展,合成基因线路的功能越来越多。但随着基因线路规模的扩大,简单的构建方法已无法满足日益增长的大规模合成基因线路的设计需要。利用合成生物学构建合成基因线路,在特定的条件下关闭或打开基因的表达,实现细胞生长的内源代谢途径和目的产物生产的异源途径之间的转换。通过建立动力学模型和代谢模型,检验合成生物网络是否具有期望的调控特性,在检测的基础上设计能处理特定信息的合成生物网络,并分析实际代谢网络中的代谢通量,为人工合成代谢网络的构建提供了指导。大量研究者将线性优化引入代谢网络分析,基于约束的建模理念和通量平衡分析,结合自动化和手工修饰,在基因组规模细胞代谢网络设计上构建了一系列模式生物的代谢网络。已有的典型模式生物的基因组规模代谢网络包括Escherichia coli、Chlamydomonas、Methanosarcina barkeri、Klebsiella pneumoniae、Geobacter metallireducens、Bacillus subtilis等物种的代谢网络模型,E. coli、Saccharomyces cerevisiae的基因调控网络模型是主要代表。由合成线路构成的、具有特殊拓扑结构的基因网络常作为研究模板,被称为基因网络模体,部分基因网络模体具备特殊的功能,如瞬态响应和倍数感知等[32]。Planes等[33]对酵母菌基因网络进行编辑,生产出青蒿素前体,在植物源药物的异源生产上获得重大突破。
3 合成生物学构建微生物工程菌的应用
随着对代谢工程和合成生物学领域探索的不断深入,人工构建微生物工程菌在合成各类产物方面具有独特的优势,成为了国内外工业生物技术研究的热点。首先通过优化合成途能够获得工程菌框架,然后通过提高其性能和稳定性,进化代谢和全局调控提高其生产能力,并进行多重比较,选择合适的宿主菌,将其转化成生产可用的工程菌。一些模型微生物(大肠杆菌、毕赤酵母、谷氨酸棒杆菌、枯草芽孢杆菌、链霉菌和解脂耶氏酵母[34])已被鉴定为异源表达和大规模生产高价值天然产物的理想基础。这些模型微生物具有作为底盘细胞的优势,因为有各种有效的合成生物学工具来设计它们,并且它们具有较高的生长率和经过充分研究的基因组和代谢网络。在底盘细胞中对特定代谢产物生物合成相关基因进行重新设计、组装和构建,从而利用底盘细胞已有的代谢网络合成目标产物,实现目标产物的异源高效合成。
3.1 氨基酸
合成生物学技术的发展对氨基酸发酵生产行业具有较大的推动作用,不仅能提升传统氨基酸产品的产量,还可以获得产特殊氨基酸产品的工程菌。Zhou Libang等[25]使用赖氨酸核糖开关和细胞内L-赖氨酸作为信号来控制与赖氨酸生物合成相竞争的必需代谢旁路,较一刀切的传统改造手段,降低了对菌体生长和代谢影响。龙梦飞[35]设计出能动态调控α-酮戊二酸脱氢酶活性的基因调控线路,并利用该调控系统下调支路关键酶活性,较传统敲除支路手段,其小幅度变动宿主原基因组,提高了反式-4-羟基-L-脯氨酸的产量。Zhang Bin等[36]在arg CJBD和arg F引入RBS置换和起始密码子置换,并在序列中插入经筛选的终止子,显著提升L-鸟氨酸的积累,缓解了直接敲除导致的谷氨酸棒杆菌菌体生长障碍。合成生物学相关技术的大力发展将为继续突破模式工程菌生产各类氨基酸提供强有力的保证,相比于化工生产用的酶法、抽提法、合成法,发酵法生产氨基酸具有条件温和、产物纯度高的优点,而进一步借助合成生物学技术,在对生产用菌全局代谢途径清晰的前提下,能对菌体使用动态的、更适当的、更精准的、影响更小的改造方式来高产目的氨基酸。
3.2 芳香族化合物
含有双键和单键交替的不饱和环的芳香族化合物在工业上应用广泛,例如作为添加剂被使用在药物、农用化学品、食品、饲料和化妆品中。芳香族化合物主要通过化学合成,也可以通过提取从植物中获得。由于一系列资源及环境问题,利用可再生生物质生产芳烃的生物技术越来越受到关注。许多微生物能够分解代谢过多的芳香族化合物,拦截这些代谢途径可能导致生物技术生产增值芳香族化合物,研究者重点关注莽草酸途径这一生成芳香族氨基酸和其他多种芳香族化合物的重要途径。莽草酸途径中,最终产物4-羟基苯甲酸(4-hydroxybenzoic acid,4HBA)是一种在防腐杀菌方面有利用价值的原料。Barker等[37]构建了一系列重组大肠杆菌菌株,获得了1 株4HBA反馈抑制得到缓解的、对菌体毒性作用下降的重组菌菌株,4HBA实验室规模产量达到12.0 g/L。Verhoef等[38]从恶臭假单胞菌P. putida S12中敲除4HBA羟化酶的基因pob A,转入载有苯丙氨酸解氨酶基因的质粒,构建了失去降解4HBA能力的S12pal B1菌株。由于恶臭假单胞菌S12缺乏利用木糖的天然能力,Meijnen等[39]将来自大肠杆菌的木糖异构酶途径引入到产对羟基苯甲酸酯的恶臭假单胞菌中,获得能利用木糖的菌株。以往4HBA从石油成分中合成,对资源和环境的损耗极大。基于合成生物学的工程菌设计思路,借助目标产物代谢途径的小规模删减和异源合成途径的移植,使本不能留存4HBA的大肠杆菌在改造后有望实现4HBA及其他莽草酸代谢途径产物的工业化生产。
3.3 糖类
氨基葡萄糖(2-amino-2-deoxy-D-glucose,GlcN)常用作膳食补充剂,GlcN及其衍生物N-乙酰氨基葡糖胺(N-acetyl-D-glucosamine,GlcNAc)具有控制疼痛、改善功能和延缓关节结构改变功效,在医药领域广泛应用。刘延峰[40]在枯草芽孢杆菌中共表达D-葡糖胺-6-磷酸合成酶和D-葡糖胺-6-磷酸乙酰化酶,成功构建GlcNAc合成途径,实现GlcNAc在枯草芽孢杆菌中积累。在该重组枯草芽孢杆菌基础上,对GlcNAc合成途径关键酶进行调控,采用双启动子系统优化GlcNAc合成途径中关键酶表达,严谨型启动子PxylA调控D-葡糖胺-6-磷酸合成酶表达,并抑制分支代谢途径,获得高产重组菌株。顾洋[41]使用强组成型启动子P43替换葡萄糖/氢离子协同转运蛋白载体和葡萄糖激酶编码基因glcP、glcK和糖酵解途径关键基因pfkA和fbaA的原始启动子,强化B. subtilis自身的转运系统,避免磷酸烯醇式丙酮酸对其转录水平的反馈调控。GlcN目前主要以甲壳素水解法生产,水解法消耗大量的酸碱,纯化工艺复杂,存在过敏效应。相对水解法,微生物发酵法生产不受资源限制,对环境污染小,不存在过敏效应,借助合成生物学技术进行GlcN代谢途径的移植和转化系统强化,改造后的枯草芽孢杆菌能顺利积累GlcN,并显著提高了发酵生产GlcN的产量与生产强度。
糖胺聚糖广泛用于临床治疗、保健品和化妆品领域,例如透明质酸(hyaluronic acid,HA)、硫酸软骨素(chondroitinsulfate,CS)等。当前糖胺聚糖的主要生产方式是从组织提取,产量有限,不同来源的提取物分子质量差异很大,且存在稳定性差、产品纯度不足等缺点。基于合成生物学策略,构建工程菌进行生产可避免上述问题,还可实现产品分子质量分布的精准控制。Hubbard等[42]发现兽疫链球菌(S. equisimilis)透明质酸合酶(hyaluronan synthase,HAS)与人HAS跨膜拓扑相似,序列同一性显著,对Se-HAS进行体外催化,将纯化后Se-HAS重构为蛋白脂质体,实现了HA的体外合成。Jeong等[43]构建了含有多种HA途径基因组合盒的表达载体,最终将来自非洲爪蟾的HAS整合至毕赤酵母基因组,工程菌HA产量达0.8~1.7 g/L。CS广泛分布在软骨连结组织中,和GlcN配合使用有一定的止疼、促进软骨再生的功效,硫酸软骨素糖胺聚糖还是大脑中生长因子信号和神经干细胞稳态的关键调节因子。CS衍生物有利于原代神经细胞的神经突触生长,He Wenqin等[44]对4-O-硫酸转移酶和6-O-硫酸转移酶等关键酶进行修饰,成功在大肠杆菌和毕赤酵母中产出CS,为借助工程菌酶法合成CS提供了基础。基于合成生物学技术及理念构建重组工程菌生产糖胺聚糖的方式必将取代传统动物组织提取,这能提高糖胺聚糖的使用安全性,同时降低其生产成本,为保健食品和医药的研发提供大量优质的原材料。
3.4 有机酸
发酵法被应用于多种有机酸生产,有机酸在医药领域需求广泛,因此有机酸生产已经逐渐成为发酵产业的重要部分。莽草酸是合成抗病毒药物奥司他韦的关键中间体,其衍生物具有抗肿瘤、防止血栓形成等多种功效,可以通过化学合成、微生物发酵和从某些植物中提取来获得,由于植物提取成本高,通过发酵在重组微生物中生产莽草酸可能成为既定的优选途径。侯建屾等[45]利用生长依赖启动子和去电子构建了动态分子开关,这种动态分子开关将细胞生长与莽草酸合成分开,发酵72 h后生成莽草酸14.33 g/L。Liu Chang等[46]根据大肠杆菌和谷氨酸棒杆菌遗传密码子使用偏好性,构建适配性增强的3-脱氢奎宁酸脱水酶元件库,为合成生物学应用定制了新DNA序列,并获得可用于设计代谢工程和合成生物学的莽草酸途径人工模块。Niu Fuxing等[47]开发了生物传感器介导、等离子体诱变与基因组改组相结合的菌株改良策略,通过引入外源β-呋喃果糖苷酶基因,对产蔗糖大肠杆菌进行改造,得到的利用蔗糖的大肠杆菌菌株莽草酸产量达(24.64±0.32)g/L。以往从植物分离莽草酸的成本较高,产量有限,限制了其作为合成原料的应用。采用合成生物学手段,借助异源合成途径的移植,使本不能合成莽草酸的大肠杆菌在改造后有望实现莽草酸及其代谢途径产物的工业化生产。
α-酮异戊酸是多个生物合成和化学合成领域中重要的中间体,广泛应用于制药和保健品行业,例如合成临床药物α-酮酸片用于治疗尿毒症。Liu Long等[48]在一株能够产支链氨基酸氨基转移酶、将L-亮氨酸和丙酮酸转化为α-酮异戊酸的蜡样芽孢杆菌中,利用人工神经网络支持遗传算法,对酶催化过程进行建模和优化,经优化发酵8 h后α-酮异戊酸质量浓度达到5.63 g/L,比未优化组提高了3 倍。宋阳[49]进行N端密码子替换,将催化底物L-亮氨酸脱氨合成α-酮异己酸的L-氨基酸脱氨酶活力提高24.89%。李雅婷[50]构建微氧调控开关,协调辅酶循环,调控α-酮异戊酸合成途径和丙酮酸消耗途径,实现了α-酮异戊酸高效发酵合成。α-酮己酸在食品、饲料和生物制药工业中的应用日益增多,α-酮己酸的高效生产越来越受到人们的关注,Song Yang等[51]在大肠杆菌中,从转录和翻译水平对L-氨基酸脱氨酶基因的表达进行微调,以提高α-酮异己酸效价,通过优化具有不同拷贝数的质粒来源、调节起始密码子下游的信使RNA结构以及设计RBS的序列,构建的重组工程菌产α-酮异己酸质量浓度最高达到86.55 g/L。α-酮异戊酸、α-酮己酸目前以化学合成为主,合成过程需要高成本的催化剂,生产成本较高。在传统改造方法所构建的菌株之上,借助人工网络的模拟计算,结合感受器和开关联动,微调关键酶的表达,更为精细地对代谢途径进行优化改造,为α-酮酸的发酵工业化合成提供了新思路。
3.5 天然产物
天然产物结构复杂多样,具有多种活性,但其天然合成途径和代谢调控较复杂,需要对宿主合成途径进行系统设计和改造,以提高天然产物的生物合成效率。随着合成生物学技术日益成熟,异源表达已被广泛应用于天然产物生产。梁蓉等[52]利用毕赤酵母真核表达体系实现拟南芥AtPOFUT1蛋白异源表达,初步检测明确了该重组蛋白具有O-岩藻糖基转移酶活性。Paddon等[53]通过重新改造合成途径过表达植物脱氢酶和细胞色素P450,使得青蒿酸在酿酒酵母中发酵产量达到25 g/L,为青蒿素化学合成的前体大量生产提供了可能。Bian Guangkai等[54]开发了萜类化合物生物合成平台,通过在酿酒酵母中过表达tHMG1,去除跨膜结构域,得到能高产萜类化合物菌株。Zhou Yongjin等[55]建立模块化工程途径并在酿酒酵母中组装合成次丹参酮二烯通路。Dai Zhubo等[56]在酿酒酵母中引入不同植物来源的齐墩果酸合酶、原人参二醇合酶、原人参三醇合成酶等,构建了原人参二醇、原人参三醇和齐墩果酸的合成途径。Yamanaka等[57]构建了基于转化相关重组的基因平台,该平台可以直接克隆、重组和异源表达沉默生物合成途径,从而产生新抗生素,利用该方法成功表达了来自海洋放线菌糖单孢菌的非核糖体肽合成酶生物合成基因簇,并在模型表达宿主链霉菌中产生了二氯化脂肽抗生素他霉素A,为新天然产物发现和药物开发开辟了途径。通过分析天然产物生物合成途径,它们均遵循生物合成逻辑而通过体内相关酶催化反应产生。因此,通过构建文库和平台,对次生代谢产物生物合成基因元件进行改造和拼装,可人工构建天然产物的生物合成途径,优化工程菌自身代谢,有效提高天然产物产量。
近年单纯合成天然产物不再满足人们的需要,研究者致力于改造天然产物以提高其水溶性和活性,重构合成途径,减少其结构类似副产物,获得高纯度高活性的目标天然产物。Liu Xiaochen等[58]在大肠杆菌中重组表达欧洲山芥来源的糖基转移酶,并利用尿苷二磷酸-糖基转移酶对产物进行糖基化修饰,最终获得水溶性和生物活性显著提高的甘草次酸-3-O-单葡萄糖。天然产物二聚化也是一种有助于提高活性的修饰方法,Matsuda[59]在烟曲霉中发现天然产物Neosartorin合成基因簇,并通过一系列基因缺失实验研究了其生物合成,还发现了用于异二聚化的P450单加氧酶接受非天然底物以提供新的Neosartorin二聚体,二聚化产物的抗菌活性较初产物有显著提升。Liu Yiqi等[60]在酵母中通过重新设计天然转录调节回路,构建了信号强度增加20 倍的葡萄糖抑制和乙醇诱导的转录信号扩增装置,并利用该装置生物合成了一种降血脂药物中间体莫纳可林J,首次构建并系统工程化了一株产抗生素二氢莫纳可林L的菌株。Wang Zheng等[61]对需钠弧菌(Vibrio natriegens)进行工程改造,通过利用诱导型启动子表达异源酪氨酸酶基因来合成黑色素,并证明了黑色素的产生比以前报道的异源系统快得多。在重组工程菌的基础上进行不断的改进和调整,是天然产物发酵生产研究的前景方向。
4 结 语
合成生物学相关技术研究为构建和生产预期化合物工程菌株提供了强有力技术支撑,是发酵工业领域研究的重要方向。采用合成生物学技术构建高效微生物工程菌,能够在保持菌体生长正常,使工业微生物在代谢稳定的基础上提高已有的产能和生理性能。将合成生物学工具应用于定向进化,能缩短工程菌定向进化周期,增加突变体筛选效率,将其应用于代谢工程,在将生物系统作为一个整体进行工程改造前提下,通过动态控制各复杂途径表达量,可以迅速提升产品多样性。以开发氨基酸、GlcN等功能性成分为代表的工程菌发酵研究目前取得了显著进展。在理论研究、技术探索和借鉴典型合成回路的基础上,进一步扩宽合成的目标范围、构建智能化工程菌和实现大规模生产是未来合成生物学技术的重要方向。
[1]GARDNER T S, CANTOR C R, COLLINS J J. Construction of a genetic toggle switch in Escherichia coli[J]. Nature, 2000, 403: 339-342. DOI:10.1038/35002131.
[2]LE Cong, LIN Shuailiang, DAVID C, et al. Multiplex genome engineering using CRISPR/Cas systems[J]. Science, 2013, 339: 819-823. DOI:10.1126/science.1231143.
[3]汪莲, 王浩君, 罗云孜. CRISPR技术在微生物合成生物学中的应用[J].生命科学, 2019, 31(5): 493-507. DOI:10.13376/j.cbls/2019060.
[4]BERND Z, JONATHAN S G, OMAR O A, et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system[J]. Cell,2015, 163(3): 759-771. DOI:10.1016/j.cell.2015.09.038.
[5]KRUMBACH K, SONNTAG C K, EGGELING L, et al. CRISPR/Cas12a mediated genome editing to introduce amino acid substitutions into the mechanosensitive channel MscCG of Corynebacterium glutamicum[J]. ACS Synthetic Biology, 2019, 8(12): 2726-2734.DOI:10.1021/acssynbio.9b00361.
[6]DING Dan, CHEN Kaiyuan, CHEN Yuedan, et al. Engineering introns to express RNA guides for Cas9- and Cpf1-mediated multiplex genome editing[J]. Molecular Plant, 2018, 11(4): 542-552.DOI:10.1016/j.molp.2018.02.005.
[7]FERENCZI A, PYOTT D E, XIPNITOU A, et al. Efficient targeted DNA editing and replacement in Chlamydomonas reinhardtii using Cpf1 ribonucleoproteins and single-stranded DNA[J]. Proceedings of the National Academy of Sciences of the United States of America,2017, 114(51): 13567-13572. DOI:10.1073/pnas.1710597114.
[8]DANIEL G G, GWYNEDD A B, CYNTHIA A P, et al. Complete chemical synthesis, assembly, and cloning of a Mycoplasma genitalium genome[J]. Science, 2008, 319: 1215-1220. DOI:10.1126/science.1151721.
[9]HUTCHISON III C A, CHUANG R Y, NOSKOV V N, et al. Design and synthesis of a minimal bacterial genome[J]. Science, 2016, 351:aad6253. DOI:10.1126/science.aad6253.
[10]SHEN Yue, WANG Yun, CHEN Tai, et al. Deep functional analysis of synII, a 770-kilobase synthetic yeast chromosome[J]. Science, 2017,355: eaaf4791. DOI:10.1126/science.aaf4791.
[11]SHAO Yangyang, LU Ning, WU Zhenfang, et al. Creating a functional single-chromosome yeast[J]. Nature, 2018, 560: 331-335.DOI:10.1038/s41586-018-0382-x.
[12]贾斌. 精确控制合成型基因组重排[D]. 天津: 天津大学, 2017: 12.
[13]SALIS H M, MIRSKY E A, VOIGT C A. Automated design of synthetic ribosome binding sites to control protein expression[J].Nature Biotechnology, 2009, 27(10): 946-950. DOI:10.1038/nbt.1568.
[14]SHI Feng, FAN Zhengyu, ZHANG Shuping, et al. Optimization of ribosomal binding site sequences for gene expression and 4-hydroxyisoleucine biosynthesis in recombinant Corynebacterium glutamicum[J]. Enzyme and Microbial Technology, 2020, 140:109622. DOI:10.1016/j.enzmictec.2020.109622.
[15]潘海博, 覃璐琪, 黄燕婷, 等. 人工神经网络结合遗传算法优化保护剂提高罗伊氏乳杆菌抗冻性能[J]. 食品科学, 2021, 42(14): 70-77.DOI:10.7506/spkx1002-6630-20200323-350.
[16]叶超. 新一代工业微生物生物网络模型的构建及应用[D]. 无锡:江南大学, 2019: 22.
[17]汪建峰. 大肠杆菌萜类异源合成系统的设计与构建[D]. 上海: 华东理工大学, 2014: 16.
[18]孔德莉, 罗思, 彭瑞晨, 等. 基于无标记金纳米簇的新型荧光生物传感器在赭曲霉毒素A快速检测中的应用[J]. 食品科学, 2021, 42(4):263-270. DOI:10.7506/spkx1002-6630-20200212-108.
[19]王兴. 基于LuxI/R群体效应的生物触发器系统构建[D]. 天津: 天津大学, 2008: 17.
[20]UCHIYAMA T, MIYAZAKI K. Product-induced gene expression, a product-responsive reporter assay used to screen metagenomic libraries for enzyme-encoding genes[J]. Applied and Environmental Microbiology,2010, 76(21): 7029-7035. DOI:10.1128/AEM.00464-10.
[21]ROGERS J K, CHURCH G M. Genetically encoded sensors enable real-time observation of metabolite production[J]. Proceedings of the National Academy of Sciences, 2016, 113(9): 2388-2393.DOI:10.1073/pnas.1600375113.
[22]KORTMANN M, MACK C, BAUMGART M, et al. Pyruvate carboxylase variants enabling improved lysine production from glucose identified by biosensor-based high-throughput fluorescenceactivated cell sorting screening[J]. ACS Synthetic Biology, 2019, 8(2):274-281. DOI:10.1021/acssynbio.8b00510.
[23]BINDER D, FROHWITTER J, MAHR R. Light-controlled cell factories: employing photocaged isopropyl-β-D-thiogalactopyranoside for light-mediated optimization of lac promoter-based gene expression and (+)-valencenebiosynthesis in Corynebacterium glutamicum[J].Applied and Environmental Microbiology, 2016, 82(20): 6141-6149.DOI:10.1128/AEM.01457-16.
[24]CHENG Lin, ZHANG Junsheng, QI Yanjun, et al. Production of α-ketobutyrate using engineered Escherichia coli via temperature shift[J]. Biotechnology and Bioengineering, 2016, 113(9): 2054-2059.DOI:10.1002/bit.25959.
[25]ZHOU Libang, ZENG Anping. Exploring lysine riboswitch for metabolic flux control and improvement of L-lysine synthesis in Corynebacterium glutamicum[J]. ACS Synthetic Biology, 2015, 4(6):729-734 DOI:10.1021/sb500332c.
[26]修宇. 基于RNA核糖开关调控的类黄酮生物传感器的构建与应用[D].北京: 北京化工大学, 2017: 16.
[27]CANNON K A, PARK R U, BOYKEN S E, et al. Design and structure of two new protein cages illustrate successes and ongoing challenges in protein engineering[J]. Protein Science, 2020, 29(4): 919-929.DOI:10.1002/pro.3802.
[28]HUANG P S, FELDMEIER K, PARMEGGIANI F, et al. De novo design of a four-fold symmetric TIM-barrel protein with atomiclevel accuracy[J]. Nature Chemical Biology, 2016, 12(1): 29-34.DOI:10.1038/nchembio.1966.
[29]DOYLE L, JAZMINE H, JILL B, et al. Rational design of α-helical tandem repeat proteins with closed architectures[J]. Nature, 2015, 528:585-598. DOI:10.1038/nature16191.
[30]SIEGEL J B, LEE SMITH A, POUST S, et al. Computational protein design enables a novel one-carbon assimilation pathway[J].Proceedings of the National Academy of Sciences, 2015, 112(12):3704-3709. DOI:10.1073/pnas.1500545112.
[31]徐显皓. 枯草芽孢杆菌中心代谢级联调控回路的设计、构建与应用[D]. 无锡: 江南大学, 2021: 6.
[32]GOENTORO L, SHOVAL O, KIRSCHNER M W, et al. The incoherent feedforward loop can provide fold-change detection in gene regulation[J]. Molecular Cell, 2009, 36(5): 894-899. DOI:10.1016/j.molcel.2009.11.018.
[33]PLANES F J, BEASLEY J E. A critical examination of stoichiometric and path-finding approaches to metabolic pathways[J]. Discrete Applied Mathematics, 2008, 157(10): 2244-2256. DOI:10.1093/bib/bbn018.
[34]赵禹, 刘士琦, 李建, 等. 解脂耶氏酵母作为微生物细胞工厂的应用研究进展[J].食品科学, 2021, 42(19): 388-400. DOI:10.7506/spkx1002-6630-20200802-027.
[35]龙梦飞. 系统代谢改造大肠杆菌高效合成反式-4-羟基-L-脯氨酸[D].无锡: 江南大学, 2021: 11.
[36]ZHANG Bin, YU Miao, ZHOU Ying, et al. Improvement of L-ornithine production by attenuation of argF in engineered Corynebacterium glutamicum S9114[J]. AMB Express, 2018, 8(1): 26. DOI:10.1186/s13568-018-0557-8.
[37]BARKER J L, FROST J W. Microbial synthesis of p-hydroxybenzoic acid from glucose[J]. Biotechnology and Bioengineering, 2001, 76(4):376-390. DOI:10.1002/bit.10160.
[38]VERHOEF S, BALLERSTEDT H, VOLKERS J M, et al. Comparative transcriptomics and proteomics of p-hydroxybenzoate producing Pseudomonas putida S12: novel responses and implications for strain improvement[J]. Applied Microbiology and Biotechnology, 2010,87(2): 679-690. DOI:10.1007/s00253-010-2626-z.
[39]MEIJNEN J P, VERHOEF S, BRIEDJLAL A A, et al. Improved p-hydroxybenzoate production by engineered Pseudomonas putida S12 by using a mixed-substrate feeding strategy[J]. Applied Microbiology and Biotechnology, 2011, 90(3): 885-893. DOI:10.1007/s00253-011-3089-6.
[40]刘延峰. 代谢工程改造枯草芽孢杆菌高效合成N-乙酰氨基葡萄糖[D].无锡: 江南大学, 2015: 14.
[41]顾洋. 重构枯草芽孢杆菌糖转运途径及中心代谢网络高效合成N-乙酰氨基葡萄糖[D]. 无锡: 江南大学, 2020: 17.
[42]HUBBARD C, MCNAMARA T J, AZUMAYA C, et al. The hyaluronan synthase catalyzes the synthesis and membrane translocation of hyaluronan[J]. Journal of Molecular Biology, 2012,418(1/2): 21-31 DOI:10.1016/j.jmb.2012.01.053.
[43]JEONG E, SHIM W Y, KIM J H. Metabolic engineering of Pichia pastoris for production of hyaluronic acid with high molecular weight[J]. Journal of Biotechnology, 2014, 185(1): 28-36.DOI:10.1016/j.jbiotec.2014.05.018.
[44]HE Wenqin, ZHU Yuanyuan, SHIRKE A, et al. Expression of chondroitin-4-O-sulfotransferase in Escherichia coli and Pichia pastoris[J]. Applied Microbiology and Biotechnology, 2017, 101(18):6919-6928. DOI:10.1007/s00253-017-8411-5.
[45]侯建屾, 高聪, 陈修来, 等. 使用动态分子开关调控大肠杆菌生产莽草酸[J]. 生物工程学报, 2020, 36(10): 2104-2112. DOI:10.13345/j.cjb.200078.
[46]LIU Chang, LIU Yiming, SUN Qinglan, et al. Unraveling the kinetic diversity of microbial 3-dehydroquinate dehydratases of shikimate pathway[J]. AMB Express, 2015, 5(1): 7. DOI:10.1186/s13568-014-0087-y.
[47]NIU Fuxing, HE Xin, HUANG Yuanbin, et al. Biosensor-guided atmospheric and room-temperature plasma mutagenesis and shuffling for high-level production of shikimic acid from sucrose in Escherichia coli[J]. Journal of Agricultural and Food Chemistry, 2020, 68(42):11765-11773. DOI:10.1021/acs.jafc.0c05253.
[48]LIU Long, LI Jianghua, JUN Fang, et al. Process modeling and optimization of whole-cell biotransformation synthesis of α-ketoisocaproate by Bacillus cereus producing branched-chain amino acid aminotransferase with artificial neural network coupling genetic algorithm[J]. Journal of Bioscience, 2009, 108(1): S127-S128.DOI:10.1016/j.jbiosc.2009.08.372.
[49]宋阳. L-氨基酸脱氨酶分子改造及生物合成α-酮异己酸[D]. 无锡:江南大学, 2018: 21.
[50]李雅婷. 代谢工程改造大肠杆菌生产α-酮异戊酸[D]. 无锡: 江南大学, 2020: 16.
[51]SONG Yang, LI Jianghua, SHIN H D, et al. Tuning the transcription and translation of L-amino acid deaminase in Escherichia coli improves α-ketoisocaproate production from L-leucine[J]. PLoS ONE,2017, 12(6): e0179229. DOI:10.1371/journal.pone.0179229.
[52]梁蓉, 谢尚强, 赵小明, 等. 拟南芥O-岩藻糖基转移酶AtPOFUT1的鉴定及生理功能初探[J]. 西北农业学报, 2021, 30(1): 116-123.
[53]PADDON C J, WESTFALL P J, PITERA D J, et al. High-level semisynthetic production of the potent antimalarial artemisinin[J]. Nature,2013, 496: 528-532. DOI:10.1038/nature12051.
[54]BIAN Guangkai, HOU Anwei, YUAN Yujie, et al. Metabolic engineering-based rapid characterization of a sesquiterpene cyclase and the skeletons of fusariumdiene and fusagramineol from Fusarium graminearum[J]. Organic Letters, 2018, 20(6): 1626-1629.DOI:10.1021/acs.orglett.8b00366.
[55]ZHOU Yongjin, GAO Wei, RONG Qixian, et al. Modular pathway engineering of diterpenoid synthases and the mevalonic acid pathway for miltiradiene production[J]. Journal of the American Chemical Society, 2012, 134 (6): 3234-3241. DOI:10.1021/ja2114486.
[56]DAI Zhubo, WANG Beibei, LIU Yi, et al. Producing aglycons of ginsenosides in bakers’ yeast[J]. Scientific Reports, 2014, 4(1): 689-699. DOI:10.1038/srep03698.
[57]YAMANAKA K, REYNOLDS K A, KERSTEN R D, et al. Direct cloning and refactoring of a silent lipopeptide biosynthetic gene cluster yields the antibiotic taromycin A[J]. Proceedings of the National Academy of Sciences, 2014, 111(5): 1957-1962. DOI:10.1073/pnas.1319584111.
[58]LIU Xiaochen, ZHANG Liang, FENG Xudong, et al. Biosynthesis of glycyrrhetinic acid-3-O-monoglucose using glycosyltransferase UGT73C11 from Barbarea vulgaris[J]. Industrial & Engineering Chemistry Research,2017, 56(51): 14949-14958. DOI:10.1021/acs.iecr.7b03391.
[59]MATSUDA Y, GOTFREDSEN C H, LARSEN T O. Genetic characterization of neosartorin biosynthesis provides insight into heterodimeric natural product generation[J]. Organic Letters, 2018,20(22): 7197-7200. DOI:10.1021/acs.orglett.8b03123.
[60]LIU Yiqi, BAI Chenxiao, LIU Qi, et al. Engineered ethanol-driven biosynthetic system for improving production of acetyl-CoA derived drugs in Crabtree-negative yeast[J]. Metabolic Engineering, 2019,54(1): 275-284. DOI:10.1016/j.ymben.2019.05.001.
[61]WANG Zheng, TSCHIRHART T, SCHULTZHAUS Z, et al. Melanin produced by the fast-growing marine bacterium Vibrio natriegens through heterologous biosynthesis: characterization and application[J].Applied and Environmental Microbiology, 2020, 86(5): 19.DOI:10.1128/AEM.02749-19.