胶原蛋白是哺乳动物体内含量最丰富的一类蛋白质,占体内总蛋白含量的30%以上,广泛分布于皮肤、动脉、肌腱、软骨和大多数细胞外基质中[1]。胶原蛋白含有大量重复的“Gly-X-Y”氨基酸序列(Gly为甘氨酸;X、Y为任意氨基酸,其中X主要为脯氨酸,Y主要为羟脯氨酸),可形成三股肽链规律缠绕的螺旋结构,从而赋予胶原蛋白较高的机械强度及稳定性[2]。胶原蛋白的这一特性在支持组织结构和保护机体器官上具有重要作用,但是却因其结构稳定、难以水解,导致蛋白利用率较低。目前,水解胶原蛋白的方法主要有化学法和酶法。化学法主要通过酸或碱作用使胶原蛋白纤维溶胀,肽键断裂。此类方法经济简便,但是水解率较低,易破坏氨基酸结构,所得胶原蛋白肽质量不高。酶法水解条件温和,普通蛋白酶(如胰蛋白酶、木瓜蛋白酶和碱性蛋白酶等)酶解时,通过化学法或高温辅助使胶原蛋白分子变性为明胶则更利于深度水解。而胶原蛋白酶是一种能在生理条件下识别天然胶原蛋白特殊三股螺旋区域,并对其进行水解的蛋白酶。使用胶原蛋白酶水解胶原蛋白更易获得具有生物活性的胶原蛋白肽,是提高胶原蛋白水解水平的重要手段[3]。
胶原蛋白酶主要存在于微生物[4]和动物[5]中,在少数植物[6-7]中也见报道。它们通常以锌离子和钙离子为辅因子,具有多个结构域,在催化结构域中存在保守的锌结合基序HEXXH(图1A)。相较于动物源胶原蛋白酶,微生物源胶原蛋白酶的分子质量普遍更大、结构域更复杂(图1B)、底物适应性更强,且未发现存在酶原形式,无需通过激活酶原产生酶活性。目前已经筛选到多种可分泌胶原蛋白酶的菌株,如溶组织梭状芽孢杆菌(Clostridium histolyticum,现更名为Hathewaya histolytica)[8]、产气荚膜梭菌(Clostridium perfringens)[9]、破伤风梭菌(Clostridium tetani)[10]和蜡样芽孢杆菌(Bacillus cereus)[11]等。其中,以来源于溶组织梭状芽孢杆菌的胶原蛋白酶研究最为广泛[12-15],是目前公认的用于鉴定分析新型胶原蛋白酶的标准酶,也是可商品化获取的微生物源胶原蛋白酶[4]。

图1 微生物源和动物源胶原蛋白酶序列及结构域比较
Fig. 1 Comparison of the sequence and domain of collagenase from microbial and animal sources
1953年,Mandl等[16]首次通过示差沉淀法从溶组织梭状芽孢杆菌的发酵液中分离出含有胶原蛋白酶的组分。随后,Yoshihara[17]和Matsushita[18]等研究发现溶组织梭状芽孢杆菌可分泌两种不同的胶原蛋白酶ColG和ColH。根据两种胶原蛋白酶对天然胶原蛋白底物和特异性合成肽底物FALGPA(N-[3-(2-Furyl)acryloyl]-Leu-Gly-Pro-Ala)水解活性的差异,ColG和ColH又分别称为I型胶原蛋白酶(对天然胶原蛋白的水解活性较高)和II型胶原蛋白酶(对FALGPA水解活性较高)。近年来,学者围绕两种胶原蛋白酶进行了大量的研究报道。本文在国内外最新研究进展的基础上,对溶组织梭状芽孢杆菌胶原蛋白酶的结构特征、水解机理、生产现状和实际应用等进行归纳总结,并对其未来的研究方向进行展望,以期能够为微生物源胶原蛋白酶的进一步开发和利用提供参考依据。
1 微生物源胶原蛋白酶结构特征
微生物源胶原蛋白酶通常具有多个结构域(图2),分别是前肽、激活结构域、肽酶结构域、PKD和CBD。按照其功能又可将激活结构域和肽酶结构域归为催化功能模块,将PKD和CBD归为胶原募集模块。胶原募集模块的差异也是不同菌种来源的胶原蛋白酶在结构差异性上的重要体现。
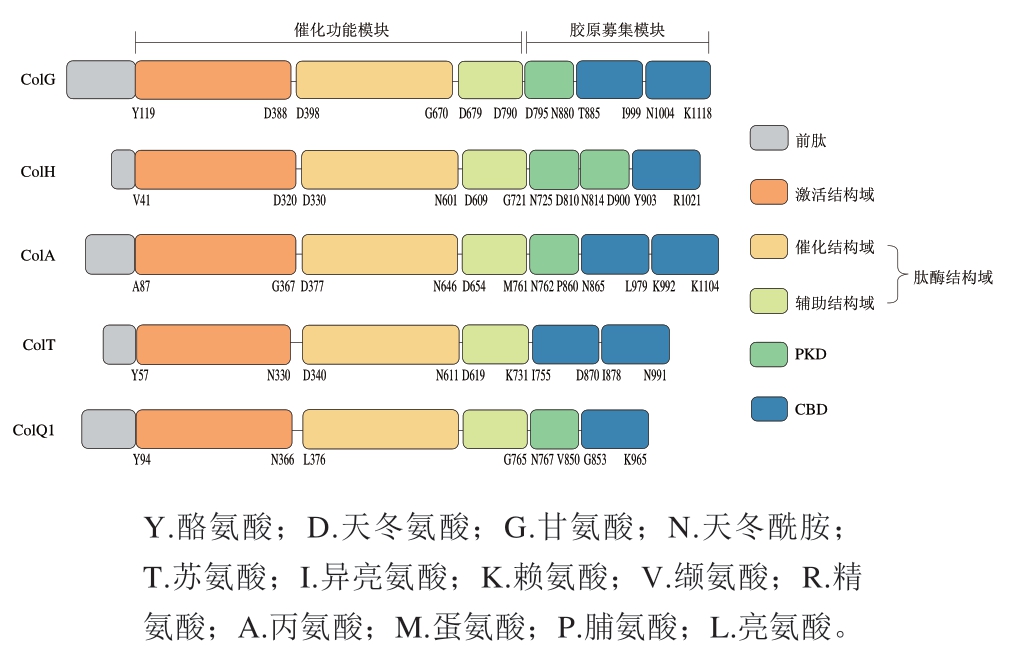
图2 微生物源胶原蛋白酶结构域比对
Fig. 2 Domains of different microbial collagenases
1994年,Yoshihara等[17]从溶组织梭状芽孢杆菌JCM 1403中克隆胶原蛋白酶基因colH,发现该基因的开放阅读框含有3 066 个碱基对,共编码1 021 个氨基酸。1999年,Matsushita等[18]从溶组织梭状芽孢杆菌JCM 1403的培养上清液中纯化出与ColH的N末端氨基酸序列不同的胶原蛋白酶ColG,该蛋白含有1 118 个氨基酸,由colG(3 357 bp)编码。
前肽可进一步分为信号肽和前导肽。ColG的前肽包括110 个氨基酸,其中前45 个氨基酸为信号肽。而ColH的前肽为40 个氨基酸,前30 个氨基酸为信号肽。信号肽是引导新合成蛋白质进入分泌通路的短肽,可将蛋白质定位到含不同膜结构的亚细胞器中。但是,目前胶原蛋白酶在溶组织梭状芽孢杆菌中的具体分泌途径未知,该信号肽介导蛋白质的分泌区域尚不清楚。Eckhard[19]和Ducka[20]等在大肠杆菌中重组表达胶原蛋白酶时发现缺少前肽序列也可以得到具有催化活性的胶原蛋白酶,认为该结构并不是蛋白正确折叠所必需的。
肽酶结构域是发挥胶原蛋白肽水解活性的重要结构,当其单独存在时,其水解短肽的能力与全长胶原蛋白酶相近,但却丧失了水解天然胶原蛋白的活性。当激活结构域与肽酶结构域共同存在时,水解天然胶原蛋白的活性恢复。溶组织梭状芽孢杆菌胶原蛋白酶是拥有多模块结构的锌金属蛋白酶,其N端的肽酶结构域中具有保守的HEXXH锌结合基序,该基序与其下游间隔28~32 个氨基酸的另一谷氨酸共同形成锌离子配位结构。Eckhard等[21]解析了ColG的晶体结构,发现Zn2+与His523、His527、Glu555和一个水分子可形成一个四面体结构,水分子以氢键的形式与Glu524相连。Schönauer等[22]解析了ColH与抑制剂复合时的晶体结构,认为His455、Glu456、His459和Glu487与Zn2+形成配位结构。
胶原募集模块中的CBD可特异性地识别结合胶原蛋白的三股螺旋结构[24-25],是胶原蛋白酶水解不溶性天然胶原蛋白的重要结构,其倾向于结合在胶原蛋白三股螺旋较松散的部分[26]。PKD与胶原蛋白的结合不紧密,但可以进一步强化CBD与纤维状胶原蛋白的结合能力[27-28]。如图3所示,ColG含有两个串联CBD和一个PKD,Ca2+的存在可以使ColG中两个CBD之间的连接部分由α-螺旋转变为β-折叠,增强CBD与胶原蛋白的结合,也使其不易被其他蛋白酶水解,提高了全长胶原蛋白酶的稳定性[25-26,29-30]。ColH含有一个CBD和两个PKD,CBD可以沿着胶原蛋白纤维滑动,找到更容易水解的位点,两个PKD的存在使其对胶原蛋白的亲和力更强。根据这一结构特征,二者可协同水解胶原蛋白,即ColG先锚定在胶原蛋白纤维最脆弱的区域使原纤维溶胀,ColH结合在这些溶胀的区域进行水解[28]。
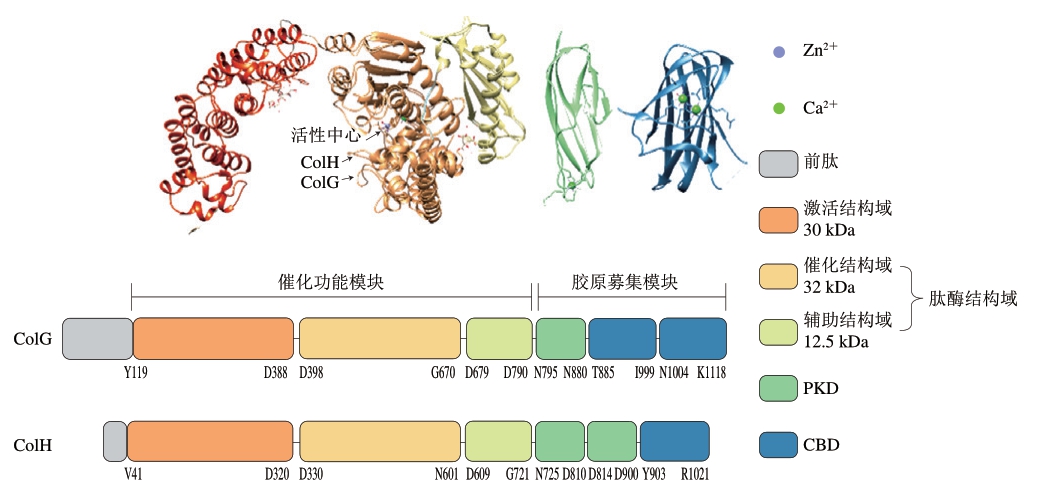
图3 ColG和ColH的结构示意图[21,23]
Fig. 3 Structures of ColG and ColH[21,23]
此外,胶原蛋白酶谱实验表明[18],这两种酶除了116 kDa的全长形式,对C末端部分水解还可得到若干残余的N末端(如源自ColG的67、78 kDa和82 kDa片段;源自ColH的98、105 kDa片段),且依旧具有酶解明胶的能力,因而也被称为明胶酶。这些残余片段对明胶的酶解活性甚至高于全长形式的胶原蛋白酶,但只有全长的胶原蛋白酶才能消化不溶性胶原蛋白[27],这也说明胶原蛋白酶C末端的胶原募集模块在水解天然胶原蛋白中发挥了重要作用。
2 胶原蛋白酶的水解机理
2011年,Eckhard等[21]首次对胶原蛋白酶ColG进行整体晶体结构解析,并通过分别异源表达该酶各个结构域推测其水解胶原蛋白和微原纤维胶原蛋白(由5 条三股螺旋缠绕而成)的机理,大致分为以下3 个步骤,如图4所示。
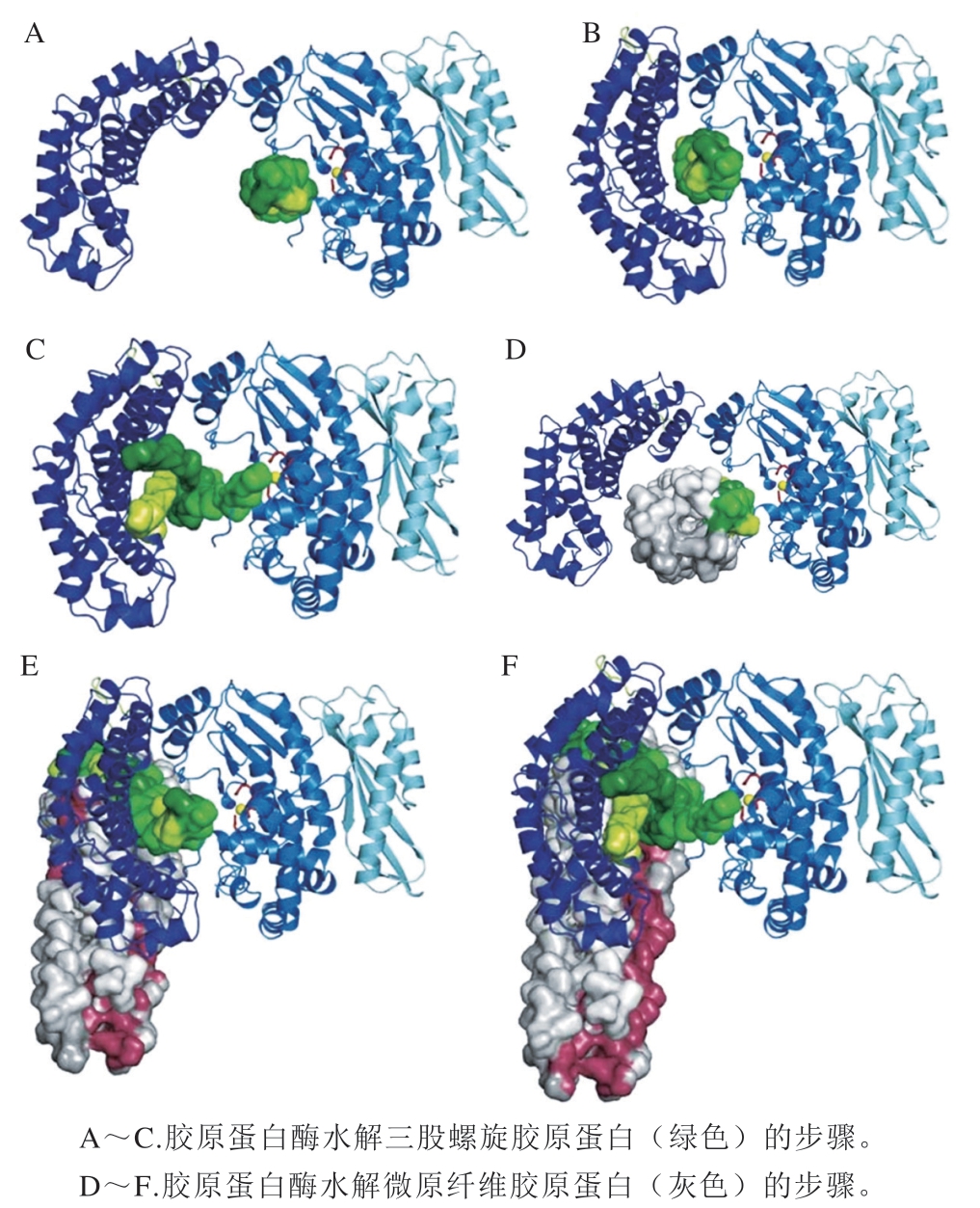
图4 胶原蛋白和微原纤维胶原蛋白的水解模型[21]
Fig. 4 Collagenolytic models of triple-helical and microfibrillar collagen[21]
首先胶原蛋白酶的肽酶结构域与胶原蛋白三股螺旋结合(图4A);此时胶原蛋白酶处于开放状态,激活结构域未与底物相互作用,胶原蛋白尚未发生水解;随后胶原蛋白酶闭合,激活结构域与三股螺旋发生相互作用(图4B)。水解过程中,胶原蛋白酶渐渐打开成半开放构象,使3 个α-链解旋并依次降解(图4C)。在三股螺旋被彻底水解后,胶原蛋白酶便恢复至初始开放状态,进行下一轮水解。对于由5 条三股螺旋缠绕形成更为复杂的微原纤维胶原蛋白,水解步骤也大致相同,但每次只水解一个三股螺旋胶原蛋白,其余部分被排到酶外等待下一次水解(图4D~F)。
胶原蛋白酶ColG和ColH的酶切位点都是胶原蛋白“Gly-X-Y”重复序列中Y与Gly之间的肽键,但二者底物偏好性不同,首选的切割位点也有差异(表1),共同存在时表现出对胶原蛋白的协同降解作用[18,31-33]。ColG的首选底物为三股螺旋胶原蛋白,首选切割位点为N末端和C末端。而ColH水解底物的亲和力从大到小依次为多肽、变性胶原蛋白和三股螺旋胶原蛋白,首选切割位点为序列内部。
表1 I型和II型溶组织梭状芽孢杆菌胶原蛋白酶的特性
Table 1 Characteristics of type I and type II collagenases from Clostridium histolyticum

名称 类型 结构 氨基酸个数分子质量/kDa 亲和力 胶原结合结构域 首选切割位点 首选底物胶原ColG I型胶原蛋白酶 单链多肽 1 118 116 较高 串联结构域(CBD1、CBD2)N末端和C末端 三股螺旋胶原蛋白ColH II型胶原蛋白酶 单链多肽 1 021 116 中等 单域(CBD) 序列内部 多肽>变性胶原蛋白>三股螺旋胶原蛋白
Eckhard等[34]利用蛋白酶裂解位点的蛋白质组学鉴定(proteomic identification of protease cleavage sites,PICS)分析了胶原蛋白酶ColG和ColH的切割位点特异性,发现胶原蛋白酶ColG和ColH在P2和P2’位置显示出对Pro的明显偏好,在P3和P1’中显示出对Gly的偏好。这与典型胶原蛋白中大量存在的Gly-Pro-X和Gly-X-Hyp序列一致,解释了溶组织梭状芽孢杆菌胶原蛋白酶水解天然胶原蛋白的高度特异性。
3 胶原蛋白酶活力检测方法
胶原蛋白酶的酶活力检测方法主要有平板法、显色法和荧光法等。
平板法可用于初步筛选具有胶原蛋白水解活性的菌株。具体方法是将具有胶原蛋白酶活性的菌株在明胶平板上进行培养后,用酸性汞试剂处理平板,未被降解的明胶变性沉淀,可观察到菌落周围出现水解圈,通过水解圈与菌落直径比值的大小可以初步判断该菌株分解胶原蛋白能力的强弱。该方法操作简便,可用于初步鉴定胶原蛋白酶水解活性,但是水解圈大小根据观察时间不同而有所区别,且灵敏度较低,多用于定性检测。
显色法中,茚三酮显色法是一种常规的胶原蛋白酶酶活检测方法,通过酶水解底物释放至体系中游离氨基酸的含量来表征胶原蛋白酶的水解活力。该方法通常以可溶性胶原蛋白、不溶性胶原蛋白和变性胶原蛋白明胶等作为底物,以甘氨酸或亮氨酸的生成当量来定义酶活力单位U。Zhang Yanfang等[35]对常规的茚三酮显色法进行优化,将660 μg明胶溶解在343 μL反应缓冲液(50 mmol/L Tris-HCl,pH 7.5,含有5 mmol/L CaCl2和1 μmol/L ZnCl2)中,加入7 μL质量浓度1 mg/mL胶原蛋白酶,37 ℃孵育,再加入等量终止液(含有12%(m/V)聚乙二醇6000和25 mmol/L乙二胺四乙酸二钠)终止反应。随后取100 μL反应液加入500 μL茚三酮显色试剂,80 ℃水浴10 min,冷却后再加入500 μL H2O并混合均匀,在570 nm波长处测定吸光度。优化后反应形成的紫色络合物更加稳定,且不需要强酸沉淀、离心和其他额外处理来去除未反应底物和胶原蛋白酶。该方法经济方便,但是特异性低,若未经纯化直接取培养液上清进行测定,容易受到菌株分泌的其他蛋白酶或者培养液中残留氨基酸的影响,从而对实验结果造成干扰。
除了以天然或变性胶原蛋白为底物,一些人工合成的特异性底物也常用于胶原蛋白酶活力的检测,如Azocoll、FALGPA(N-(3-[2-furyl]acryloyl)-L-leucylglycyl-L-prolyl-L-alanine)和Pz-PLGPA(PZ-L-prolyl-L-leucyl-glycyl-L-prolyl-D-arginine)等。Azocoll是一种与偶氮染料连接的胶原蛋白,被胶原蛋白酶水解后染料释放,可通过测定520 nm波长处吸光度来计算酶活力。FALGPA和Pz-PLGPA是人工合成的胶原蛋白酶寡肽底物,不易被其他蛋白酶水解。其中,FALGPA是美国Sigma-Aldrich公司用来定义胶原蛋白酶产品酶活力的常用底物,通过测定酶与底物反应后345 nm波长处吸光度的减少量计算水解量,1 个FALGPA unit指在25 ℃、pH 7.5的条件下,每分钟水解1 μmol的FALGPA[36]。而Pz-PLGPA法则以反应后在320 nm波长处测吸光度的增量定义酶活力单位,1 个PZ unit指在25 ℃、pH 7.1的条件下,每分钟催化水解1 μmol的Pz-PLGPA[37]。此类方法通常较为灵敏,且测得的酶活力较准确,但使用合成短肽作为底物测得的酶活力不能直接体现该胶原蛋白酶对天然胶原蛋白的水解活性。
除了这些常见的底物,Saikumari等[38]合成了一种新的荧光底物,能专一地被溶组织梭状芽孢杆菌胶原蛋白酶切割而不受其他蛋白酶影响,该底物水解后受336 nm激发光激发后,在490 nm波长处的发射荧光显著增强,反应快速灵敏。Go等[39]利用邻苯二甲醛(1,2-phthalic dicarboxaldehyde,OPA)与游离氨基酸结合产生荧光的特点,在胶原蛋白酶与底物反应后的反应液中加入质量浓度1 mg/mL OPA(0.1 mol/L PBS,pH 7.4),被360 nm激发光激发后,检测460 nm波长处的荧光强度。此类方法快捷灵敏,能在较短的时间内获得大量数据,但荧光试剂通常较为昂贵,检测成本较高。
除了上述方法,胶原蛋白酶谱法也用于胶原蛋白酶活力的检测。胶原蛋白酶谱法是一种基于十二烷基硫酸钠聚丙烯酰胺凝胶电泳(sodium dodecyl sulfatepolyacrylamide gel electrophoresis,SDS-PAGE)的检测方法。样品在含有底物蛋白的聚丙烯酰胺凝胶中电泳分离或在电泳后将凝胶浸泡在底物溶液中,再通过孵育、染色、脱色的方法显示条带。一般使用考马斯亮蓝染色后脱色,在蓝色背景下可观察到白色条带(底物降解后不被着色),条带的灰度与胶原蛋白酶活力成正比。Inanc等[40]用含有质量浓度0.3 mg/mL提取自鼠尾肌腱的I型胶原蛋白的凝胶进行电泳,经去除SDS、孵育、染色、脱色后可见清晰条带。该方法十分灵敏,检测限可低至1 ng胶原蛋白酶,并且可以基于胶原蛋白酶标准曲线来量化未知样品中的胶原蛋白酶活力性水平。此外,胶原蛋白与胶原蛋白酶的酶解反应引起的物理变化(例如浊度和黏度的变化)也被用于分析胶原蛋白酶酶活力[41-42]。
4 胶原蛋白酶生产现状
4.1 发酵生产
目前商业化的胶原蛋白酶主要来自溶组织梭状芽孢杆菌,通过培养溶组织梭状芽孢杆菌再从其培养上清液中分离纯化所需的胶原蛋白酶。该方法的优势在于溶组织梭状芽孢杆菌将胶原蛋白酶分泌到培养基中,可以在简单的液体培养基中大量培养,获得胶原蛋白酶的效率较高。但该方法所得的胶原蛋白酶粗制剂中还含有棕色色素、梭菌蛋白酶、氨基肽酶和几种中性蛋白酶。梭菌蛋白酶的存在导致粗酶制剂会对成骨细胞和胰岛细胞产生细胞毒性[43-44],也会导致纯化后期和贮存运输过程中胶原蛋白酶的降解。上清液中其他酶类以及多种C末端截短的胶原蛋白酶的存在增加了胶原蛋白酶分离纯化时工艺的难度,且容易造成不同批次的胶原蛋白酶粗制剂活性差异较大[45]。目前,研究人员主要从优化溶组织梭状芽孢杆菌的培养基和改进纯化方式两大方面着手提高胶原蛋白酶的纯度。
Wegman等[46]采用以植物氮源为主的培养基培养溶组织梭状芽孢杆菌,结果发现降低葡萄糖质量浓度(质量浓度小于5 g/L)和增加盐质量浓度(质量浓度大于5 g/L)能显著降低梭菌蛋白酶的含量,纯化过程可以在室温下进行,并且无需添加蛋白酶抑制剂,所得的胶原蛋白酶制剂酶活与标准胶原蛋白酶相当,纯度大于95%,在4 ℃存放17 d后仍能保持活性且未见降解。该方法分离的得到的I型胶原蛋白酶以可溶性鼠尾胶原蛋白(soluble rat tail collagen,SRC)[47]为底物测得酶活力为1 967~3 327 U/mg,II型胶原蛋白酶以合成肽Carbobenzooxy-glycyl-L-prolylglycyl-glycyl-L-prolyl-L-alanine(zGPGGPA)[48]为底物,测得酶活力为81 934~119 522 U/mg。
Sabatino等[49]采用非动物源性培养基培养C. histolyticum strains 004制备冷冻保存的菌种库,以蛋白胨为主要氮源制备培养液,并采用分批补料发酵方式进行发酵,发酵结束后经0.2 μm过滤器、离子交换色谱、疏水层析和切向流过滤得到纯化的胶原蛋白酶,总产量为280~350 mg/L,纯度大于97%。该方法可以扩大到20 L规模发酵,且批次质量稳定,分离的得到的I型胶原蛋白酶以SRC为底物,测得酶活力为1 700~3 500 U/mg,II型胶原蛋白酶以合成肽zGPGGPA为底物,测得酶活力为43 000~69 000 U/mg。
4.2 异源表达
为获取较高纯度的胶原蛋白酶,以便进一步对胶原蛋白酶的生化性质、酶结构、催化机理、构效关系展开研究,同时拓展其在生物技术和医学等领域应用,研究人员尝试通过基因工程的手段重组表达生产胶原蛋白酶。目前相关研究已经在不同的表达体系中开展,如大肠杆菌[17-18,20,50-52]、枯草芽孢杆菌[27,53]和产气荚膜梭菌[54]等。
大肠杆菌具有生长迅速、遗传背景清晰、易于基因编辑等优点,是研究最广泛的模式生物。1994年,Yoshihara等[17]在大肠杆菌中首次克隆了源自溶组织梭状芽孢杆菌的胶原蛋白酶基因colH。随后,相关的重组表达研究陆续开展[17-18,20,50-52]。2009年,Ducka等[20]在大肠杆菌中表达和纯化了溶组织梭状芽孢杆菌胶原蛋白酶ColG和ColH,即以pMBP-Parallel2为载体,E. coli BL21为宿主进行诱导发酵,采用固定化金属亲和色谱初步纯化、直链淀粉亲和色谱再次纯化,去除N端标签后再采用离子交换色谱和尺寸排阻色谱进行最终纯化,实现了质量浓度至少10 mg/L的产量,纯度达到95%。Bertuzzi等[52]通过密码子优化、可溶性优化、培养条件优化等策略,将密码子优化后的胶原蛋白酶ColG和ColH基因序列分别克隆到pMAL-c2X载体中,转化菌株接种到TB培养基(补充有质量浓度100 μg/mL氨苄青霉素和质量分数0.2%葡萄糖)中,30 ℃、260 r/min培养,当菌液OD600 nm≈0.6时加入终浓度0.3 mmol/L的异丙基-β-D-硫代半乳糖苷诱导重组蛋白表达,30 ℃、260 r/min培养3 h可获得约质量浓度160 mg/L的ColG或ColH。与市售的溶组织梭状芽孢杆菌源胶原蛋白酶相比,该重组胶原蛋白酶以Z-Gly-Pro-Gly-Gly-Pro-Ala为底物时酶活力约是Liberase(瑞士Roche公司)的1.7 倍,且在处理胰岛细胞时,仅用NB 8 Broad Range(德国NordMark公司)1/5的酶量便可达到相同的提取效果。
除了以大肠杆菌为表达载体,胶原蛋白酶也可在其他原核体系进行异源表达。1996年,Jung等[53]采用Bacillus subtilis DB104异源表达ColH,发现相比LB培养基,表达载体在含有0.5 mol/L琥珀酸钠的改良LB培养基中稳定性较高。重组胶原蛋白ColH产量约为0.66 mg/L,以Pz-PLGPA为底物测得酶活力为1 210 U/mg。1998年,Matsushita等[27]也在Bacillus subtilis DB104中表达了C末端融合GST标签的ColH,以Pz-PLGPA为底物测得酶活力为771.5 U/mg,以I型胶原蛋白为底物测得酶活力为1 413.8 U/mg。此外,2008年Tamai等[54]构建了质粒pFN,在产气荚膜梭菌中表达了C末端带有6×His标签的胶原蛋白酶ColH,纯化得到的蛋白质量浓度约10 mg/L,以Pz-PLGPA为底物测得酶活力为1 028 U/mg,以I型胶原蛋白为底物测得酶活力为1 216 U/mg。他们发现C末端带有6×His标签的胶原蛋白酶对I型胶原蛋白的水解活性较不带标签的ColH低,推测是由于C末端的6×His标签影响CBD结构域与胶原蛋白的亲和力,从而影响胶原蛋白酶对不溶性胶原蛋白的水解活性。此外,在胶原蛋白酶N末端的6×His标签对其酶活也有一定影响,这可能是由于6×His标签干扰了催化活性中心Zn2+的正确定位[19,21]从而影响了胶原蛋白酶的活力。目前溶组织梭状芽孢杆菌源胶原蛋白酶的异源表达主要集中在原核体系(大肠杆菌、枯草芽孢杆菌、产气荚膜梭菌),真核体系中的研究报道较少。大肠杆菌表达体系可以实现异源蛋白的高水平表达,胶原蛋白酶的产量已到达160 mg/L[52]。该系统具有丰富的功能元件和完善的分子遗传操作平台,有利于研究的深入开展,但是该系统容易形成包涵体,有待通过研究提高可溶性胶原蛋白酶的产量。枯草芽孢杆菌表达体系具有高效的蛋白胞外分泌能力,且无明显的密码子偏好性,但是该系统存在质粒不稳定的问题[53],可用的生物元件相对较少。产气荚膜梭菌与溶组织梭状芽孢杆菌同源性高,有利于溶组织梭状芽孢杆菌源胶原蛋白酶的正确折叠表达,但是该系统在代谢工程中应用较少。真核系统可实现蛋白质的翻译后修饰,如糖基化和磷酸化等,也能实现胞外分泌及可溶性表达,利于下游纯化,但是目前溶组织梭状芽孢杆菌源胶原蛋白酶在真核生物中的异源表达鲜见报道。在毕赤酵母中进行基础表达的研究曾有报道,但未进行表达优化,也未进一步对表达所得的胶原蛋白酶进行纯化研究。
综上所述,溶组织梭状芽孢杆菌源胶原蛋白酶异源表达的产量和酶活力仍有待提升,需综合考量酶自身特性和宿主的蛋白分泌特征,利用相应的代谢工程手段进行全局调控从而实现高效表达。
5 胶原蛋白酶的实际应用
5.1 生物医药
胶原蛋白是细胞外基质和结缔组织中的主要结构蛋白,其异常的合成和积累常常会导致纤维化相关疾病,因此,在生物医药领域常采用注射胶原蛋白酶的手段治疗相关疾病,如腰椎间盘突出[55-56]、掌筋膜挛缩[57]和佩罗尼病[58]等。掌筋膜挛缩是掌腱筋膜中胶原蛋白的沉积导致掌指关节发生挛缩,使手指的正常功能受损的疾病,多发于中老年男性。2010年2月美国食品药品监督管理局(Food and Drug Administration,FDA)批准了溶组织梭状芽孢杆菌胶原蛋白酶用于掌筋膜挛缩的非手术疗法。胶原蛋白酶注射疗法与掌筋膜切断术等手术疗法相比具有副作用更少、作用温和、患者满意度高等优势[59]。Syed等[60]的体外研究表明溶组织梭状芽孢杆菌胶原蛋白酶能有效地消化病灶处的细胞外基质,而不会对非胶原组织成分产生明显的细胞毒性或结构损伤,但该方法也存在复发率较高的问题。Villegas等[61]制备了一种胶原蛋白酶纳米胶囊,并在局部真皮纤维化的小鼠模型中进行了实验。该胶囊可在生理条件下逐渐释放封装的胶原蛋白酶,药效最长可达10 d。与注射游离胶原蛋白酶相比,注射胶原蛋白酶纳米胶囊的小鼠真皮纤维化面积显著减少,且并未观察到其他不良影响。
胶原蛋白酶还可应用于伤口清创,清除伤口处坏死或污染的组织有助于伤口的恢复,对于一些慢性、不愈合的伤口具有较好的效果,在临床上应用广泛[62]。以溶组织梭状芽孢杆菌胶原蛋白酶为主要成分的胶原蛋白酶软膏Santyl是唯一一种获得FDA批准用于伤口和烧伤清创的酶制剂。研究表明,溶组织梭状芽孢杆菌胶原蛋白酶可以在7 个特定的位点对胶原蛋白进行切割,清除坏死组织的同时也产生一些副产物(生物活性肽),可以刺激成纤维细胞、角质形成细胞和内皮细胞的迁移和增殖,改善伤口的炎症反应,促进血管生成和伤口愈合[63-66]。Waycaster等[67]对使用Santyl软膏治疗慢性皮肤溃疡的临床效果和治疗成本进行了分析,认为胶原蛋白酶软膏无论是单独使用或是辅以其他的治疗手段均可以有效清创,且不受使用群体和诊疗环境的限制。与使用水凝胶敷料或手术清创的方法相比,使用胶原蛋白酶治疗的患者复诊清创的次数更少,伤口恢复更快,综合而言花费的治疗成本较低[68-69]。此外,胶原蛋白酶还用于治疗青光眼[70]、子宫肌瘤[71-72]、慢性完全闭塞病变[73]、基因治疗[74-75]等方面,相关的临床研究有待进一步发展。
细胞培养是分子生物学、生物技术以及生物制药等研究和产业领域的重要技术,有效的细胞分离是其关键环节,通常使用酶来使细胞组织解聚。由于胶原蛋白是细胞外基质的重要成分且胶原蛋白酶不损伤细胞膜,用胶原蛋白酶处理含有大量胶原纤维的组织后,可获得各种细胞功能正常、形状完整且存活率高的细胞,多年来已被广泛应用于细胞分散、组织分离和实验室细胞培养,成功用于分离骨骼肌干细胞、内皮细胞、脂肪基质细胞、神经细胞以及胰岛等[76-80]。Ishii等[79]利用胶原蛋白酶分离得到的骨骼肌干细胞保留了细胞的表面抗原,并且具有良好的再生能力。胰蛋白酶虽然也常用于细胞解离,但天然胶原蛋白的三股螺旋区对胰蛋白酶有较强的抵抗作用[81]。因此,对于一些高胶原蛋白含量的纤维组织和对胰蛋白酶敏感的组织更适合使用胶原蛋白酶进行细胞分离,二者与其他酶复合使用可以得到更好的分离效果[82]。
5.2 食品加工
在食品工业中,使用酶来辅助食品加工是常见的方法,它可以提高食品的营养和功能特性。在胶原蛋白酶的作用下,胶原蛋白被水解成胶原蛋白肽。研究表明摄入胶原蛋白肽可能有助于机体合成胶原蛋白,具有多种健康益处,如改善皮肤健康、减缓关节疼痛和预防骨质疏松等[83-87]。Yang Xinghao等[88]利用胶原蛋白酶水解鱼骨、鱼皮、鱼鳞等海产品副产物,发现水解物具有较高的抗氧化活性。Song Yihang等[89]利用重组胶原蛋白酶水解牛骨胶原蛋白,通过响应面法优化水解条件,使用质量浓度110 μg/mL的胶原蛋白酶在35 ℃、pH 8条件下制得可溶性胶原蛋白肽,并从中鉴定出5 种新型抗氧化肽。Yi Jierong等[90]使用胶原蛋白酶水解草鱼鱼皮得到的水解物具有血管紧张素转换酶抑制活性和抗氧化活性,可用于开发降血压功能性食品,还能延长食品的保质期。2020年6月,美国胶原蛋白品牌Vital Proteins多数股权被雀巢健康科学收购,这是其首次收购胶原蛋白生产企业,标志着行业巨头对胶原蛋白领域的重视。近年来,我国胶原蛋白市场处于蓬勃发展阶段,2019年中国胶原蛋白的市场规模增长至9.83亿 美元,约占全球市场的6.40%,预计到2027年,中国胶原蛋白的市场规模将达到15.76亿 美元,胶原蛋白肽作为功能食品配料和膳食补充剂具有可观的应用前景。
在肉类嫩化方面,肉的嫩度与结缔组织中胶原蛋白的含量密切相关,胶原蛋白水解酶可以通过消化胶原蛋白使肉变嫩,并在肉制品中产生良好的风味,因此在肉类工业中得到了广泛的应用。其他动植物蛋白酶对底物的特异性不强,用其处理肉制品可能会带来一些不良的风味,影响肉制品的感官品质,而用胶原蛋白酶处理能针对性地水解胶原蛋白,降低结缔组织的韧性使肉类嫩化[91]。
6 结 语
溶组织梭状芽孢杆菌胶原蛋白酶可高效水解胶原蛋白,在生物医药、食品加工等行业得到了广泛应用,是最具代表性的微生物源胶原蛋白酶。目前,对溶组织梭状芽孢杆菌胶原蛋白酶的组成和结构已有了较为清晰的认识,同时该酶也在大肠杆菌、枯草芽孢杆菌和产气荚膜梭菌中实现了异源表达,但是仍然有以下问题有待解决。
在分泌机制方面,胶原蛋白酶在溶组织梭状芽孢杆菌中的具体分泌机制未知,前导肽的具体功能作用及ColG和ColH分子截短形式的产生机理有待研究。在酶产量方面,针对纯化标签对胶原蛋白酶活力的影响以及蛋白产量低等问题,可通过选择不同的表达体系(如尝试真核表达体系:酿酒酵母或毕赤酵母等),并对异源表达体系进行代谢途径改造来进一步提高生产效率。在实际应用方面,为提高胶原蛋白酶的稳定性和生物利用度,还可通过包埋法、吸附法和共价结合法等对胶原蛋白酶进行固定化。在酶学性质拓展方面,可利用计算机辅助设计进行酶工程化改造提高酶活力、稳定性和底物特异性等。此外,由于胶原蛋白酶为模块化酶,可对不同胶原蛋白酶的优势模块进行交换,获取更具有优势的胶原蛋白酶。
[1]FRATZL P. Collagen: structure and mechanics[M]. Boston: Springer,2008: 1-13.
[2]RICARD-BLUM S. The collagen family[J]. Cold Spring Harbor Perspectives in Biology, 2011, 3: a004978. DOI:10.1101/cshperspect.a004978.
[3]宋易航, 王楚浩, 方柏山. 胶原酶研究进展与应用[J]. 化工学报,2019, 70(9): 3213-3227.
[4]DUARTE A S, CORREIA A, ESTEVES A C. Bacterial collagenases:a review[J]. Critical Reviews in Microbiology, 2016, 42(1): 106-126.DOI:10.3109/1040841X.2014.904270.
[5]DABOOR S, BUDGE S, GHALY A, et al. Isolation and activation of collagenase from fish processing waste[J]. Advances in Bioscience and Biotechnology, 2012, 3: 191-203. DOI:10.4236/abb.2012.33028.
[6]RASKOVIC B, BOZOVIC O, PRODANOVIC R, et al. Identification,purification and characterization of a novel collagenolytic serine protease from fig (Ficus carica var. Brown Turkey) latex[J]. Journal of Bioscience and Bioengineering, 2014, 118(6): 622-627. DOI:10.1016/j.jbiosc.2014.05.020.
[7]KIM M, HAMILTON S E, GUDDAT L W, et al. Plant collagenase:unique collagenolytic activity of cysteine proteases from ginger[J].Biochimica et Biophysica Acta-General Subjects, 2007, 1770(12):1627-1635. DOI:10.1016/j.bbagen.2007.08.003.
[8]MANDL I, ZIPPER H, FERGUSON L T. Clostridium histolyticum collagenase: its purification and properties[J]. Archives of Biochemistry and Biophysics, 1958, 74(2): 465-475. DOI:10.1016/0003-9861(58)90017-1.
[9]MATSUSHITA O, YOSHIHARA K, KATAYAMA S, et al. Purification and characterization of Clostridium perfringens 120-kilodalton collagenase and nucleotide sequence of the corresponding gene[J].Journal of Bacteriology, 1994, 176(1): 149-156. DOI:10.1128/jb.176.1.149-156.1994.
[10]BRÜGGEMANN H, BÄUMER S, FRICKE W F, et al. The genome sequence of Clostridium tetani, the causative agent of tetanus disease[J]. Proceedings of the National Academy of Sciences of the United States of America, 2003, 100(3): 1316-1321. DOI:10.1073/pnas.0335853100.
[11]HOPPE I J, BRANDSTETTER H, SCHONAUER E. Biochemical characterisation of a collagenase from Bacillus cereus strain Q1[J].Scientific Reports, 2021, 11(1): 4187. DOI:10.1038/s41598-021-83744-6.
[12]VAN WART H E, STEINBRINK D R. Complementary substrate specificities of class I and class II collagenases from Clostridium histolyticum[J]. Biochemistry, 1985, 24(23): 6520-6526. DOI:10.1021/bi00344a032.
[13]BOND M D, VAN WART H E. Relationship between the individual collagenases of Clostridium histolyticum: evidence for evolution by gene duplication[J]. Biochemistry, 1984, 23(13): 3092-3099.DOI:10.1021/bi00308a037.
[14]BOND M D, VAN WART H E. Purification and separation of individual collagenases of Clostridium histolyticum using red dye ligand chromatography[J]. Biochemistry, 1984, 23(13): 3077-3085.DOI:10.1021/bi00308a035.
[15]BOND M D, VAN WART H E. Characterization of the individual collagenases from Clostridium histolyticum[J]. Biochemistry, 1984,23(13): 3085-3091. DOI:10.1021/bi00308a036.
[16]MANDL I, MACLENNAN J D, HOWES E L. Isolation and characterization of proteinase and collagenase from Cl. histolyticum[J].The Journal of Clinical Investigation, 1953, 32(12): 1323-1329.DOI:10.1172/JCI102861.
[17]YOSHIHARA K, MATSUSHITA O, MINAMI J, et al. Cloning and nucleotide sequence analysis of the colH gene from Clostridium histolyticum encoding a collagenase and a gelatinase[J]. Journal of Bacteriology, 1994,176(21): 6489-6496. DOI:10.1128/jb.176.21.6489-6496.1994.
[18]MATSUSHITA O, JUNG C M, KATAYAMA S, et al. Gene duplication and multiplicity of collagenases in Clostridium histolyticum[J]. Journal of Bacteriology, 1999, 181(3): 923-933. DOI:10.1128/JB.181.3.923-933.1999.
[19]ECKHARD U, SCHÖNAUER E, DUCKA P, et al. Biochemical characterization of the catalytic domains of three different clostridial collagenases[J]. Biological Chemistry, 2009, 390(1): 11-18.DOI:10.1515/BC.2009.004.
[20]DUCKA P, ECKHARD U, SCHONAUER E, et al. A universal strategy for high-yield production of soluble and functional clostridial collagenases in E. coli[J]. Applied Microbiology and Biotechnology,2009, 83(6): 1055-1065. DOI:10.1007/s00253-009-1953-4.
[21]ECKHARD U, SCHONAUER E, NUSS D, et al. Structure of collagenase G reveals a chew-and-digest mechanism of bacterial collagenolysis[J]. Nature Structural & Molecular Biology, 2011, 18:1109-1114. DOI:10.1038/nsmb.2127.
[22]SCHÖNAUER E, KANY A M, HAUPENTHAL J, et al. Discovery of a potent inhibitor class with high selectivity toward clostridial collagenases[J]. Journal of the American Chemical Society, 2017,139(36): 12696-12703. DOI:10.1021/jacs.7b06935.
[23]ECKHARD U, SCHONAUER E, BRANDSTETTER H. Structural basis for activity regulation and substrate preference of clostridial collagenases G, H, and T[J]. Journal of Biological Chemistry, 2013,288(28): 20184-20194. DOI:10.1074/jbc.M112.448548.
[24]MATSUSHITA O, KOIDE T, KOBAYASHI R, et al. Substrate recognition by the collagen-binding domain of Clostridium histolyticum class I collagenase[J]. Journal of Biological Chemistry,2001, 276(12): 8761-8770. DOI:10.1074/jbc.M003450200.
[25]PHILOMINATHAN S T L, KOIDE T, HAMADA K, et al.Unidirectional binding of clostridial collagenase to triple helical substrates[J]. Journal of Biological Chemistry, 2009, 284(16): 10868-10876. DOI:10.1074/jbc.M807684200.
[26]BAUER R, WILSON J J, PHILOMINATHAN S T, et al. Structural comparison of ColH and ColG collagen-binding domains from Clostridium histolyticum[J]. Journal of Bacteriology, 2013, 195(2):318-327. DOI:10.1128/JB.00010-12.
[27]MATSUSHITA O, JUNG C M, MINAMI J, et al. A study of the collagen-binding domain of a 116-kDa Clostridium histolyticum collagenase[J]. Journal of Biological Chemistry, 1998, 273(6): 3643-3648. DOI:10.1074/jbc.273.6.3643.
[28]BAUER R, JANOWSKA K, TAYLOR K, et al. Structures of three polycystic kidney disease-like domains from Clostridium histolyticum collagenases ColG and ColH[J]. Acta Crystallographica Section D,2015, 71: 565-577. DOI:10.1107/S1399004714027722.
[29]PHILOMINATHAN S T L, MATSUSHITA O, GENSURE R, et al.Ca2+-induced linker transformation leads to a compact and rigid collagen-binding domain of Clostridium histolyticum collagenase[J].The FEBS Journal, 2009, 276(13): 3589-3601. DOI:10.1111/j.1742-4658.2009.07078.x.
[30]OHBAYASHI N, YAMAGATA N, GOTO M, et al. Enhancement of the structural stability of full-length clostridial collagenase by calcium ions[J]. Applied and Environmental Microbiology, 2012, 78(16): 5839-5844. DOI:10.1128/AEM.00808-12.
[31]FRENCH M F, MOOKHTIAR K A, VAN WART H E. Limited proteolysis of type I collagen at hyperreactive sites by class I and II Clostridium histolyticum collagenases: complementary digestion patterns[J]. Biochemistry, 1987, 26(3): 681-687. DOI:10.1021/bi00377a004.
[32]FRENCH M F, BHOWN A, VAN WART H E. Identification of Clostridium histolyticum collagenase hyperreactive sites in type I, II, and III collagens: lack of correlation with local triple helical stability[J]. Journal of Protein Chemistry, 1992, 11: 83-97.DOI:10.1007/bf01025095.
[33]BREITE A G, MCCARTHY R C, DWULET F E. Characterization and functional assessment of Clostridium histolyticum class I (C1)collagenases and the synergistic degradation of native collagen in enzyme mixtures containing class II (C2) collagenase[J].Transplantation Proceedings, 2011, 43(9): 3171-3175. DOI:10.1016/j.transproceed.2011.09.059.
[34]ECKHARD U, HUESGEN P F, BRANDSTETTER H, et al. Proteomic protease specificity profiling of clostridial collagenases reveals their intrinsic nature as dedicated degraders of collagen[J]. Journal of Proteomics, 2014, 100: 102-114. DOI:10.1016/j.jprot.2013.10.004.
[35]ZHANG Yanfang, FU Yun, ZHOU Sufeng, et al. A straightforward ninhydrin-based method for collagenase activity and inhibitor screening of collagenase using spectrophotometry[J]. Analytical Biochemistry, 2013, 437(1): 46-48. DOI:10.1016/j.ab.2013.02.030.
[36]VAN WART H E, STEINBRINK D R. A continuous spectrophotometric assay for Clostridium histolyticum collagenase[J].Analytical Biochemistry, 1981, 113(2): 356-365. DOI:10.1016/0003-2697(81)90089-0.
[37]WÜNSCH E, HEIDRICH H G. Zur quantitativen bestimmung der kollagenase[J]. Hoppe-Seyler’s Zeitschrift Für Physiologische Chemie,1963, 333: 149-151. DOI:10.1515/bchm2.1963.333.1.149.
[38]SAIKUMARI Y K, BALARAM P. An internally quenched fluorescent substrate for collagenase[J]. Biopolymers, 2008, 90(2): 131-137.DOI:10.1002/bip.20952.
[39]GO K, HORIKAWA Y, GARCIA R, et al. Fluorescent method for detection of cleaved collagens using O-phthaldialdehyde (OPA)[J].Journal of Biochemical and Biophysical Methods, 2008, 70(6): 878-882. DOI:10.1016/j.jbbm.2007.05.004.
[40]INANC S, KELES D, OKTAY G. An improved collagen zymography approach for evaluating the collagenases MMP-1, MMP-8, and MMP-13[J].BioTechniques, 2017, 63(4): 174-180. DOI:10.2144/000114597.
[41]BLEEG H S. Collagenolytic enzymes assayed by spectrophotometry with suspensions of reconstituted collagen fibrils[J]. Connective Tissue Research, 1991, 26(4): 247-257. DOI:10.3109/03008209109152442.
[42]GALLOP P M, SEIFTER S, MEILMAN E. Studies on collagen: I.The partial purification, assay, and mode of activation of bacterial collagenase[J]. Journal of Biological Chemistry, 1957, 227(2): 891-906. DOI:10.1016/S0021-9258(18)70769-2.
[43]SALAMONE M, SEIDITA G, CUTTITTA A, et al. A new method to value efficiency of enzyme blends for pancreatic tissue digestion[J].Transplantation Proceedings, 2010, 42(6): 2043-2048. DOI:10.1016/j.transproceed.2010.05.107.
[44]HEFLEY T, CUSHING J, BRAND J S. Enzymatic isolation of cells from bone: cytotoxic enzymes of bacterial collagenase[J]. American Journal of Physiology-Cell Physiology, 1981, 240(5): C234-C238.DOI:10.1152/ajpcell.1981.240.5.C234.
[45]KIN T, JOHNSON P R V, SHAPIRO A M J, et al. Factors influencing the collagenase digestion phase of human islet isolation[J]. Transplantation,2007, 83(1): 7-12. DOI:10.1097/01.tp.0000243169.09644.e6.
[46]WEGMAN T L, YU B. Compositions and methods for producing clostridial collagenases: US010119131B2[P]. 2018-11-06[2021-06-01].
[47]MALLYA S K, MOOKHTIAR K A, VAN WART H E. Accurate,quantitative assays for the hydrolysis of soluble type I, II, and III 3H-acetylated collagens by bacterial and tissue collagenases[J].Analytical Biochemistry, 1986, 158(2): 334-345. DOI:10.1016/0003-2697(86)90558-0.
[48]GRASSMANN W, NORDWIG A. Quantitative colorimetric test for collagenase[J]. Hoppe-Seyler’s Zeitschrift fur Physiologische Chemie,1960, 322: 267-272. DOI:10.1515/bchm2.1960.322.1.267.
[49]SABATINO G L, DEL TITO B J, BASSETT P J, et al.Compositions and methods for treating collagen-mediated diseases:US20110243920[P]. 2010-10-12[2021-06-01].
[50]HESSE F, BURTSCHER H, POPP F, et al. Recombinant enzymes for islet isolation: purification of a collagenase from Clostridium histolyticum and cloning/expression of the gene[J]. Transplantation Proceedings, 1995, 27(6): 3287-3289.
[51]CRANENBURGH R M, SABATINO G L, DEL TITO B J. Production of recombinant collagenases ColG and ColH in Escherichia coli:US20080233614A1[P]. 2008-09-25[2021-06-01].
[52]BERTUZZI F, CUTTITTA A, GHERSI G, et al. Clostridium histolyticum recombinant collagenases and method for the manufacture thereof: US008715985B2[P]. 2014-05-06[2021-06-01].
[53]JUNG C M, MATSUSHITA O, KATAYAMA S, et al. Expression of the colH gene encoding Clostridium histolyticum collagenase in Bacillus subtilis and its application to enzyme purification[J].Microbiology and Immunology, 1996, 40(12): 923-929. DOI:10.1111/j.1348-0421.1996.tb01161.x.
[54]TAMAI E, MIYATA S, TANAKA H, et al. High-level expression of his-tagged clostridial collagenase in Clostridium perfringens[J].Applied Microbiology and Biotechnology, 2008, 80(4): 627-635.DOI:10.1007/s00253-008-1592-1.
[55]ZHANG D Y, ZHANG Y, WANG Z J, et al. Target radiofrequency combined with collagenase chemonucleolysis in the treatment of lumbar intervertebral disc herniation[J]. International Journal of Clinical and Experimental Medicine, 2015, 8(1): 526-532.
[56]张辉. 注射用胶原酶联合其他疗法治疗椎间盘突出症的临床研究进展[J]. 上海医药, 2016, 37(17): 10-13; 15.
[57]WARWICK D, ARANDES-RENU J M, PAJARDI G, et al.Collagenase Clostridium histolyticum: emerging practice patterns and treatment advances[J]. Journal of Plastic Surgery and Hand Surgery,2016, 50(5): 251-261. DOI:10.3109/2000656X.2016.1159568.
[58]MILLS S A, GELBARD M K. Sixty years in the making: collagenase Clostridium histolyticum, from benchtop to FDA approval and beyond[J]. World Journal of Urology, 2020, 38(2): 269-277.DOI:10.1007/s00345-019-02818-3.
[59]PEIMER C A, WILBRAND S, GERBER R A, et al. Safety and tolerability of collagenase Clostridium histolyticum and fasciectomy for Dupuytren’s contracture[J]. Journal of Hand Surgery, 2015, 40(2):141-149. DOI:10.1177/1753193414528843.
[60]SYED F, THOMAS A N, SINGH S, et al. In vitro study of novel collagenase (XIAFLEX®) on Dupuytren’s disease fibroblasts displays unique drug related properties[J]. PLoS ONE, 2012, 7(2): e31430.DOI:10.1371/journal.pone.0031430.
[61]VILLEGAS M R, BAEZA A, USATEGUI A, et al. Collagenase nanocapsules: an approach to fibrosis treatment[J]. Acta Biomaterialia,2018, 74: 430-438. DOI:10.1016/j.actbio.2018.05.007.
[62]RAMUNDO J, GRAY M. Collagenase for enzymatic debridement: a systematic review[J]. Journal of Wound Ostomy &Continence Nursing, 2009, 36(Suppl 6): S4-S11. DOI:10.1097/WON.0b013e3181bfdf83.
[63]HU Y, WEBB E, SINGH J, et al. Rapid determination of substrate specificity of Clostridium histolyticum beta-collagenase using an immobilized peptide library[J]. Journal of Biological Chemistry, 2002,277(10): 8366-8371. DOI:10.1074/jbc.M111042200.
[64]SHEETS A R, DEMIDOVA-RICE T N, SHI L, et al. Identification and characterization of novel matrix-derived bioactive peptides: a role for collagenase from Santyl® Ointment in post-debridement wound healing?[J]. PLoS ONE, 2016, 11(7): e0159598. DOI:10.1371/journal.pone.0159598.
[65]DAS A, DATTA S, ROCHE E, et al. Novel mechanisms of Collagenase Santyl Ointment (CSO) in wound macrophage polarization and resolution of wound inflammation[J]. Scientific Reports, 2018, 8(1):1696. DOI:10.1038/s41598-018-19879-w.
[66]DEMIDOVA-RICE T N, GEEVARGHESE A, HERMAN I M.Bioactive peptides derived from vascular endothelial cell extracellular matrices promote microvascular morphogenesis and wound healing in vitro[J]. Wound Repair and Regeneration, 2011, 19(1): 59-70.DOI:10.1111/j.1524-475X.2010.00642.x.
[67]WAYCASTER C, CARTER M J, GILLIGAN A M, et al. Comparative cost and clinical effectiveness of clostridial collagenase ointment for chronic dermal ulcers[J]. Journal of Comparative Effectiveness Research, 2018, 7(2): 149-165. DOI:10.2217/cer-2017-0066.
[68]WAYCASTER C, MILNE C T. Clinical and economic benefit of enzymatic debridement of pressure ulcers compared to autolytic debridement with a hydrogel dressing[J]. Journal of Medical Economics, 2013, 16(7): 976-986. DOI:10.3111/13696998.2013.807268.
[69]KARAGOL B S, OKUMUS N, DURSUN A, et al. Early and successful enzymatıc debridement via collagenase application to pinna in a preterm neonate[J]. Pediatric Dermatology, 2011, 28(5): 600-601.DOI:10.1111/j.1525-1470.2011.01546.x.
[70]HONKANEN R. Use of collagenase to treat glaucoma:US9636385B2[P]. 2017-05-02[2021-06-01].
[71]BRUNENGRABER L N, JAYES F L, LEPPERT P C. Injectable Clostridium Histolyticum collagenase as a potential treatment for uterine fibroids[J]. Reproductive Sciences, 2014, 21(12): 1452-1459.DOI:10.1177/1933719114553449.
[72]JAYES F L, LIU B, MOUTOS F T, et al. Loss of stiffness in collagenrich uterine fibroids after digestion with purified collagenase Clostridium histolyticum[J]. American Journal of Obstetrics and Gynecology, 2016,215(5): 596.e1-596.e8. DOI:10.1016/j.ajog.2016.05.006.
[73]STRAUSS B H, OSHEROV A B, RADHAKRISHNAN S, et al.Collagenase total Occlusion-1 (CTO-1) trial[J]. Circulation, 2012,125(3): 522-528. DOI:10.1161/CIRCULATIONAHA.111.063198.
[74]EIKENES L, TUFTO I, SCHNELL E A, et al. Effect of collagenase and hyaluronidase on free and anomalous diffusion in multicellular spheroids and xenografts[J]. Anticancer Research, 2010, 30(2): 359-368.
[75]DOLOR A, SZOKA F C. Digesting a path forward: the utility of collagenase tumor treatment for improved drug delivery[J].Molecular Pharmaceutics, 2018, 15(6): 2069-2083. DOI:10.1021/acs.molpharmaceut.8b00319.
[76]BHAGWAT P K, DANDGE P B. Collagen and collagenolytic proteases: a review[J]. Biocatalysis and Agricultural Biotechnology,2018, 15: 43-55. DOI:10.1016/j.bcab.2018.05.005.
[77]FUJIO A, MURAYAMA K, YAMAGATA Y, et al. Collagenase H is crucial for isolation of rat pancreatic islets[J]. Cell Transplantation,2014, 23(10): 1187-1198. DOI:10.3727/096368913x668654.
[78]BRANDHORST H, KURFÜRST M, JOHNSON P R, et al.Comparison of neutral proteases and collagenase class I as essential enzymes for human islet isolation[J]. Transplantation Direct, 2016,2(1): e47. DOI:10.1097/TXD.0000000000000552.
[79]ISHII K, SUZUKI N, MABUCHI Y, et al. Technical advantage of recombinant collagenase for isolation of muscle stem cells[J].Regenerative Therapy, 2017, 7: 1-7. DOI:10.1016/j.reth.2017.06.001.
[80]BERKOVÁ Z, SAUDEK F, LEONTOVYČ I, et al. Testing of a new collagenase blend for pancreatic islet isolation produced by Clostridium histolyticum[J]. Advances in Bioscience and Biotechnology, 2018, 9(1): 26-35. DOI:10.4236/abb.2018.91003.
[81]YU Z, VISSE R, INOUYE M, et al. Defining requirements for collagenase cleavage in collagen type III using a bacterial collagen system[J]. Journal of Biological Chemistry, 2012, 287(27): 22988-22997. DOI:10.1074/jbc.M112.348979.
[82]MIERSCH C, STANGE K, RÖNTGEN M. Effects of trypsinization and of a combined trypsin, collagenase, and DNase digestion on liberation and in vitro function of satellite cells isolated from juvenile porcine muscles[J]. In Vitro Cellular & Developmental Biology-Animal, 2018, 54(6): 406-412. DOI:10.1007/s11626-018-0263-5.
[83]KUMAR S, SUGIHARA F, SUZUKI K, et al. A double-blind,placebo-controlled, randomised, clinical study on the effectiveness of collagen peptide on osteoarthritis[J]. Journal of the Science of Food and Agriculture, 2015, 95(4): 702-707. DOI:10.1002/jsfa.6752.
[84]KIM D U, CHUNG H C, CHOI J, et al. Oral intake of low-molecularweight collagen peptide improves hydration, elasticity, and wrinkling in human skin: a randomized, double-blind, placebo-controlled study[J]. Nutrients, 2018, 10(7): 826. DOI:10.3390/nu10070826.
[85]ZHU L, XIE Y, WEN B, et al. Porcine bone collagen peptides promote osteoblast proliferation and differentiation by activating the PI3K/Akt signaling pathway[J]. Journal of Functional Foods, 2020, 64: 103697.DOI:10.1016/j.jff.2019.103697.
[86]LEE H J, JANG H L, AHN D K, et al. Orally administered collagen peptide protects against UVB-induced skin aging through the absorption of dipeptide forms, Gly-Pro and Pro-Hyp[J]. Bioscience,Biotechnology, and Biochemistry, 2019, 83(6): 1146-1156. DOI:10.1080/09168451.2019.1580559.
[87]MEI F, LIU J, WU J, et al. Collagen peptides isolated from salmo salar and tilapia nilotica skin accelerate wound healing by altering cutaneous microbiome colonization via upregulated NOD2 and BD14[J]. Journal of Agricultural and Food Chemistry, 2020, 68(6):1621-1633. DOI:10.1021/acs.jafc.9b08002.
[88]YANG Xinghao, XIAO Xiao, LIU Dan, et al. Optimization of collagenase production by Pseudoalteromonas sp. SJN2 and application of collagenases in the preparation of antioxidative hydrolysates[J].Marine Drugs, 2017, 15(12): 377. DOI:10.3390/md15120377.
[89]SONG Yihang, FU Yousi, HUANG Shiyang, et al. Identification and antioxidant activity of bovine bone collagen-derived novel peptides prepared by recombinant collagenase from Bacillus cereus[J]. Food Chemistry, 2021, 349: 129143. DOI:10.1016/j.foodchem.2021.129143.
[90]YI Jierong, DE GOBBA C, SKIBSTED L H, et al. Angiotensin-I converting enzyme inhibitory and antioxidant activity of bioactive peptides produced by enzymatic hydrolysis of skin from grass carp(Ctenopharyngodon idella)[J]. International Journal of Food Properties,2017, 20(5): 1129-1144. DOI:10.1080/10942912.2016.1203932.
[91]CRONLUND A L, WOYCHIK J H. Solubilization of collagen in restructured beef with collagenases and α-amylase[J]. Journal of Food Science, 1987, 52(4): 857-860. DOI:10.1111/j.1365-2621.1987.tb14227.x.